池蝶蚌致雄性化基因fem-1c及其蛋白分子的结构特征分析
2014-03-29熊文芳史建伍蒋谋炎陈永玲盛军庆王军花洪一江
熊文芳 史建伍 蒋谋炎 陈永玲 彭 扣 盛军庆 王军花 洪一江
(南昌大学生命科学与食品工程学院, 南昌 330031)
池蝶蚌致雄性化基因fem-1c及其蛋白分子的结构特征分析
熊文芳 史建伍 蒋谋炎 陈永玲 彭 扣 盛军庆 王军花 洪一江
(南昌大学生命科学与食品工程学院, 南昌 330031)
研究首次获得淡水珍珠贝池蝶蚌(Hyriopsis schlegelii)的致雄性化基因feminization-1C亚型, 即fem-1c基因, 并进行全长克隆及序列分析。从转录组中Race-PCR扩增fem-1c基因全长cDNA序列, 用生物信息学方法对 fem-1c基因进行基因组结构分析、多序列同源性比较、跨膜区段、亲疏水性分析、功能结构域预测等。研究结果如下: 该cDNA序列全长2328 bp, 包含一个1869 bp的最大开放阅读框, 编码622个氨基酸, 经同源比对和进化树分析, fem-1基因c亚型编码的氨基酸与太平洋牡蛎的同源性最高, 为79%; 疏水性分析显示该蛋白为亲水性蛋白; 二级结构预测发现该蛋白有大量的 α螺旋和随机卷曲, 形成 9个锚蛋白重复序列(Ankyrin repeat, ANK), 同时, 三级结构预测中可以清楚的看到9个ANK模体, 跟SMART预测的结构位点结果相近。从无脊椎动物到脊椎动物 fem-1c基因的保守性表明它们有共同的起源, 暗示这个基因家族在功能上具备保守性, 推测池蝶蚌fem-1c基因可能具备与线虫fem-1基因在性别决定方面上相似的功能。
池蝶蚌; fem-1c基因; 克隆; 序列分析; 结构预测
fem基因包括fem-1、fem-2和fem-3, 其中fem-1家族包括3个家庭成员: fem-1a、fem-1b、fem-1c[1]。fem-1是秀丽隐杆线虫控制性别决定信号通路上的基因[2], 其编码的蛋白 FEM-1是一种细胞内蛋白[3],具有锚蛋白重复序列(ANK)结构域, 介导蛋白质与蛋白质之间的相互作用[4]。FEM-2是种丝氨酸/苏氨酸特异性磷酸酶 PP2C[5,6], 它可通过去磷酸化作用直接与FEM-3的锚定蛋白重复序列蛋白相互作用[7,8]。秀丽隐杆线虫有两种性别, 一种是只有1条X染色体的雄性个体(XO), 另一种是具有2条X染色体、并能够自体受精的雌雄同体个体(XX)[9]。 X染色体与常染色体组的比率提供了初级遗传信号, 进而影响her-1基因(Hermaphrodization)的活性。从her-1、tra-2[8]、fem-1、fem-2、fem-3基因[6,10]到tra-1基因,需经过一系列的负调控来控制, 最终由tra-1基因引导个体性别分化[11]。Her-1蛋白是一种分泌蛋白, 在XX 个体中, 该蛋白不表达[12], 导致跨膜蛋白TRA-2抑制FEM蛋白的功能, 从而使TRA-1蛋白参与调控。其中TRA-2蛋白具有跨膜蛋白接收器的结构特点, 能够跨膜与胞内蛋白 FEM 蛋白反应。TRA-1是一种具有锌指结构的 DNA 结合蛋白[13],也是一种转录因子, 它能够抑制有助于雄性性别方向发展基因的表达, 从而使个体体细胞组织性别往雌性发展。在XO个体中, 分泌蛋白HER-1抑制了TRA-2A的活性[14,15], 同时解除了对FEM蛋白的抑制, 这样FEM蛋白就可以抑制TRA-1A蛋白的活性,使其不能调控下游基因的转录, 最终决定了个体性别方向是往雄性发展。不管是在XX个体还是XO个体中, fem-1基因对精子的生成都是必不可少的[16,17]。
淡水珍珠蚌池蝶蚌(Hyriopsis schlegelii)为日本引进品种, 分泌珍珠质能力强[18]。本项目组已对其育珠性能[19]、性腺发育[20]和选育[21,22]等方面进行了研究, 发现其性腺发育过程中存在雌雄同体阶段,但成熟后的个体单性, 表明其性别分化可能存在独特的调控机制。目前, 在低等贝类的性腺发育过程中有关性别决定及分化机制的报道鲜见。为探讨淡水贝类的性别发育, 本研究对池蝶蚌的 fem-1c基因进行克隆, 并运用生物信息学软件对其全长cDNA序列和蛋白质结构特征进行分析, 为进一步揭示池蝶蚌性别决定及分化机制奠定基础。
1 材料与方法
1.1 材料
池蝶蚌取自江西省抚州市洪门水库开发公司国家级池蝶蚌良种场。4龄, 体质健壮, 生长线明显,外套膜厚实, 内脏柔软饱满, 外腮完整无伤。取回后置于水族箱暂养一周, 水温(18—25) , ℃ 每天换已曝气的自来水一次。
1.2 方法
总 RNA的提取与 cDNA的合成 采用Trizol法提取池蝶蚌总 RNA, 使用 1.5% 变性琼脂糖胶和紫外分光光度计检测RNA质量和浓度, 参照SMART RACE Kit (美国, Clonetech)说明进行反转录及目的基因两端序列的克隆。
PCR 引物设计 根据本实验室高通量测序测得的转录组数据筛选出fem-1c基因的部分片段设计引物 orfF、orfR; 3′-RACE引物 GSP1、GSP2; 5′-RACE引物GSP3、GSP4 (表1), 由上海生物工程技术服务有限公司合成。

表1 实验中使用到的引物序列Tab. 1 Sequence of primers used in the experiment
cDNA序列的克隆 利用引物orfF、orfR扩增中间片段, 反应体系为: 10×Extaq PCR Buffer 5 μL, 2.5 mmol/L dNTP Mix 2.5 μL, halorfF 2 μL, halorfR 2 μL, ExTaq酶 0.4 μL, cDNA 1.5 μL, ddH2O 36.6 μL。扩增条件: 95℃预变性5min; 95 ℃ 30s, 60 ℃ 30s, 72℃ 2min, 35个循环; 72 ℃ 延伸10min。
利用通用引物UPM (Long︰Short︰ddH2O为1︰4︰19)和特异性引物 GSP1, 以 3′-RACE-Ready cDNA为模板进行第一轮 PCR, 将扩增的产物稀释10倍作为第二轮巢式 PCR的模版, 以特异性引物GSP2和通用引物UPM进行第二轮PCR。反应体系为10×Extaq PCR Buffer 5 μL, 2.5 mmol/L dNTP Mix 2.5 μL, GSP1 2 μL, UPM 5 μL, Extaq酶 0.4 μL, cDNA 1.5 μL, ddH2O 33.6 μL。第一轮PCR反应程序: 95℃预变性5min; 95 ℃ 30s, 72 ℃1min30s, 5个循环; 95 ℃ 30s, 70 ℃ 30s, 72 ℃ 1min, 5个循环; 95 ℃30s, 60 ℃ 30s, 72 ℃ 1min, 25个循环; 72 ℃ 延伸10min。第二轮 PCR反应程序: 95℃预变性 5min; 95 ℃30s, 72 ℃1min30s, 5个循环; 95 ℃30s, 70℃ 30s, 72℃ 1min, 5个循环; 95 ℃ 30s, 68 ℃ 30s, 72 ℃ 1min, 35个循环; 72 ℃ 延伸10min。5′-RACE扩增反应同理。
1%琼脂糖凝胶电泳检测 PCR产物, 使用OMEGA切胶回收试剂盒回收特异性目的条带, 将回收产物连接到pMD19-T Vector上, 再转入DH5α感受态大肠杆菌中, 用氨苄青霉素筛选阳性菌落,挑选阳性菌落送至上海生工生物工程技术服务有限公司测序。
fem-1基因 c亚型的全长序列拼接及序列分析利用 MEGA4.0软件去掉测序结果的载体序列, 再用Chromas软件观察其测序荧光值, 并经人工校对,拼接获得fem-1c基因的全长cDNA。
BLAST program (http://blast.ncbi.nlm.nih.gov/Blast)与 Expasy (http://www.expasy.org/)在线软件分析序列的同源性、开放阅读框及保守结构域。氨基酸序列经Clustalw2 program (http://www.ebi.ac.uk/Tools/msa/ clustalw2/)排序比对分析保守性; MEGA4.0软件邻位相连法(NJ)[23]建立系统进化树; ProtScale软件分析编码蛋白疏水性进行。
2 结果
2.1 池蝶蚌 fem-1c基因全长的获得及其 cDNA核苷酸序列分析
PCR扩增获得了一条 1900 bp大小的片段, 5′-RACE获得了一条介于100和250 bp的特异性条带, 3′-RACE获得了一条介于250和500 bp的特异性条带(图1)。将这3段特异性条带回收纯化、克隆、测序, 分别得到1900、154和376 bp大小的序列。用MEGA4.0软件将这这3条序列进行拼接, 得到总长度为2328 bp的全长序列。使用ORF finder查找到一个1869 bp的最大开放阅读框(ORF), 编码622个氨基酸, 该阅读框起始密码子ATG的-3位碱基为嘌呤碱基 G, +4位为 G, 符合 Kozak规则[24]。5′端UTR 长137 bp, 3′端UTR长322 bp, 含有典型的加尾信号序列AATAA以及PloyA。
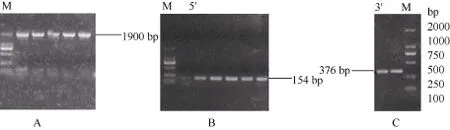
图1 fem-1c基因中间片段产物(A)、5′-RACE产物(B)及3′-RACE产物(C)电泳结果Fig. 1 Electrophoresis result of the middle (A), 5′-RACE (B)and 3′-RACE (C) fragments M: DL2000 plus
2.2 池蝶蚌 Fem1c蛋白理化性质、结构域分析及二、三级结构预测
池蝶蚌 Fem1c蛋白的理化性质 ORF finder分析表明 fem-1c基因的开放阅读框位于(138—2006) bp区域, 编码一条含有622个氨基酸的蛋白质, 分子量为69.95 kD, 理论等电点(PI)为7.05,原子数 9836, 分子式为 C3101H4937N857O900S41, 无信号肽, 不稳定系数34.62。其编码的蛋白亲疏水性分析结果表明其大部分区域为亲水区, 亲水性总平均值为–0.108, 为稳定亲水性蛋白。用http://psort.hgc. jp/form2.html 在线软件预测蛋白的亚细胞定位, 得知细胞质中含52.2%, 线粒体中含21.7%, 细胞核中占17.4%, 液泡中含8.7%。因此, Fem1c蛋白很可能也为胞内蛋白。
池蝶蚌Fem1c蛋白结构域分析及二、三级结构预测 利用SMART软件分析表明, fem-1c基因编码的蛋白含有9个保守结构域(图2)且都属于ANK蛋白家族, ANK重复序列约33个氨基酸长, 一般是包括两个α螺旋和一个环, 环以90°角与两个α螺旋连接, 形成了一个螺旋-环-螺旋结构。在 PROSITE在线软件中分析同样显示Fem1c蛋白含有9个ANK模体(图3), 与SMART软件分析结果相似。
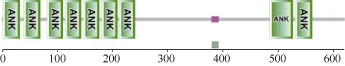
图2 池蝶蚌Fem1c蛋白结构域SMART预测分析Fig. 2 Domain architectures of Fem1c in the Hyriopsis schlegelii by SMART
通过在线软件 https://www.predictprotein.org/ 预测二级结构, 结果表明Fem1c蛋白有大量的 α-螺旋, 达52.09%; 其次为无规则卷曲。利用瑞士生物信息所(Swiss Institute of Bioinformatics, SIB)的 ExPASy服务器上提供的SWISS-MODEL程序http://swissmodel.expasy.org/对池蝶蚌 Fem1c蛋白的三级结构进行初步预测, 结果(图 4)可以清楚地看到9个ANK模体, 与二级结构预测及SMART结构预测结果都比较符合。
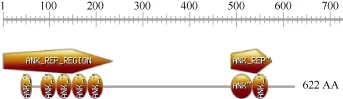
图3 池蝶蚌Fem1c蛋白结构域PROSITE预测分析Fig. 3 Domain architectures of Fem1c in the Hyriopsis schlegelii by PROSITE
2.3 池蝶蚌Fem1c蛋白同源性及系统进化树分析
利用 Clustal软件对池蝶蚌及其他多个物种的Fem1c氨基酸序列进行多序列同源性比对, 结果发现池蝶蚌Fem1c与牡蛎、人和老鼠的Fem1c序列一致率分别为79%、58%和58%。在比对的各个物种的C端发现存在一段固有的序列: P xx L xx F xxx H (x为随机氨基酸)[1]。
用Mega4.0软件邻位连接法将Fem1c氨基酸序列与其他14个物种的Fem1c氨基酸序列构建系统进化树(图5), 比较分析表明池蝶蚌Fem1c与太平洋牡蛎亲缘关系最近, 其次是与海蜗牛接近, 而与人、老鼠等高等动物亲缘关系相对较远, 说明池蝶蚌在进化地位上保守性很强。
3 讨论
fem-1基因对线虫性别决定非常重要, 其产物在信号通路上起作用, 控制着线虫性别分化的方向[7,25,26],它有两个功能: 一是抑制 tra-1基因的活性, 从而使性别往雄性发展; 二是在生殖细胞中它对于精子的生成必不可少。对线虫fem-1基因的研究表明性别决定基因在结构和功能可能具备一定的进化保守性[27]。
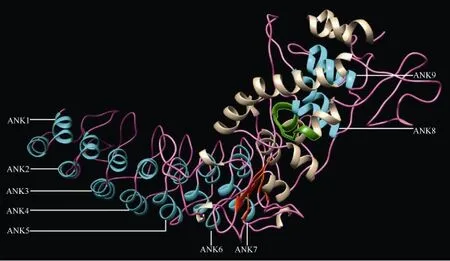
图4 池蝶蚌Fem1c蛋白三级结构预测Fig. 4 The tertiary structure prediction of Fem1c in the Hyriopsis schlegelii
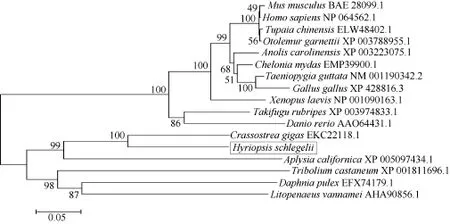
图5 池蝶蚌Fem1c蛋白与其他物种Fem1c蛋白的进化树分析Fig. 5 Phylogenetic tree of the Fem1c of Hyriopsis schlegelii
本研究首次从淡水贝类中成功克隆到fem-1c基因cDNA的全长序列, 基因全长为2328 bp, 开放阅读框长1869 bp, 编码622个氨基酸, 其蛋白二级结构以 α螺旋为主, 其次为随机卷曲; 池蝶蚌 Fem1c蛋白的一个重要特征是含有9个ANK模体。从已有报道显示, ANK结构域的生物功能主要是介导蛋白质与蛋白质之间的相互作用。INK4家族是ANK结构研究得最清楚的一类蛋白质。1998年 Russ等发现该家族的四个成员p16INK4a、p15INK4b、p18INK4c和p19INK4d含有4或5个ANK模体, ANK结构域是重要的功能区, 它们可以特异性地抑制CDK4和CDK6的激酶活性, 对哺乳动物细胞周期G1后期的调控产生影响[28]; Jacobs和Harrison [29]在1998年报道过IkB蛋白质家族成员都含有6个ANK序列, 通过与 NFkB上的 RHR结构域发生作用, 抑制NFkB转入细胞核或者阻止 NFkB 与 DNA 的结合; 2000年 Foord等[30]研究出 Swi6/Cdc10家族蛋白质成员Swi4/6、Mbp1、Res1/2、Cdc10等都具有由5个ANK序列组成的核心结构域, 其参与整个细胞周期中的信号传导事件, 抑制CDK的活性, 从而影响转录激活。从这些研究中发现ANK结构域通过介导蛋白质之间的相互作用可以参与细胞内的信号传导、转录调控和发育调控等等。池蝶蚌Fem1c蛋白ANK重复序列结构域具有高度的保守性, 通过比较发现,不同物种的同源基因 ANK模体是各个序列中最保守的部分, 且这种保守性在 ANK家族蛋白中普遍存在, 从而认为 ANK模体在这类蛋白的生化功能上有一定的重要性[31]。 因此, 池蝶蚌fem-1c基因中ANK 模体的高程度出现, 可能在功能上与线虫fem-1基因的性别决定作用有一定的进化保守性。ANK模体在蛋白中多是以最少4个连续的ANK模体出现来调节蛋白间的相互间作用[32], 这说明在池蝶蚌中Fem1c蛋白可能也是通过与其他蛋白相互作用来参与调控。研究表明小鼠的Fem1a和Fem1b蛋白都编码一段相同的区域: 7个重复串联的ANK模体[33], 秀丽隐杆线虫的Fem1蛋白也有6个ANK模体[7], 人和斑马鱼的 Fem1蛋白也同样存在串联的ANK模体[1]。而这些物种的fem-1基因在性别决定信号通路上起着举足轻重的作用, 由此暗示了池蝶蚌fem-1c基因在细胞命运决定信号通路中具有进化保守性特征。
池蝶蚌fem-1c氨基酸序列与太平洋牡蛎fem-1c氨基酸序列同源性达到了 72%, 显示出了该蛋白在进化上的高度保守性。通过与选取的各个物种氨基酸序列比对发现, 它们在C端以一个含有11个氨基酸保守结构域终止, 这个结构域氨基酸序列为: Pro-X-X-Leu-X-X-Phe-X-X-X-His(X为随机氨基酸); 同时发现其中一些物种C端末为精氨酸残基, 在一定程度上我们可以通过这些物种相同序列后面的C端精氨酸残基来进行区分, C端精氨酸残基的功能目前并不清楚, 但是在这些物种中的保守性表明了它功能的重要性[1]。同时, 从无脊椎动物到脊椎动物fem-1c基因的保守性表明它们有共同的起源, 可能暗示这个基因家族在这些生物的发育或者生理功能上具备保守性, 推测池蝶蚌fem-1c基因可能具备与线虫fem-1基因在性别决定方面上相似的功能。
[1] Tereza V H, Deyin L, Si X, et al. The Fem1c genes: conserved members of the Fem1 gene family in vertebrates [J]. Gene, 2003, 314: 133—139
[2] Hodgkin J, Doniach T, Shen M. The sex determination pathway in the nematode Caenorhabditis elegans: variations on a theme [J]. Cold Spring Harb Symposia on Quantitative Biology, 1985, 50: 585—593
[3] Ahringer J, Rosenquist T A, Lawson D N. The Caenorhabditis elegans sex determining gene fem-3 is regulated post-transcriptionally [J]. The EMBO Journal, 1922, 11(6): 2303—2310
[4] Du H N, Hu H Y. Ankyrin repeat mediated protein-protein interaction [J]. Progress in Biochemistry and Biophysics, 2002, 29(1): 6—9 [杜海宁, 胡红雨. 锚蛋白重复序列介导的蛋白质-蛋白质相互作用. 生物化学与生物物理进展, 2002, 29(1): 6—9]
[5] Pilgrim D, McGregor A, Jackle P, et al. The C. elegans sex-determining gene fem-2 encodes a putative protein phosphatase [J]. Molecular Biology of the Cell, 1995, 6(9): 1159—1171
[6] Chin-Sang I D, Spence A M. Caenorhabditis elegans sex-determining protein FEM-2 is a protein phosphatase that promotes male development and interacts directly with FEM-3 [J]. Genes & Development, 1996, 10(18): 2314—2325
[7] Spence A M, Coulson A, Hodgkin J. The product of fem-1, a nematode sex-determining gene, contains a motif found in cell cycle control proteins and receptors for cell-cell interactions [J]. Cell, 1990, 60(6): 981—990
[8] Mehra A, Gaudet J, Heck L, et al. Negative regulation of male development in Caenorhabditis elegans by a protein–protein interaction between TRA-2A and FEM-3 [J]. Genes & Development, 1999, 13(11): 1453—1463
[9] Madl J E, Herman R K. Polyploids and sex determination in Caenorhabditis elegans [J]. Genetics, 1979, 93(2): 393—402
[10] Barton M K, Schedl T B, Kimble J. Gain-of-function mutations of fem-3, a sex-determination gene in Caenorhabditis elegans [J]. Genetics, 1987, 115(1): 107—119
[11] Liu H, Chen S L, Wang G X. Transformer genes and sex determination of C. elegans and D. melanogaster [J]. Hereditas, 2005, (1): 150—154 [刘辉, 陈思礼, 王国秀. Transformer基因与果蝇和线虫的性别决定. 遗传, 2005, (1): 150—154]
[12] Perry M D, Li W, Trent C, et al. Molecular characterization of the her-1 gene suggests a direct role in cell signaling during Caenorhabditis elegans sex determination [J]. Genes & Development, 1993, 7(2): 216—228
[13] Zarkower D, Hodgkin J. Molecular analysis of the C. elegans sex-determining gene tra-1: A gene encoding two zinc finger proteins [J]. Cell, 1992, 70(2): 237—249
[14] Kuwabara P E. A novel regulatory mutation in the C. elegans sex determination gene tra-2 defines a candidate ligand/ receptor interaction site [J]. Development (Cambridge, U.K.), 1996, 122(7): 2089—2098
[15] Sokol S B, Kuwabara P E. Proteolysis in Caenorhabditis elegans sex determination: cleavage of TRA-2A by TRA-3 [J]. Genes & Development, 2000, 14(8): 901—906
[16] Doniach T, Hodgkin J. A sex-determining gene, fem-1, required for both male and hermaphrodite development in Caenorhabditis elegans [J]. Developmental Biology, 1984, 106(1): 223—235
[17] Hodgkin J. Sex determination in the nematode C. elegans: analysis of tra-3 suppressors and characterization of fem genes [J]. Genetics, 1986, 114(1): 15—52
[18] He S H. Characteristics and expression analysis of twoferritin subunits from freshwater pearl mussel, Hyriopsis schlegelii [D]. Nanchang University. 2012 [何书豪. 池蝶蚌铁蛋白基因的特征与表达分析. 硕士学位论文, 南昌大学. 2012]
[19] Xu M X. The research on biological characteristics and pearl performance of Hyriopsis schlegelii [D]. Nanchang University. 2008 [徐毛喜. 池蝶蚌生物学特性及育珠性能研究. 硕士学位论文, 南昌大学. 2008]
[20] Wang B H. Study on Development of the gonad of Hyriopsis schlegelii [D]. Nanchang University. 2010 [王滨花. 池蝶蚌性腺发育的观察. 硕士学位论文, 南昌大学. 2010]
[21] Guo H J, Luo J, Hong Y J,et al.Growth and genetic analysis on the selected breeding Hyriopsis schlegelii [J]. Acta Hydrobiologica Sinica, 2008, 32(2): 220—224 [郭红军, 罗洁, 洪一江, 等. 人工选育池蝶蚌的生长及不同世代遗传分析. 水生生物学报, 2008, 32(2): 220—224]
[22] Wang Y F. Families selection and genetic analysis of growth traits for Hyriopsis schlegelii in the first generatioin [D]. Nanchang University. 2010 [汪勇飞. 池蝶蚌F1代家系选育与生长性状的遗传分析. 硕士学位论文, 南昌大学. 2010]
[23] Saitou N, Nei M. The neighbor—joining method: A new method for reconstructing phylogenetic trees EJ3 [J]. Mo1ecular Bio1ogy and Evo1ution, 1987, 4(4): 406—425
[24] Kozak M. Initiation of translation in prokaryotes and eukaryotes [J]. Gene, 1999, 234(2): 187—208
[25] Hodgkin J. Sex determination and dosage compensation in Caenorhabditis elegans [J]. Annual Review of Genetics, 1987, 21: 133—154
[26] Doniach T, Hodgkin J. A sex-determining gene, fem-1, required for both male and hermaphrodite development in Caenorhabditis elegans [J]. Developmental Biology, 1984, 106(1): 223—235
[27] Raymond C S, Shamu C E, Shen M M, et al. Evidence for evolutionary conservation of sex-determining genes [J]. Nature, 1998, 391(6668): 691— 695
[28] Russo A, Tong L, Pavletich N P, et al. Structural basis for inhibition of the cyclin-dependent kinase Cdk6 by the tumour suppressor p16INK4a [J]. Nature, 1998, 395(6699): 237—243
[29] Jacobs M D, Harrison S C. Structure of an IkBα/NF-αB complex [J]. Cell, 1998, 95(6): 749—758
[30] Foord R, Taylor I A, Smerdon S J, et al. X-ray structural analysis of the yeast cell cycle regulator Swi6 reveals variations of the ankyrin fold and has implications for Swi6 function [J]. Nature Structural Biology, 1999, 6(2): 157—165
[31] Bork P. Hundreds of ankyrin-like repeats in functionallydiverse proteins: Mobile modules that cross phylogenetic phylahorizontally [J]. Proteins-Structure Function and Genetics, 1993, 17(4): 363—374
[32] Michaely P, Bennett V. The ANK repeat: a ubiquitous motif involved in macromolecular recognition Trends [J]. Trends in Cell Biology, 1992, 2(5): 127—129
[33] Tereza V H, Seldin M F, Li W H, et al. The murine Fem1 gene family: homologs of the Caenorhabditis elegans sex-determination protein FEM-1 [J]. Genomics, 1998, 54(2): 221—230
STRUCTURE CHARACTERISTICS OF A MASCULINIZED FEM-1C GENE AND ITS PROTEIN FROM HYRIOPSIS SCHLEGELII
XIONG Wen-Fang, SHI Jian-Wu, JIANG Mou-Yan, CHEN Yong-Ling, PENG Kou, SHENG Jun-Qing, WANG Jun-Hua and HONG Yi-Jiang
(School of Life Sciences and Food Engineering, Nanchang University, Nanchang 330031, China)
We first reported the masculinized feminization gene (fem-1c) in the freshwater pearl mussel Hyriopsis schlegelii, which was cloned by Race-PCR from gonad tissue. Gene structure, multiple sequence homology alignment, transmembrane region, hydrophilia and hydrophobic property, and functional domain prediction was further analyzed using bioinformatics approaches. The full length of fem-1c gene was 2328 bp that contained an open reading frame of 1869 bp encoding 622 amino acid residues. The corresponding protein was stable and hydrophilic. Multiple sequence homology alignment showed that the Fem-1c proteins from H. schlegelii and C. gigas shared the highest amino acid identity (79%). The secondary structure of fem-1c protein mainly contained α-helix and random coil forming 9 ankyrin repeat (ANK) motifs. We also found 9 ANK motifs clearly in tertiary structure prediction by SMART soft. The conservation of the fem-1c from invertebrates to vertebrates suggested that these genes have a common ancient origin and that they may play a conserved role in the functions of these organisms. These results suggest that fem-1c gene in Hyriopsis schlegelii may have the similar functions with nematode Caenorhabditis elegans in sex determination.
Hyriopsis schlegelii; fem-1c gene; Clone; Sequence analysis; Structure Prediction
Q781
A
1000-3207(2014)06-1002-06
10.7541/2014.148
2013-10-20;
2014-03-22
国家自然科学基金(31160534); 江西省科技落地计划(12001); 江西省科技支撑计划项目(20112BBF60013, 20121BBF 60036); 江西省自然科学基金(20122BAB204016); 江西省教育厅科技项目(GJJ12020) 资助
熊文芳(1989—), 女, 江西丰城人; 硕士; 主要研究方向为水生生物免疫与保护学。E-mail: xwfpepo1989@163.com
洪一江(1963—), 男, 福建南安人; 教授, 博导; 主要研究方向为水生动物遗传育种学。E-mail: yijianghong@126.com
