鳙Sox基因克隆及序列进化分析
2014-03-29郭稳杰俞小牧童金苟
郭稳杰俞小牧童金苟
(1. 中国科学院水生生物研究所, 武汉 430072; 2. 中国科学院大学, 北京 100049)
鳙Sox基因克隆及序列进化分析
郭稳杰1,2俞小牧1童金苟1
(1. 中国科学院水生生物研究所, 武汉 430072; 2. 中国科学院大学, 北京 100049)
利用简并引物SoxN和Sox9在鳙基因组DNA中进行PCR扩增和产物克隆测序, 并对序列进行同源性比较和系统进化分析。结果表明本文鉴定出鳙15个Sox基因HMG盒序列, 分别属于SoxB、SoxC和SoxE组, 依据斑马鱼同源基因将其分别命名为Sox1a、Sox1b、Sox2、Sox3、Sox4a、Sox4b、Sox9a、Sox9b、Sox10、Sox11b、Sox12、Sox14a、Sox14b、Sox19和Sox21a。基于鳙和斑马鱼 Sox1、Sox4和Sox9 基因核苷酸序列构建的系统进化树显示这3个Sox基因的复制时间发生在鳙和斑马鱼的分化之前, 结果支持了鱼类特异的基因组复制假说。以Sox1a、Sox1b和Sox4基因为分子钟标记构建系统进化树探讨鳙和斑马鱼的分化时间, 结果显示, 同属于鲤科鱼类的鳙(鲤亚科)和斑马鱼(鱼丹亚科)在原始的鱼丹亚科鱼类中存在一个共同祖先, 大约出现在63.7百万年前。研究结果为进一步研究鱼类Sox基因复制和基因组进化等问题提供了重要参考资料。
鳙; Sox基因; 基因组加倍; 系统进化树; 分化时间
Sox(Sry-related HMG-box)是一类与哺乳动物性别决定基因Sry (Sex determining region of Y chromosome)相关基因构成的基因家族。该家族成员都含有一个保守的HMG (High mobility group) 盒, 其编码产物能特异性地识别和结合 DNA序列, 使DNA发生弯曲, 是一类重要的转录调控因子[1]。Sox基因广泛参与神经系统发育、血细胞生成、晶状体发育以及性别决定和分化等重要的生物学过程。例如, SOX9与SRY共同作用调控睾丸的发育[2]; Sox1、Sox2、Sox3、Sox4和Sox11等参与神经系统的发育调控[3—5]。目前, Bowles, et al.[6]根据HMG盒的相似性, 将Sox基因分成A-J 10个亚族。
由于在进化过程中基因加倍等原因, 鱼类和哺乳动物的Sox基因家族成员有所差别。例如, Sox1、Sox4、Sox6、Sox8、Sox9、Sox11、Sox14和 Sox21在人类和小鼠基因组中只存在一个拷贝; 而在斑马鱼中, 这些基因被证实有两个旁系同源基因[7](Paralogous genes)。关于鱼类 Sox基因复制的原因尚无定论, 主要包括全基因组复制和基因片段复制两种观点[8]。目前许多研究都支持 Sox基因加倍是全基因组复制的结果[9,10]。
研究表明利用碱基同义突变速率和分子钟模型构建的系统进化树, 能够估计物种间的大致分化时间[11,12]。由于Sox基因广泛存在于脊椎动物基因组中, 且易于克隆, 成为物种系统进化研究非常重要的材料。鲤科鱼类(Cyprinidae)是现有鱼类中最大的一个科, 也是生物学家探索水生动物进化极好的代表群体[13]。目前, 已有些研究利用 Sox基因估算鲤科鱼类物种间分化时间[9,10]。鳙(Aristichthys nobilis)属于鲤形目鲤科鲢亚科, 是新近形成的一个物种[14],对其Sox基因HMG盒进行克隆和序列进化分析, 可为探索鱼类Sox基因加倍机制和鲤科鱼类系统进化研究提供很好的分子资料。
1 材料与方法
1.1 材料和DNA提取
实验样本采自武汉涨渡湖渔场, 剪取一尾鳙少量鳍条组织浸泡在 95%乙醇中, 然后置于 4℃下保存备用。采用经典的酚氯仿法[15]提取基因组DNA。
1.2 基因扩增、克隆和测序
针对脊椎动物Sox基因HMG盒保守的氨基酸序列, 设计两对简并引物 SoxN和 Sox9[16], 由上海生工生物工程技术服务有限公司合成。引物对SoxN序列为5′-ATGAAYGCNTTYATGGTNTGG-3′和5′-GGNCGRTAYTTRTARTCNGG-3′, 引物对Sox9序列为 5′-ATGAAYGCSTTYATGGTITGG-3′和 5′-GTCIGGGTGRTCYTTCTTRTGYTG-3′。PCR扩增反应体系总体积为50 µL, 包括160—200 ng的基因组DNA,上下游引物(5 µmol/L)各 2 µL, 1.6 µL dNTP (2.5 mmol/L), 0.4 µL Taq DNA酶(5 U/µL)(Takara), 1.25 µL 10×Buffer, 最后补足灭菌双蒸水至终体积。PCR反应条件如下: 94℃预变性4min; 扩增35个循环, 每个循环 94℃变性 1min、53℃退火 1min、72℃延伸1min; 72℃终延伸10min。PCR扩增产物经1%的琼脂糖凝胶检测后, 并用 Biospin胶回收试剂盒(BioFlux, China)纯化回收扩增产物。将回收产物连接到 pMD18-T克隆载体上(Takara, Japan), 再转化到大肠杆菌DH5α, PCR检测阳性克隆, 交由北京诺赛基因组研究中心有限公司测序。
1.3 序列进化分析
本文分析中所用到的斑马鱼(Danio rerio)、红鳍东方 鲀(Temmincket Schlegel)、海鲈(Dicentrarchus labrax)一些Sox基因序列的来源见表1。
Sox基因的鉴定 将引物序列从测得的基因序列中去除, 然后用 Clustalx 1.81[17]软件进行序列比对, 根据比对结果选取有一定变异的一组核酸序列作基因鉴定分析。将筛选出的核酸序列用MEGA 4.0[18]软件翻译成氨基酸序列, 在 GenBank数据库中进行同源性搜索, 鉴定Sox基因。
系统进化树分析 采用MEGA 4.0软件, 用斑马鱼Sox基因和所有鉴定出的鳙Sox基因氨基酸序列一起进行最小进化法(Minimum-evolution method, ME)分析, 并以人的TCF7基因(MN_201632)为外类群。
Sox基因复制时间的推算 由于密码子具有兼并性, 通常氨基酸第 3个密码子碱基置换不改变氨基酸序列, 其替换速率能全面地反映基因突变率,可用于编码基因的进化分析[11,19]。本研究以鳙和斑马鱼Sox1、Sox4和Sox9基因核苷酸序列为材料, 根据第3位密码子碱基置换速率, 利用ME法构建系统进化树, 推算Sox基因的复制时间。

表1 本研究用于序列分析的Sox基因Tab. 1 Sox genes used for sequence analysis in the study
鳙与斑马鱼分化时间估算 本研究以鳙、斑马鱼、红鳍东方 鲀和海鲈Sox1a、Sox1b和Sox4基因数据集为分子钟标记, 根据第 3位密码子碱基置换速率, 利用非加权组平均法(UPGMA method)构建系统进化树, 估算鳙和斑马鱼的分化时间。
2 结果
2.1 基因扩增、克隆与测序
在鳙基因组DNA中, SoxN引物扩增出一条约为220 bp的条带; Sox9引物扩增出大小不等的4条带, 分别为200、300、700和1200 bp左右。对这些扩增产物进行克隆, 经菌落PCR鉴定, 共挑选60个阳性克隆进行测序。
2.2 序列分析
鳙Sox基因命名及与斑马鱼蛋白的系统进化树分析 经Clustal序列比对和Blast同源性比较, 共鉴定出15个鳙Sox基因, 分别命名为: Sox1a、Sox1b、Sox2、Sox3、Sox4a、Sox4b、Sox9a、Sox9b、Sox10、Sox11b、Sox12、Sox14a、Sox14b、Sox19和 Sox21a (GenBank登录号为: KC883624-KC883629, KC867495,KC867494, KC867496, KC883630-KC8836 35)。
以鳙 Sox基因氨基酸序列与斑马鱼相应的 Sox蛋白以ME法构建系统进化树(图1)。结果显示, 本文所得到的15个鳙Sox基因归属于SoxB、SoxC和SoxE三个亚族, 其中引物 SoxN扩增所得到的鳙Sox基因属于SoxB和SoxC, 引物Sox9扩增得到的鳙Sox基因属于SoxB和SoxE组。
Sox基因复制时间的推算 为了推算Sox基因的复制时间, 以鳙和斑马鱼 Sox1、Sox4和 Sox9基因的核苷酸序列第 3位密码子碱基置换速率, 利用ME法构建了系统进化树(图2)。系统进化树结果显示, Sox1、Sox4和Sox9基因的复制时间发生在鳙和斑马鱼的分化之前。
鳙与斑马鱼的分化时间 利用鳙、斑马鱼、红鳍东方 鲀和海鲈Sox1a、Sox1b和Sox4基因数据集第3位密码子碱基置换速率, 采用UPGMA法构建系统进化树, 以期根据这些基因的进化推测物种间的分化时间。以斑马鱼与红鳍东方 鲀的分化时间(290百万年前)为标定点[20], 利用以 Sox1a、Sox1b及 Sox4基因为分子钟标记所构建的系统进化树(图 3), 推测鳙(鲤形目, 鲤科, 鳙属)与斑马鱼(鲤形目,鲤科, 鱼丹属)的分化发生在63.7百万年前。

图2 利用鳙和斑马鱼Sox1、Sox9和Sox11基因第3位密码子碱基替换速率构建系统进化树Fig. 2 Phylogenetic tree of Sox1, Sox9 and Sox11 genes from bighead carp and zebrafish using third-codon position substitution rates
3 讨论
3.1 鳙Sox基因HMG盒序列克隆用于Sox基因家族进化的研究
Sox基因家族成员最显著特征是含有一个保守的HMG盒, 可以根据HMG盒序列进行Sox基因的鉴定及其家族进化的研究。利用SoxN和Sox9两对简并引物对鳙基因组 DNA进行扩增, 为了保证测序结果的可信度, 本文鉴定出的每个 Sox基因序列至少是两个不同克隆测序结果。测得的序列经blast比对分析, 我们共鉴定出15个鳙Sox基因, 分属于SoxB、SoxC和SoxE。有趣的是, 归属于SoxB亚族的Sox19基因, 在哺乳动物中不存在其同源基因[21],而在斑马鱼[22]和 红鳍东方 鲀[23]等真骨鱼中则存在其同源基因。Sox19基因在哺乳动物基因组中的缺失可能是在进化过程中, 哺乳动物丢失了该基因,也可能是真骨鱼类通过基因复制逐渐由某一基因的复制子演化成Sox19基因。Koopman, et al.[23]认为Sox19基因很可能是由Sox3基因的一个复制子进化而来, 图1中Sox3和Sox19的进化关系可能是这种观点的一个佐证。鳙这些Sox基因的鉴定将有助于硬骨鱼类Sox基因家族的进化研究。
3.2 Sox基因的复制及其复制时间
在鱼类基因组研究中已经发现基因复制是非常常见的现象, 但重复基因在不同的物种中有不同的命运, 主要包括选择性丢失或协同进化产生新的功能[24,25]。Blast同源性比较和进化树分析表明, Sox1、Sox4、Sox9和Sox14在鳙基因组中发生了基因复制。其中Sox1、Sox4和Sox9的复制并没有改变其氨基酸序列, 说明这3个Sox基因在进化过程中受到很大的选择压力, 可能在鳙个体发育过程中发挥重要的调控作用。如果在辐鳍鱼进化历程中鱼类特异的全基因组复制事件存立的话, 那么这次事件一定发生在鳙和斑马鱼分化之前。然而, 关于鱼类特异性的Sox基因复制, 是全基因组复制还是Sox基因片段复制的结果, 迄今还没有定论。本文的Sox基因复制时间估算结果表明, Sox1、Sox4和Sox11基因的复制时间确实发生在鳙和斑马鱼的分化之前(图 2), 这也在一定程度上支持了“鱼类特异性的基因组复制”学说[26]。
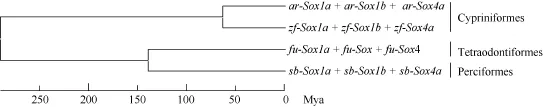
图3 利用鳙、斑马鱼、红鳍东方 鲀和海鲈Sox1a、Sox1b和Sox4基因数据集第3位密码子碱基替换速率构建系统进化树Fig. 3 Phylogenetic tree of concatenated dataset of Sox1a, Sox1b and Sox4 genes from bighead carp, zebrafish, fugu and sea bass using third-codon position substitution rates
3.3 鳙和斑马鱼的分化时间
鳙是鲤科鱼类进化过程中新近形成的一个物种,对其Sox基因HMG盒进行克隆和序列分析, 可为研究鱼类Sox基因进化历程提供较好的分子信息。陈宜瑜等[14]?通过对现有鱼类化石资料的观察和鉴定认为这些化石大都属于原始的 鲃亚科和鱼丹亚科, 且都出现在距今65百万年至距今26百万年的老第三纪。由于地质的一系列变化带来温暖地带向南退缩,生活在热带和亚热带的原始鱼丹亚科不适应气候变化在北方几近灭绝, 从而派生出适应寒冷气候的雅罗鱼亚科鱼类成为鲤科鱼类重要的组成成分。随着青藏高原急剧隆升, 形成了长江中下游河流纵横交错的复合生态系统, 由适应较冷气候环境的原始雅罗鱼亚科, 逐步派生出 鲌亚科和鲢亚科。综上所述,鳙、草鱼和斑马鱼在原始鱼丹亚科中存在一个共同的祖先。本研究以Sox1a、Sox1b及Sox4基因为分子钟标记, 推测鳙(鲤形目, 鲤科, 鲢亚科, 鳙属)与斑马鱼(鲤形目, 鲤科, 鱼丹亚科, 鱼丹属)共同的祖先大约存在于63.7百万年前, 正好处于老第三纪的古新世(Paleocene)(距今65百万年至53百万年)。本文的这个推算结果与Zhong, et al.[10]研究草鱼(鲤形目, 鲤科, 雅罗鱼亚科, 草鱼属)和斑马鱼的分化时间(63百万年前左右)一致, 从而从基因进化的角度证实了陈宜瑜等[14]对鲤科鱼类物种进化的推断。
[1] Rehberg S, Lischka P, Glaser G, et al. Sox10 is an active nucleo-cytoplasmic shuttle protein and shuttling is crucial for Sox10-mediated transactivation [J]. Molecular and Cellular Biology, 2002, 22: 5826—5834
[2] Zanaria E, Muscatelli F, Bardoni B, et al. An unusual member of the nuclear hormone receptor superfamily responsible for X-linked adrenal hypoplasia congenita [J]. Nature, 1994, 372: 635—641
[3] Uwanogho D, Rex M, Cartwright E J, et al. Embryonic expression of the chicken Sox2, Sox3 and Sox11 genes suggests an interactive role in neuronal development [J]. Mechanisms of Development, 1995, 49(1): 23—36
[4] Collignon J, Socknathan S, Hacker A, et al. A comparison of the properties of Sox3 with Sry and two related genes Sox1 and Sox2 [J]. Development, 1996, 122(2): 509
[5] Cheung M, Abu-Elmagd M, Clevers H, et al. Roles of Sox4 in central nervous system development [J]. Molecular Brain Research, 2000, 79(1): 180—191
[6] Bowles J, Schepers G, Koopman P. Phylogeny of the Sox family of developmental transcription factors based on sequence and structural indicators [J]. Development Biology, 2000, 227(2): 239—255
[7] Okuda Y, Yoda H, Uchikawa M, et al. Comparative genomic and expression analysis of group B1 sox genes in zebrafish indicates their diversification during vertebrate evolution [J]. Developmental Dynamics, 2006, 235(3): 811—825
[8] Vandepoele K, De Vos W, Taylor J S, et al. Major events in the genome evolution of vertebrates: paranome age and size differ considerably between ray-finned fishes and land vertebrates [J]. Proceedings of the National Academy of the Sciences ot the United States of America, 2004, 101(6): 1638—1643
[9] Zheng J, Long L, Hu J, et al. Isolation and sequence analysis of Sox genes in the red crucian carp (Carassius carassius, red variety) [J]. Russian Journal of Genetics, 2008, 44(11): 1325—1330
[10] Zhong L, Yu X M, Tong J G. Sox genes in grass carp (Ctenopharyngodon idella) with their implications forgenome duplication and evolution [J]. Genetics Selection Evolution, 2006, 38: 673—687
[11] Nei M, Rogozin L B, Piontkivska H, et al. Purifying selection and birth-and-death evolution in the ubiquitin gene family [J]. Proceeding of the National Academy of the Sciences, 2000, 97(20): 10866—10871
[12] Nei M, Kumar S. Molecular Evolution and Phylogenetics [M]. New York: Oxford University Press. 2000, 203—206
[13] 韩亚伟, 童超波, 何舜平, 等. 鲤科鱼类系统进化过程中SINEs的插人事件. 自然科学进展, 2007, 17(10): 1345—1351
[14] Chen Y Y, Cao W X, Zheng C Y. Ichthyofauna of the Zhujiang river with a discussion on zoogeographical divisions for freshwater fishes [J]. Acta Hydrobiologica Sinica, 1986, 10(3): 228—236 [陈宜瑜, 曹文宣, 郑慈英.珠江的鱼类区系及其动物地理区划的讨论. 水生生物学报, 1986, 10(3): 228—236]
[15] Taggart J B, Hynes R A, Prodöuhl P A, et al. A simplified protocol for routine total DNA isolation from salmonid fishes [J]. Journal of Fish Biology, 1992, 40(6): 963—965
[16] Galay-Burgos M, Llewellyn L, Mylonas C C, et al. Analysis of the Sox gene family in the European sea bass (Dicentrarchus labrax) [J]. Comparative Biochemistry and Physiology Part B: Biochemistry and Molecular Biology, 2004, 137(2): 279—284
[17] Thompson J D, Gibson T J, Plewniak F, et al. The Clustal_X Windows interface: Flexible strategies for multiple sequences alignment aided by quality analysis Tools [J]. Nucleic Acids Research, 1997, 25(24): 4876—4882
[18] Tamura K, Dudley J, Nei M, et al. Mega 4: Molecular evolutionary genetics analysis (Mega) software version 4.0 [J]. Molecular Biology and Evolution, 2007, 24(8): 1596—1599
[19] Hughes A L. Adaptive Evolution of Genes and Genomes [M]. New York: Oxford University Press. 1999, 15—37
[20] Kumazawa Y, Yamaguchi M, Nishida M. The Biology of Biodiversity [M]. Hong Kong: Springer-Verlag. 1999, 35—52
[21] Hett A K, Ludwig A. SRY-related (Sox) genes in the genome of European Atlantic sturgeon (Acipenser sturio) [J]. Genome, 2005, 48(2): 181—186
[22] Vriz S, Lovell-Badge R. The zebrafish Zf-Sox 19 protein: A novel member of the Sox family which reveals highly conserved motifs outside of the DNA-binding domain [J]. Gene, 1995, 153(2): 275—276
[23] Koopman P, Schepers G, Brenner S, et al. Origin and diversity of the Sox transcription factor gene family: Genome-wide analysis in Fugu rubripes [J]. Gene, 2004, 328: 177—186
[24] Postlethwait J, Amores A, Cresko W, et al. Subfunction partitioning, the teleost radiation and the annotation of the human genome [J]. Trends in Genetics, 2004, 20(10): 481—490
[25] Maere S, De Bodt S, Raes J, et al. Modeling gene and genome duplication in eukaryotes [J]. Proceedings of the National Academy of Sciences of the United States of America, 2005, 102(10): 5454—5459
[26] Zhou L, Wang Y, Gui J F. Fish-specific genome duplication [J]. Zoological Research, 2006, 27(5): 525—532 [周莉, 汪洋, 桂建芳. 鱼类特异的基因组复制. 动物学研究, 2006, 27(5): 525—532]
CLONING AND SEQUENCE EVOLUTION ANALYSIS OF SOX GENES IN BIGHEAD CARP (ARISTICHTHYS NOBILIS)
GUO Wen-Jie1,2, YU Xiao-Mu1and TONG Jin-Gou1
(1. Institute of Hydrobiology, Chinese Academy of Sciences, Wuhan 430072, China; 2. University of Chinese Academy of Sciences, Beijing 100049, China)
To amplify Sox genes from bighead carp genome, two pairs of degenerate primers (SoxN and Sox9) were designed for PCR amplification that were utilized for sequencing and analysis of sequence homology and phylogenetic relationships. The results showed that 15 distinct Sox genes encoding the HMG domains were identified in bighead carp, which were assigned to group B, C and E. According to their homology to orthologs of zebrafish, 15 Sox genes were designated as Sox1a, Sox1b, Sox2, Sox3, Sox4a, Sox4b, Sox9a, Sox9b, Sox10, Sox11b, Sox12, Sox14a, Sox14b, Sox19 and Sox21a, respectively. Phylogenetic tree of Sox1, Sox4 and Sox9 nucleotide sequences indicated that the duplication of these three Sox genes occurred before the divergence of bighead carp and zebrafish, and this observation supported the“fish-specific whole-genome duplication” theory. Using Sox1a, Sox1b and Sox4 as molecular clock markers in phylogenetic analysis, the estimation of the divergence time between bighead carp and zebrafish demonstrated that in original Danioninae, a common ancestor appeared approximately 63.7 million years ago for bighead carp and zebrafish, both of which belong to Cyprinidae. This study would provide important information for further studies of Sox gene replication and genome evolution in fish.
Aristichthys nobilis; Sox gene; Genome duplication; Phylogenetic tree; Divergenc time
Q781
A
1000-3207(2014)04-0664-05
10.7541/2014.94
2013-04-23;
2014-01-09
公益性(农业)行业科研项目(200903045); 国家自然科学基金项目(31272647)资助
郭稳杰(1985—), 男, 湖北黄梅县人; 博士研究生; 主要从事鱼类遗传与基因组学研究。E-mail: chaliangle12@163. com
童金苟, E-mail: jgtong@ihb.ac.cn
