水痘带状疱疹病毒基因亚型的研究进展
2014-03-27屈园园综述普雄明审校
屈园园(综述),普雄明(审校)
(新疆维吾尔自治区人民医院皮肤性病科,乌鲁木齐 830001)
水痘带状疱疹病毒(varicella-zoster virus,VZV)属人类疱疹病毒α亚科,又称人类疱疹病毒3型(human herpes virus typs 3,HHV-3),是水痘和带状疱疹的病原体。Boot等[1]利用水痘病毒Oka株(Oka parent,pOka)成功研制出水痘减毒活疫苗(Oka vaccine,vOka),预防接种可显著减少儿童水痘的发生率。疫苗接种者仍可发生水痘样皮疹,接种的病毒疫苗也可潜伏体内而引发带状疱疹[2]。疫苗接种后发生的水痘或带状疱疹既可为VZV野生株感染引起,也可为pOka或vOka感染引起[3]。区分由野生病毒或偶尔有疫苗病毒引起的感染,对于基因型与地理分布、疫苗质量及接种效果评价等具有重要意义。VZV只有一个血清型,但不同毒株间基因序列仍有一定的差异。利用VZV毒株间基因序列差异可进行基因分型,而VZV毒株的基因分型研究对VZV感染的流行病学研究等具有重要意义。
1 VZV基因组结构
1986年Davison等[4]用双脱氧核苷酸技术测序获得了VZV Dumas株的全基因组序列。以后的研究又获得了不同的VZV株(如VZV Oka疫苗株)、VZV-MSP、VZV-BC、VZV-CA123等基因组序列[5-7]。VZV基因组为线性双链DNA分子,长约124 884 bp,由共价连接的长片段(L)和短片段(S)组成。长片段(UL)长约10 500 bp,两端有小片段反向重复序列(TRL和IRL),短片段(US)长约5232 bp,两端有大片段反向重复序列(TRS和IRS)。因此,VZV基因组的结构模式为TRL-UL-IRL-IRS-US-TRS。
VZV基因组由71个开放读码框(open reading frame,ORF)组成,约编码69种蛋白,其中ORF42和ORF45接合转录、翻译成一种蛋白产物。另外,ORF62、ORF63、ORF64在TRS和IRS区分别重复一次即形成ORF71、ORF70、ORF69,因此VZV基因组实际包含69个独特基因[8]。VZV基因组中还包含5个富含鸟嘌呤和胞嘧啶的串联重复序列(R1-R5)。不同分离株R区串联重复单位拷贝数存在差异,所以R区亦称R可变区。R1、R2、R3区分别位于ORF11、ORF14、ORF22的基因编码区,R4区位于IRS和TRS两个区,分别在ORF62与ORF63之间和ORF70与ORF71之间;R5位于ORF60和ORF61之间。
2 VZV基因组变异
虽然VZV基因组具有较好的遗传稳定性,但不同毒株间基因组仍存在变异,VZV毒株间基因变异可达500个碱基,且多位于R1-R5及病毒复制起点Oris[9]。不同毒株间基因变异主要表现为单核苷酸多态性(single nucleotide polymorphism,SNP)。通过选择不同的SNP位点及其组合进行分析,可将不同国家或地区的VZV分离株分为不同的基因型。
3 VZV基因分型技术
常用于VZV基因分型的技术包括限制性片段长度多态性(restriction fragment length polymorphic,RFLP)和聚合酶链反应(Polymerase Chain Reaction,PCR)-RFLP分析、实时PCR、核酸测序分析、系统进化树分析、异源双链泳动法、寡聚核苷酸微阵列分析、二分检索等。
3.1RFLP和PCR-RFLP分析 RFLP最早用于证实VZV为引起水痘和带状疱疹的病原体,并用于区分VZV野生株和疫苗株,也为VZV基因的高度保守性提供了依据。常用于VZV基因分型的酶切位点包括ORF6中AluⅠ(SNP5745)、ORF21中Sau96Ⅰ(SNP33646)、ORF38中PstⅠ(SNP69349)、ORF54中BglⅠ(SNP95241)、ORF62中SmaⅠ(SNP106262)、NaeⅠ(SNP107252)和BssHⅡ(SNP107136),其中PstⅠ、BglⅠ、SmaⅠ最为常见[10]。PstⅠ酶切位点为大部分野生株共有,BglⅠ和SmaⅠ酶切位点为Oka株独有。在北美和欧洲,绝大多数VZV病毒株在ORF38中(SNP69349)都含有Pst Ⅰ 酶切位点(PstⅠ+),在ORF54中(SNP95241)不含Bgl Ⅰ 酶切位点(BglⅠ-),但在日本有30%的病毒株为PstⅠ-,随后PstⅠ-在美国夏威夷和澳大利亚也相继被发现[7,11]。我国在内的其他亚洲国家也可能存在类似情况,故ORF38中的PstⅠ位点不适用于区分日本流行的野毒株和疫苗株[12]。SmaⅠ、NaeⅠ、BssHⅡ酶切位点位于ORF62,为疫苗株独有,借助ORF62 PCR产物的酶切图谱即可鉴别vOka和pOka[11,13]。RFLP方法简便,但要求DNA量大、纯度高、且酶切图谱分辨率差。现多与PCR相结合(PCR-RFLP),即先用PCR扩增包含待测位点的DNA片段后用限制性内切酶酶切,根据PCR产物的酶切图谱进行基因分析。PCR-RFLP操作简单,费用低廉,适用于大样本分析,但只能检测已知突变,因而多用于区分野生株和疫苗株。如待测样本同时含有野生株和疫苗株,酶切后可能出现复杂条带而影响判断。PCR-RFLP需要多种限制性内切酶,如病毒基因变异不在限制性内切酶的切点范围,或使用的内切酶数量不够,也可造成漏检或分型不准确。
3.2实时荧光定量PCR 用Light-Cycler实时PCR检测VZV阳性标本ORF62中SNP106262和ORF38中SNP69649,结果与PCR-RFLP分析结果一致,两者均可用于鉴别VZV疫苗株和野生株[14]。与 PCR-RFLP相比,实时PCR无需酶切位点存在,操作更简单、快速、杂交探针特异性更强,而且PCR扩增和产物荧光检测均在闭管状态下进行,可避免扩增产物污染所致假阳性。
3.3核酸测序 目前已完成全基因组测序的VZV共有23株,均可在基因库获得其全基因组序列[15]。对编码VZV的一些糖蛋白和ORF62的DNA序列分析,已作为一种新的分析技术用于VZV基因分型。Faga等[16]对10株VZV的5种糖蛋白(gH/ORF37、gI/ORF67、gL/ORF60、gB/ORF31和gE/ORF68)以及ORF62基因的全序列进行分析,得出A,B,C,D四个基因型。核算测序最近被改进并应用于新加坡和泰国的VZV株分析[17]。核酸序列分析用于VZV基因分型最精确,但技术要求高、费用高,需反复比较分析才能得出结论,难以用于大规模检测。
3.4系统进化树分析 在已有VZV基因组序列基础上,利用系统进化树分析即可对VZV进行基因分型。Loparev等[11]运用系统进化分析结合其他分析技术对来自不同人群的大量临床样本进行了VZV多态性和多样性分析,得到E,J,M三个主要基因型。系统进化树分析还可用于验证其他基因分型方法的准确性,其发展得益于大规模测序和分析技术的完善,但它只能在拥有大量序列信息的基础上去做回顾性分析。
3.5异源双链泳动法 异源双链泳动法是在PCR反应体系中,使用同一对引物扩增不同来源的DNA片段后,形成相同大小的同源和异源双链DNA。异源双链DNA的泳动速度与其双链同源性呈正比,同源性越高则泳动越快。通过比较待测样品与VZV亚型的异源双链DNA泳动率可对VZV进行基因分型。Barrett-Muir等[18]应用异源双链泳动法技术对ORF1、ORF21、ORF50、ORF54中的SNP位点,对VZV英国分离株进行分析时,得到VZV4个主要基因型,分别为A型(非洲、亚洲)、J型(日本)、B型和C型(欧洲)。异源双链泳动法技术作为一种快速、简便、特异和经济的VZV基因分型法,可用于大样本毒株的基因分型,准确性比核酸测序法略差,但两者结果仍高度一致。
3.6寡核苷酸微阵列分析 寡核苷酸微阵列分析又称基因芯片法,是将大量特定寡核苷酸片段作为基因分型探针高密度而有序地固定于特制载玻片上,与荧光标记的待测核酸样品杂交后,通过激光共聚焦系统检测杂交信号强度,经计算机分析处理获取样品杂交信号的数量和序列信息,从而对核酸序列进行大规模和高通量分析。Sergeev等[19]应用此方法分析位于VZV ORF22中的5个可变点,分析了VZV参考株6株和2001~2002年美国临床分离株130株的基因型,结果与用其他方法的分型结果一致,可快速、灵敏地将VZV分为E型(81.5%),J型(3.0%)和M型(15.5%)三种主要基因型,另外还检测到M型的新亚型。基因芯片技术分型的灵敏度高,且杂交片洗涤后可直接进行扫描,操作简便,发展潜力巨大,但需要特殊设备,且费用昂贵。
3.7二分检索 二分检索又称折半检索,适用于不经常变动而检索频繁的有序列表。Schmidt-Chanasit等[20]用二分检索分析法选择VZV基因组中从ORF51~ORF58四个位点,将德国野生株和Oka疫苗株进行对比分析,结果显示德国野生株中33%为A型,67%为D型;Oka疫苗株为B型,他们用同样方法分析了其他地方分离株,得到的结果与用其他分析方法(如异源双链泳动法和系统进化分析)所得的结果一致。二分检索的优点是比较次数少,检索速度快,平均性能好;缺点是要求待查表为有序表,且插入删除困难。
4 VZV基因分型方案
目前VZV基因分型方案多基于SNP进行。较常用的方案有基于散在SNP方案:Barrett-Muirt等[18]分析VZV ORF1、ORF21、ORF50、ORF54中的SNP,把在英国流行的VZV株分为A型、B型、C型和J型四个基因型,B型可能是A型和C型的组合。基于ORF22方案:美国疾病控制与预防中心的Loparev等[11]对从27个国家分离得到的326株VZV的ORF22进行测序分析,并根据其中的6个SNP位点(37902,38019,38055,38081,38177和38229)将这些毒株划分为三个基因型,分别命名为E,J,M型,其中与Dumas株同源者定为E型,与pOka株同源者定位J型,介于两者间者定为M型,E型、J型间差异最大,M型介于两者之间并被认为是E型和J型的组合,ORF22 SNP方案如联合ORF21或ORF50中SNP,可区分出五个主要基因型(E1,J,E2,M2,M1)和两个次要基因型(M4和M3),其对应的VZV基因型通用名即进化枝1,2,3,4,5,Ⅵ,Ⅶ[6]。基于糖蛋白/IE62方案:Faga等[16]分析了gB(ORF31)、gE(ORF68)、gH(ORF37)、gI(ORF67)、gL(ORF60)和IE62(ORF62)6个基因多态性位点后,将VZV分为A,B,C和D四个基因型,该方法被改进后还用于新家坡和泰国VZV毒株基因分型[15]。
2008年英国伦敦举行的VZV基因型命名专题会议上建议根据VZV全基因组序列统一将VZV分成五个进化枝(即进化枝1,2,3,4和5)和临时进化枝(进化枝Ⅵ和Ⅶ)[15]。VZV各进化枝对应的参考毒株及四种基因分型方案之间的关联见表1[21]。
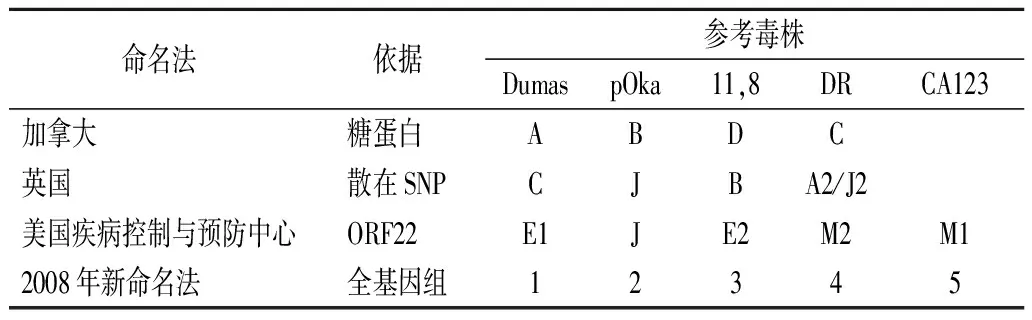
表1 VZV参考毒株及四种基因分型方案之间的关联
VZV基因型在世界各地分布具有显著的地域特征,其基因型的变化还可以在一定程度上反映移民情况,如在移民比较多的国家(如美国、英国和巴西等)VZV基因型尽管以B和C为主,但是所有其他基因型均可被检出[21]。
5 展 望
VZV基因分型的意义主要体现在流行病学和疫苗研制方面。在流行病学上,VZV基因分型可以帮助掌握VZV分子流行病学特征,迅速查明毒株来源,追踪VZV在易患人群中的传播状况,切断传播路线,控制疫情的蔓延,同时有助于解释不通患者特有的临床表现;在疫苗研制和使用上,水痘疫苗要想达到对某个地区最大的保护作用,就必须知道该地区VZV基因型,做到疫苗的特异使用。基因分型与地理分布、病毒感染与疫苗预防效果之间的关系,还需要进行更大样本研究。随着分子生物学的发展,对VZV基因型的研究将更加深入,这对了解各个国家VZV分子流行病学特点,探索其发病机制,以及感染的预防及治疗都具有重要意义。
[1] Boot HJ,de Melker HE,Stolk EA,etal.Assessing the introduction of universal varicella vaccination in the Netherlands[J].Vaccine,2006,24(37/39):6288-6299.
[2] Oxman MN,Levin MJ,Johnson GR,etal.A vaccine to prevent herpes zoster and postherpetic neuralgia in older adults[J].N Engl J Med,2005,352(22):2271-2284.
[3] Quinlivan ML,Gershon AA,Steinberg SP,etal.Rashes occurring after immunization with a mixture of viruses in the Oka vaccine are derived from single clones of virus[J].J Infect Dis,2004,190(4):793-796.
[4] Davison AJ,Scott JE.The complete DNA sequence of varicella-zoster virus[J].J Gen Virol,1986,67(Pt 9):1759-1816.
[5] Gomi Y,Sunamachi H,Mori Y,etal.Comparison of the complete DNA sequences of the Oka varicella vaccine and its parental virus[J].J Virol,2002,76(22):11447-11459.
[6] Grose C,Tyler S,Peters G,etal.Complete DNA sequence analyses of the first two varicella-zoster virus glycoprotein E (D150N) mutant viruses found in North America:evolution of genotypes with an accelerated cell spread phenotype[J].J Virol,2004,78(13):6799-6807.
[7] Loparev VN,Rubtcova EN,Bostik V,etal.Identification of five major and two minor genotypes of varicella-zoster virus strains:a practical two-amplicon approach used to genotype clinical isolates in Australia and New Zealand[J].J Virol,2007,81(23):12758-12765.
[8] Wood MJ.History of varicella zoster virus[J].Herpes,2000,7(3):60-65.
[9] Tyler SD,Peters GA,Grose C,etal.Genomic cartography of varicella-zoster virus:a complete genome-based analysis of strain variability with implications for attenuation and phenotypic differences[J].Virology,2007,359(2):447-458.
[10] Kaushik KS,Lahiri KK,Kapila K,etal.Differentiation of wild-type varicella-zoster strains from India and the Oka vaccine strain using a VZV open reading frame-62 based PCR-RFLP technique[J].Braz J Infect Dis,2008,12(4):313-315.
[11] Loparev VN,Gonzalez A,Deleon-Carnes M,etal.Global identification of three major genotypes of varicella-zoster virus:longitudinal clustering and strategies for genotyping[J].J Virol,2004,78(15):8349-8358.
[12] Takada M,Suzutani T,Yoshida I,etal.Identification of varicella-zoster virus strains by PCR analysis of three repeat elements and a PstI-site-less region[J].J Clin Microbiol,1995,33(3):658-660.
[13] 甘霖,王明丽,赵俊,等.水痘-带状疱疹病毒临床分离株基因特征分析[J].中华流行病学杂志,2010,31(2):189-193.
[14] Tipples GA,Safronetz D,Gray M.A real-time PCR assay for the detection of varicella-zoster virus DNA and differentiation of vaccine,wild-type and control strains[J].J Virol Methods,2003,113(2):113-116.
[15] Breuer J,Grose C,Norberg P,etal.A proposal for a common nomenclature for viral clades that form the species varicella-zoster virus:summary of VZV Nomenclature Meeting 2008,Barts and the London School of Medicine and Dentistry,24-25 July 2008[J].J Gen Virol,2010,91(Pt 4):821-828.
[16] Faga B,Maury W,Bruckner DA,etal.Identification and mapping of single nucleotide polymorphisms in the varicella-zoster virus genome[J].Virology,2001,280(1):1-6.
[17] Wagenaar TR,Chow VT,Buranathai C,etal.The out of Africa model of varicella-zoster virus evolution:single nucleotide polymorphisms and private alleles distinguish Asian clades from European/North American clades[J].Vaccine,2003,21(11/12):1072-1081.
[18] Barrett-Muir W,Scott FT,Aaby P,etal.Genetic variation of varicella-zoster virus:evidence for geographical separation of strains[J].J Med Virol,2003,70 Suppl 1:S42-S47.
[19] Sergeev N,Rubtcova E,Chizikov V,etal.New mosaic subgenotype of varicella-zoster virus in the USA:VZV detection and genotyping by oligonucleotide-microarray[J].J Virol Methods,2006,136(1/2):8-16.
[20] Schmidt-Chanasit J,Stürmer M,Hahn A,etal.Novel approach for genotyping varicella-zoster virus strains from Germany[J].J Clin Microbiol,2007,45(11):3540-3545.
[21] Sengupta N,Taha Y,Scott FT,etal.Varicella-zoster-virus genotypes in East London:a prospective study in patients with herpes zoster[J].J Infect Dis,2007,196(7):1014-1020.
