云南松基因组微卫星富集文库的构建
2014-03-17许玉兰,张瑞丽,田斌等
云南松基因组微卫星富集文库的构建
许玉兰1,2张瑞丽2田斌2蔡年辉2康向阳1段安安2
(1.北京林业大学 林木育种国家工程实验室,北京 100083;2.西南林业大学 国家林业局西南地区生物多样性保育重点实验室,昆明 650224)
采用磁珠富集法构建云南松微卫星富集文库。云南松基因组DNA经RsaⅠ酶切,与特定接头连接,再用接头特异引物进行PCR扩增。连接扩增产物与用生物素标记的(AG)12、(AT)12、(CG)12、(GT)12、(ACG)12、(ACT)12和(CCA)8探针杂交,通过链霉亲和素偶联的磁珠捕捉含接头和微卫星序列的片段并扩增,将获得的片段连接到pMD-19T载体上,转化至大肠杆菌JM109感受态细胞中,成功构建了云南松微卫星富集文库。通过PCR检测从文库中筛选阳性克隆,在383富集阳性菌落中获得阳性克隆257个,经测序分析,在获得的159条序列中,有143条含有SSR,其中完美型占65.73%,非完美型占23.78%,混合型占10.49%。结果表明,磁珠富集法构建云南松基因组微卫星文库高效、可行的,文库的构建为微卫星位点的分离、遗传多样性的分析等奠定基础。
云南松 富集文库 微卫星
云南松(Pinus yunnanensis)自然分布于东经96-108°、北纬23-30°之间,是分布区域内瘠薄荒山造林的先锋树种和治理水土流失的重要树种,占云南省林地面积的52%、占有林地蓄积的32%,
在云南林业经济生产和生态建设中占有举足轻重的作用[1,2]。然而,目前云南松的退化现象比较明显,急需对现有的资源进行评价,以制定切实可行的遗传保护策略。遗传变异的研究在开展植物育种及保护策略中的作用已引起广泛的重视[3],分子标记是分析遗传变异的有效途径[4,5],其中微卫星标记具有高度的多态性而成为常用的分子标记。这种多态性早在1989年Weber和May[6]就提出检测的方法,即采用成对的特异引物(由少数核苷酸组成,通过与微卫星侧翼序列来设计)对基因组总DNA扩增,凝胶电泳检测,从而实现对不同个体扩增产物多态性的检测。扩增引物的分离需要克隆和测序获得[7]。目前,微卫星分子标记在云南松的研究中应用较少。鉴于此,本研究利用磁珠富集法[8]构建云南松微卫星DNA富集文库,为进一步筛选云南松微卫星遗传标记奠定基础,以期利用微卫星标记进行云南松群体遗传结构分析,为云南松种质资源的评价、核心群体的构建以及保护策略的制定提供遗传学信息。
1 材料与方法
1.1 材料
1.1.1 材料来源 云南松样品材料取自2年生云南松幼嫩新鲜针叶。
1.1.2 主要试剂 所需基因组提取试剂盒购于Tiangen,DNA限制性内切酶RsaⅠ、XmnⅠ和连接酶T4购于New England Biolabs(NEB),TaqDNA聚合酶购于TaKaRa,Dynabeads链霉亲和素磁珠M-280试剂盒(Dynalbeads M-280 Streptavidin,112-05D)、磁珠收集器(Dynal MPC,123-20D)和琼脂糖购于Invitrogen,克隆连接载体pGEMR-T Easy购于Promega,转化用的大肠杆菌JM109感受态细胞及其它生化试剂购于TaKaRa,所需接头、引物及探针均由生工生物工程(上海)有限公司合成。
1.2 方法
1.2.1 基因组DNA的提取 利用基因组提取试剂盒(Tiangen),按照试剂盒说明书对云南松幼嫩新鲜针叶进行基因组DNA提取。
1.2.2 接头制备 采用SuperSNX接头,序列为:SuperSNX-F:5'-GTTTAAGGCCTAGCTAGCAGAATC -3';SuperSNX-R:5'-pGATTCTGCTAGCTAGGCCTTAAACAAAA-3'。
将SuperSNX-F(100 μmol/L)和SuperSNX-R(100 μmol/L)各1.4 μL,再加入NaCl(0.5 mol/L)1.4 μL以及ddH2O 2.8 μL,将该混合物7 μL快速混匀,在PCR仪上95℃,5 min,关机缓慢冷却至室温,即制成双链接头。
1.2.3 基因组DNA的酶切及与接头的连接 用RsaⅠ限制性内切酶消化基因组DNA,同时加入XmnⅠ酶防止接头发生自连,1.5%琼脂糖检测。
酶切片段在T4连接酶的作用下加上双链接头,混匀,16℃过夜。以SuperSNX-F为引物进行PCR扩增检测连接反应,反应体系为25 μL,包括 10×PCR Buffer 2.50 μL,100×BSA 2.50 μL,10 mmol/L dNTPs 1.50 μL,25 mmol/L MgCl22.00 μL,100 μmol/L SuperSNX-F 1.30 μL,5 U/μLTaq酶0.20 μL,连接产物模板2.00 μL,ddH2O补足。反应程序为:72℃ 2 min,95℃ 2 min;95℃ 20 s,60℃ 20 s,72℃ 90 s,25个循环;15℃ 10 min。扩增产物用1.0%琼脂糖检测。
1.2.4 磁珠富集法分离微卫星 用于磁珠亲和捕捉的微卫星探针为:(AG)12、(AT)12、(CG)12、(GT)12、(ACG)12、(ACT)12和(CCA)8, 分 别在其3'端标记上生物素,用这些微卫星探针与连接产物进行杂交,按Dynabeads链霉亲和素磁珠M-280试剂盒操作指南,捕捉含微卫星的DNA片段,洗脱掉其它非杂交片段。对富集杂交产物PCR扩增,反应体系为25 μL,包括10×PCR Buffer 2.50 μL,100×BSA0.62 μL,10 mmol/L dNTPs 1.38 μL,25 mmol/L MgCl22.00 μL,100 μmol/L SuperSNX-F 1.30 μL,5 U/μLTaq酶0.20 μL,连接产物模板4.00 μL,ddH2O补足。反应程序为:95℃ 2 min;95℃20 s,60℃ 20 s,72℃ 90 s,30个循环;72℃ 30 min,15℃ 10 min。扩增产物用1.0%琼脂糖检测。
1.2.5 富集文库构建与鉴定 将富集获得的富含微卫星DNA片段与pGEMR-T Easy载体相连接,再转化到感受态细胞JM109建立微卫星富集文库。涂布于含有IPTG、X-Gal和氨苄青霉素的LB平板上,37℃上培养10-16 h。富集文库中的阳性克隆采用PCR筛选,以M13+/M13-为引物,LB平板上挑选的
阳性菌落为模板,反应体系为20 μL,包括10×PCR Buffer 2.50 μL,10 mmol/L dNTPs 0.50 μL,25 mmol/L MgCl22.00 μL,10 pmol/L M13+/M13-各1.00 μL,5 U/μLTaq酶0.20 μL,模板2.00 μL,ddH2O补足。反应程序为:95℃ 3 min;95℃ 30 s,60℃ 30 s,72℃ 30 s,35个循环;72℃ 8 min,15℃ 10 min。扩增产物用1.0%琼脂糖检测[9]。
对PCR检测到的阳性克隆进行测序,对测序结果进行分析以鉴定文库中微卫星DNA 的富集效率。对序列去除载体后用SSRHunter进行微卫星的查找[10]。
2 结果
2.1 基因组DNA提取
提取的基因组DNA如图1所示,琼脂琼脂糖检测显示,基因组比较完整,无明显降解,浓度和纯度均较高,可用于后续试验。
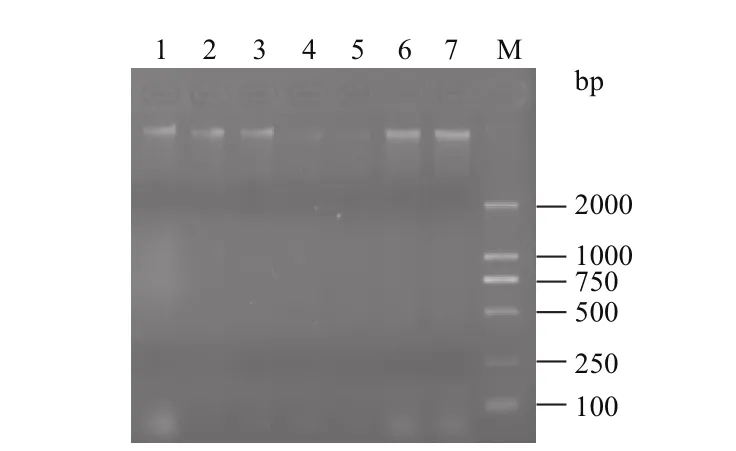
图1 云南松基因组DNA
2.2 基因组酶切
用RsaⅠ限制性内切酶消化基因组DNA,酶切产物用1.5%琼脂糖检测,结果如图2。从图2可知,大部分酶切片段集中在300-1 000 bp之间,说明DNA已被酶切,满足构建云南松基因组微卫星富集文库的需要。

图2 云南松基因组RsaⅠ酶切
2.3 酶切片段与接头的PCR检测
酶切片段在聚合酶的作用下与人工双链接头连接,对连接的产物进行PCR扩增,1.0%琼脂糖检测,结果如图3。片段在500-1 000 bp均匀弥散分布,DNA酶切片段和接头连接成功,得到的产物可用于构建云南松基因组微卫星富集文库。
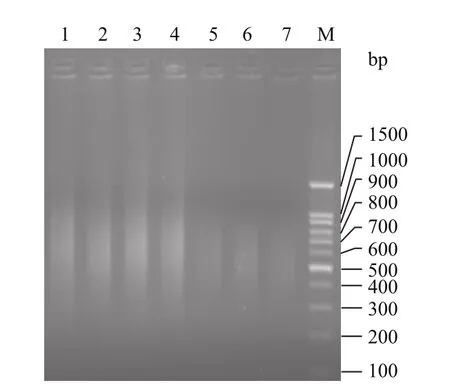
图3 接头连接产物PCR扩增
2.4 杂交富集产物检测
杂交、富集后的产物为模板获得的PCR扩增产物,经1.0%琼脂糖检测(图4),片段主要分布于300-1 000 bp之间,富集效果较好。
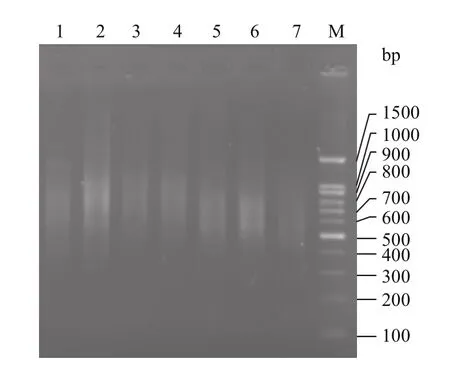
图4 云南松微卫星富集片段PCR扩增
2.5 微卫星富集文库构建及鉴定
以富集反应得到的DNA产物为模板进行PCR扩增,将PCR扩增产物与载体连接后转入大肠杆菌JM109感受态细胞,获得微卫星富集文库。从微卫星富集文库中随机挑取白色阳性菌落,PCR扩增检测菌落的插入片段,筛选可能含有微卫星片段的阳
性克隆,扩增产物经1.0%琼脂糖检测,结果如图5 所示。

图5 云南松微卫星富集文库阳性克隆PCR鉴定
有一条扩增带的即为插入片段全长带,该克隆内可能含有微卫星插入片段。从图5可以看出,从左往右,泳道1、4、6、7、8、9、10、11、12、13、14、15、16、17、19、20、21、22、24中有扩增条带,均有可能含有微卫星插入片段。在383个阳性菌落中,257个菌落获得PCR扩增条带,插入片段在400-1 000 bp之间,即PCR检测阳性率为67.10%。
2.6 克隆测序
对含PCR扩增条带的257个重组克隆进行DNA测序,在159条已获得的序列中,有143条含有重复次数不少于3次的微卫星序列,PCR检测筛选的微卫星序列效率为55.64%(143/257),富集效率为37.34%(143/383)。获得的序列去除载体序列后,采用SSRHunter软件进行SSR的搜索。搜索后的序列,按Weber的分类标准把微卫星序列划分为完美型(Perfect)、非完美型(Imperfect)和复合型(Compound)3类;将测序得到的143条微卫星序列进行分类,其中完美型94条(65.73%)、非完美型34条(23.78%)和复合型15条(10.49%),部分序列的重复序列和侧翼序列结果表1。
3 讨论
微卫星标记可以通过基因组分离、数据库序列信息以及近缘种的转移等方面获得。传统微卫星的分离是先构建基因组文库,然后选择合适的探针对基因组文库进行扫描,这一过程需要大量的人力和时间。为避免传统分离微卫星方法中进行基因组文库的构建和扫描,研究者相继提出一些改进的方法,如起初的在随机扩增多态性DNA方法[11]的基础上改进以扩增未知的微卫星,通过重复锚定随机引物[12]或RAPD引物扩增,随后用微卫星探针对扩增条带进行Southern杂交[13,14],此外还有阳性选择性克隆[15]、RAPD扩增产物的克隆和阵列克隆等[16],这些方法很快就取代传统耗时耗力建库和扫描的方法[17],与此同时,也有采用引物延伸来构建微卫星富集文库,较早采用这种方法分离微卫星的是AC富集文库,阳性克隆得率40%-50%[18],甚至高达100%[19],但步骤繁琐,没有得到很好的应用[17]。与这些方法相比,磁珠富集法是先杂交富集再建库,省去了建立较大的基因组文库,并在此基础上进行大量筛选工作,具有周期短、操作简便、目的性强、阳性克隆率高、一次可获得大量的微卫星序列等优点[20]。经阳性菌落的挑选、扩增、测序分析,PCR检测筛选的微卫星序列效率为55.64%(143/257),富集效率为37.34%(143/383),相比较而言,传统方法在植物中的阳性克隆比例仅为0.059%-5.8%,平均2.3%[17]。此外,本研究PCR检测准确率高达89.94%(143/159),与采用含有SSR的序列为引物的方法PIMA(PCR-based isolation of microsatellite arrays)相差无几。邓欣等[9]研究报道,从104个克隆测序分析中得到97个含有微卫星序列,筛选的微卫星序列准确率达到93.27%。说明利用磁珠富集
法进行云南松微卫星文库的构建是高效、可行的。经测序分析,在143条含有SSR的序列中,完美型占65.73%,非完美型占23.78%,混合型占10.49%。Winter等[21]研究认为,在真核生物基因组中,一般完美型比例要显著高于非完美型,本研究与该规律相符。
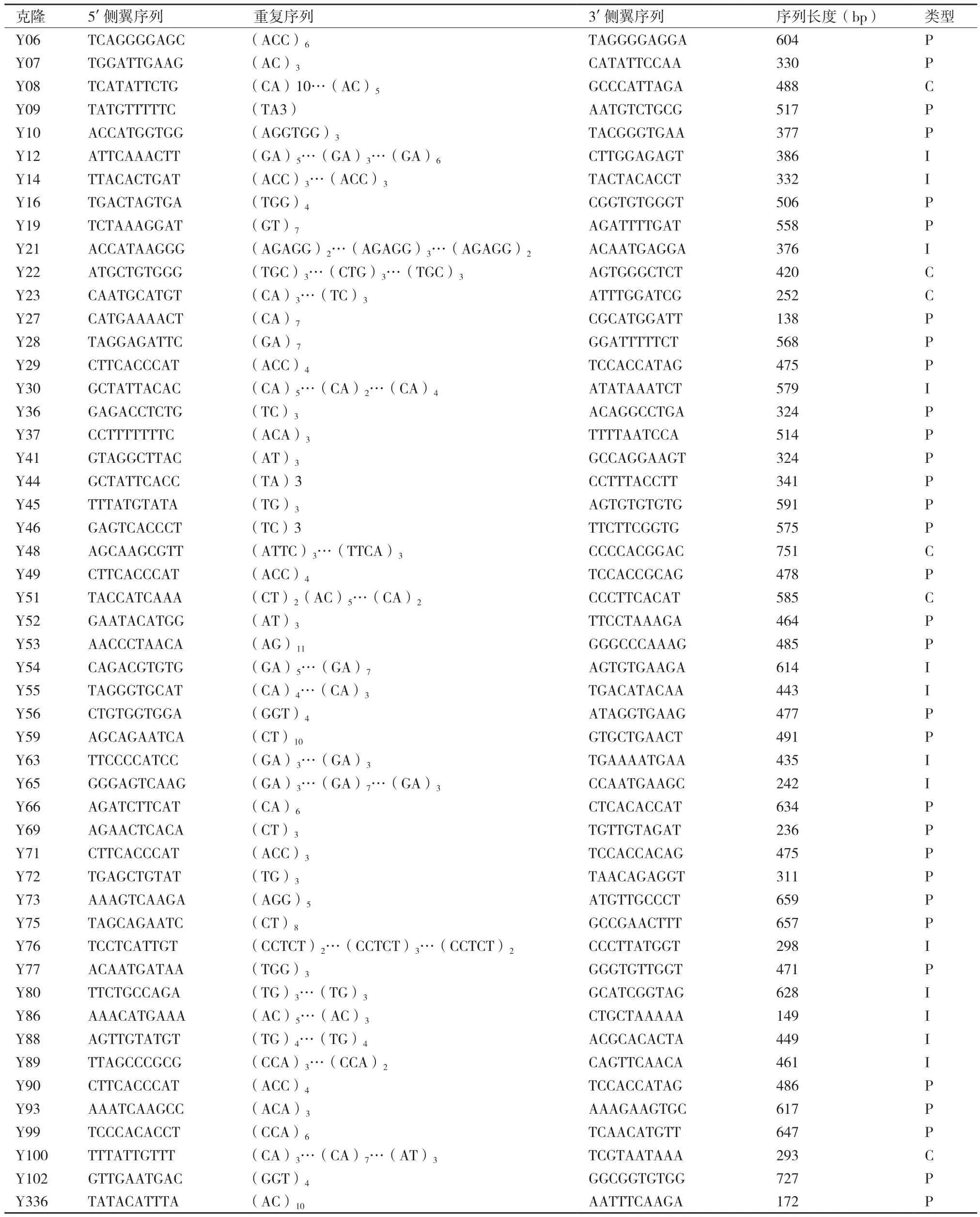
表1 云南松部分微卫星重复序列及其侧翼序列
磁珠富集法涉及到高质量基因组DNA的获得、酶切消化、连头与酶切片段DNA的连接、连接产物的富集、载体连接克隆等步骤,各个过程是连续且相互依存的,较关键的一步是磁珠富集的效率和特异性[9],不含SSR的DNA也被吸附到磁珠表面形成非专一性吸附[22],直接影响微卫星序列获得的效率。对不同的探针长度、杂交条件和洗脱条件报道较多,然而利用这些条件对微卫星富集效率的影响开展调查分析较少[17]。起初Kandpal等[23]对洗脱时的温度提出一定的标准,以尽可能地洗掉非专一性吸附的多余片段,仅保留富含微卫星序列的目的片段。Gao等[20]研究报道将杂交温度和首次洗涤温度提高到60℃可基本消除非专一性吸附的现象。邓欣等[9]在花生的微卫星分离中,将杂交温度和首次洗涤温度提高到68℃,取得了较满意的效果。本研究中将该温度提高到66℃,也获得比较好的结果。因此,不同物种SSR富集文库构建时,其富集的条件可能不一样,富集过程杂交和洗涤的温度需要进行比较和优化。
4 结论
采用磁珠富集法进行云南松基因组微卫星文库的富集,在随机挑选的383个阳性克隆中,有257个菌落含有扩增条带;在获得的159条序列中,有143条含有SSR,其中完美型占65.73%,非完美型占23.78%,混合型占10.49%。磁珠富集法构建云南松基因组微卫星文库高效、可行。
[1] 中国科学院中国植物志编辑委员会.中国植物志[M]. 第7卷.北京:科学出版社, 1978:211-282.
[2] 金振洲, 彭鉴.云南松[M].昆明:云南科技出版社, 2004:6, 21, 332.
[3] Sehgal D, Raina SN. DNA markers and germplasm resource diagnostics:new perspectives in crop improvement and conservation strategies[M]//Arya ID, Arya S. Utilization of biotechnology in plant sciences. Meerut, India:Arya Rastogi Press, 2009:39-54.
[4] Varshney RK, Graner A, Sorrells ME. Genic microsatellite markers in plants:features and applications[J]. Trends Biotechnol, 2005, 23(1):48-55.
[5] Kalia RK, Rai MK, Kalia S, et al. Microsatellite markers:an overview of the recent progress in plants[J]. Euphytica, 2011, 177(3):309-334.
[6] Weber JL, May PE. Abundant class of human DNA polymorphisms which can be typed using the polymerase chain reaction[J]. The American Journal of Human Genetics, 1989, 44(3):388-396.
[7] Powell W, Machray GC, Provan J. Polymorphism revealed by simple sequence repeats[J]. Trends in Plant Science, 1996, 1(7):215-222.
[8] Glenn TC, Schable NA. Isolating microsatellite DNA loci[J]. Methods in Enzymology, 2005, 395:202-222.
[9] 邓欣, 陈信波, 龙松华, 等.用磁珠富集法分离亚麻基因组微卫星分子标记[J].作物学报, 2008, 34(12):2099-2105.
[10] Weber JL. Informativeness of human(dC-dA)n·(dG-dT)npolymorphisms[J]. Genomics, 1990, 7(4):524-530.
[11] Williams JG, Kubelik AR, Livak KJ, et al. DNA polymorphisms amplified by arbitrary primers are useful as genetic markers[J]. Nucleic Acids Research, 1990, 18(22):6531-6535.
[12] Wu K, Jones R, Danneberger L, Scolnik PA. Detection of microsatellite polymorphisms without cloning[J]. Nucleic Acids Research, 1994, 22:3257-3258.
[13] Cifarelli RA, Gallitelli M, Cellini F. Random amplified hybridization microsatellites(RAHM):isolation of a new class of microsatellite-containing DNA clones[J]. Nucleic Acids Research, 1995, 23(18):3802-3803.
[14] Richardson T, Cato S, Ramser J, Ket al. Hybridization of microsatellites to RAPD:a new source of polymorphic markers[J]. Nucleic Acids Research, 1995, 23(18), 3798-3799.
[15] Ender A, Schwenk K, Stadler T, et al. RAPD identification of microsatellites in Daphnia[J]. Molecular Ecology, 1996, 5(3):437-441.
[16] Lunt DH, Hutchinson WF, Carvalho GR. An efficient method for PCR-based isolation of microsatellite arrays(PIMA)[J]. Molecular Ecology, 1999, 8:891-894.
[17] Zane L, Bargelloni L, Patarnello T. Strategies for microsatellite isolation:a review[J]. Mol Ecol, 2002, 11:1-16.
[18] Ostrander EA, Jong PM, Rine J, Duyk G. Construction of smallinsert genomic DNA libraries highly enriched for microsatellite repeat sequences[J]. Proceedings of the National Academy of Sciences of the USA, 1992, 89(8):3419-3423.
[19] Paetkau D. Microsatellites obtained using strand extension:An enrichment protocol[J]. Biotechniques, 1999, 26(4), 690-692, 694-697.
[20] Gao GQ, He GH, Li YR. Microsatellite enrichment from AFLP fragments by magnetic beads[J]. Acta Botanica Sinica, 2003, 45(11):1266-1269.
[21] Winter AK, Fredholm M, Thomsen PD. Variable(dG-dT)n.(dC-dA) n sequences in the porcine genome[J]. Genomics, 1992, 12(2):281-288.
[22] Refseth UH, Fangan BM, Jakobsen KS. Hybridization capture of microsatellite directly from genomic DNA[J]. Electrophoresis, 1997, 18:1519-1523.
[23] Kandpal RP, Kandpal G, Weissman SM. Construction of libraries enriched for sequence repeats and jumping clones, and hybridization selection for region-specific markers[J]. Proceedings of the National Academy of Sciences of the USA, 1994, 91(1):88-92.
(责任编辑 李楠)
Construction of Microsatellite-enriched Library of Pinus yunnanensis
Xu Yulan1,2Zhang Ruili2Tian Bin2Cai Nianhui2Kang Xiangyang1Duan Anan2
(1. College of Biological Science and Technology of Beijing Forestry University,National Engineering Laboratory for Tree Breeding,Beijing Forestry University,Beijing 100083;2. Key Laboratory of Biodiversity Conservation in Southwest China,State Forestry Administration,Southwest Forestry University,Kunming 650204)
Microsatellite-enriched libraries in Pinus yunnanensis were constructed using an enrichment method with biotin-labeled probes and streptavidin-coated magnetic beads.Genomic DNAs of P. yunnanensis were extracted and digested with restriction enzyme Rsa 1. Adapters were ligated to the end of restriction fragments. Ligated products were amplified by PCR with primers complementary to adapters. The PCR products were hybridized with biotin-labeled simple sequence repeat probes(AG)12, (AT)12, (CG)12, (GT)12, (ACG)12, (ACT)12and(CCA)8. The hybridized complex was caught by magnetic beads coated with streptavidin. The biotinylated hybrids were isolated on the magnetic particles according to the strong affinity between biotin and streptavidin. The selected DNAs were amplified by PCR with primers complementary to adapters and cloned into pMD-19T plasmid vector, and then transformed into E. coli JM109 hosts. SSR enriched genomic DNA library of P. yunnanensis was constructed. A total of 257 positive clones were selected by PCR from 383 transformants in the microsatellite-enriched library, and 143 clones were found to contain simple sequence repeats(SSR)among 159 sequences. Among them, 94(65.73%)were perfect repeat, 34 imperfect(23.78%)and 15(10.49%)compound repeat. The results indicated that the method was efficient to construct the microsatelliteenriched library of P. yunnanensis. The library will be useful for further research on the isolation of microsatellite markers and the analysis of genetic diversity. .
Pinus yunnanensis Enriched library Microsatellite
2013-07-17
国家自然科学基金项目(31260191),云南省自然科学基金项目(2010CD065),云南省省院省校教育合作咨询共建重点学科(211015),云南省教育厅研究生项目(2012J052)
许玉兰,女,副教授,博士研究生,研究方向:林木遗传育种的研究;E-mail:xvyulan@163.com
康向阳,男,博士,博士生导师,教授,研究方向:林木遗传育种的教学与研究;E-mail:kangxy@bjfu.edu.cn
段安安,男,博士,博士生导师,教授,研究方向:林木遗传育种的教学与研究;E-mail:duananan@gmail.com
