克罗诺杆菌MALDI-TOF-MS数据库的建立及应用
2014-03-09赵贵明杨海荣赵勇胜
赵贵明,刘 洋,陈 颖,杨海荣,赵勇胜,王 娉
克罗诺杆菌MALDI-TOF-MS数据库的建立及应用
赵贵明,刘 洋,陈 颖*,杨海荣,赵勇胜,王 娉
(中国检验检疫科学研究院,北京 100123)
应用基质辅助激光解吸电离飞行时间质谱(matrix-assisted laser desorption ionisation time-of-flight mass spectrometry,MALDI-TOF-MS)技术建立了克罗诺杆菌MALDI-TOF-MS数据库,用于该菌属内种和亚种的高通量鉴定及菌株分型。在优化培养条件、样品处理方法及确定MALDI-TOF-MS蛋白质指纹图谱采集参数的基础上,将采集的8 种参考菌株蛋白质质谱数据通过Biotyper软件构建克罗诺杆菌的MALDI-TOF-MS数据库,再用相近肠杆菌及该属分离株验证数据库的准确性。结果表明:应用建立的克罗诺杆菌的MALDI-TOF-MS数据库对135 株克罗诺杆菌分离株进行分析,鉴定分值均不小于2.0,达到了种水平鉴定要求,通过聚类分析,可进一步分型。构建的MALDI-TOF-MS数据库为克罗诺杆菌高通量鉴定与分型提供了一种新的手段。
克罗诺杆菌;MALDI-TOF-MS;数据库;鉴定;分型
克罗诺杆菌(Cronobacter spp.)原名为阪崎肠杆菌(Enterobacter sakazakii)[1],是一种革兰氏阴性食源性条件致病菌[2],广泛存在于人和动物肠道、土壤、水及日常食品中[3],极易污染婴幼儿乳粉,引起早产儿、新生儿脑膜炎、致死性小肠结肠炎、败血症等疾病[4-5]。鉴于该种菌表现出的生物多样性,2008年Iversen等[6]根据最新实验结果建议将阪崎肠杆菌重新划分为一个新属,即克罗诺杆菌属(Cronobacter spp.),属下包括1 个基因种(C. genomospecies)、5 个新种,分别为阪崎克罗诺杆 菌(C. sakazakii)、丙二酸盐阳性克罗诺杆菌(C. malonaticus)、苏黎世克罗诺杆菌(C. turicensis)、穆汀斯克罗诺杆菌 (C. muytjensii)、都柏林克罗诺杆菌(C. dubli nensis),其中都柏林克罗诺杆菌包括3 个亚种,分别为都柏林克罗诺杆菌都柏林亚种(C. dublinensis subsp. dubl i nensi s)、都柏林克罗诺杆菌乳粉亚种(C. dublinensis subsp. lactaridi)和都柏林克罗诺杆菌洛桑亚种(C. dublinensis subsp. lausannensis)。目前鉴定克罗诺杆菌的方法包括生化反应方法[7]、分子生物学方法(普通聚合酶链式反应(polymerase chain reaction,PCR)检测[8]、实时PCR检测[9]、环介导恒温扩增)等[10],但是传统的生化方法耗时长、操作繁琐;分子检测方法虽灵敏但获得结果至少需要4 h。基质辅助激光解吸电离飞行时间质谱(matrix-assisted laser desorption ionisation timeof-f l ight mass spectrometry,MALDI-TOF-MS)是近些年新发展起来的鉴定微生物的新方法[11],不仅用于不同领域中微生物的鉴定[12-14],还用于微生物的分析[15-16]。在微生物鉴定方面,它不同于传统生化方法需通过观察细菌生长过程中对糖醇类物质的利用情况得出鉴定结果,也无需像分子生物学方法那样先提取核酸,再进行扩增才能获得鉴定结果,可以将待鉴定细菌培养物直接点在靶板上,通过检测微生物的特征性生物标识物(主要集中在2~20 kD,受生长环境和状态影响很小的持续高表达蛋白),获得质谱数据,与已知微生物的标准蛋白质组指纹质谱数据库进行比较,一个样品从菌落到取得结果只需5 min,1.5 h可以分析约100 个样品,从而实现对微生物的快速、准确鉴定[17]。已商业化的用于细菌鉴定的MALDITOF-MS不仅操作方法简单、通量大、重现性高,而且配备了相应的细菌鉴定数据库[18],但是,由于克罗诺杆菌分类学发生变化,目前的数据库没有与重新分类的克罗诺杆菌属相匹配的分析数据库,本实验拟利用Biotyper软件应用MALDI-TOF-MS采集分析克罗诺杆菌属蛋白质质量图谱构建数据库,解决克罗诺杆菌的快速鉴定与分型问题。
1 材料与方法
1.1 材料与试剂
1.1.1 菌株
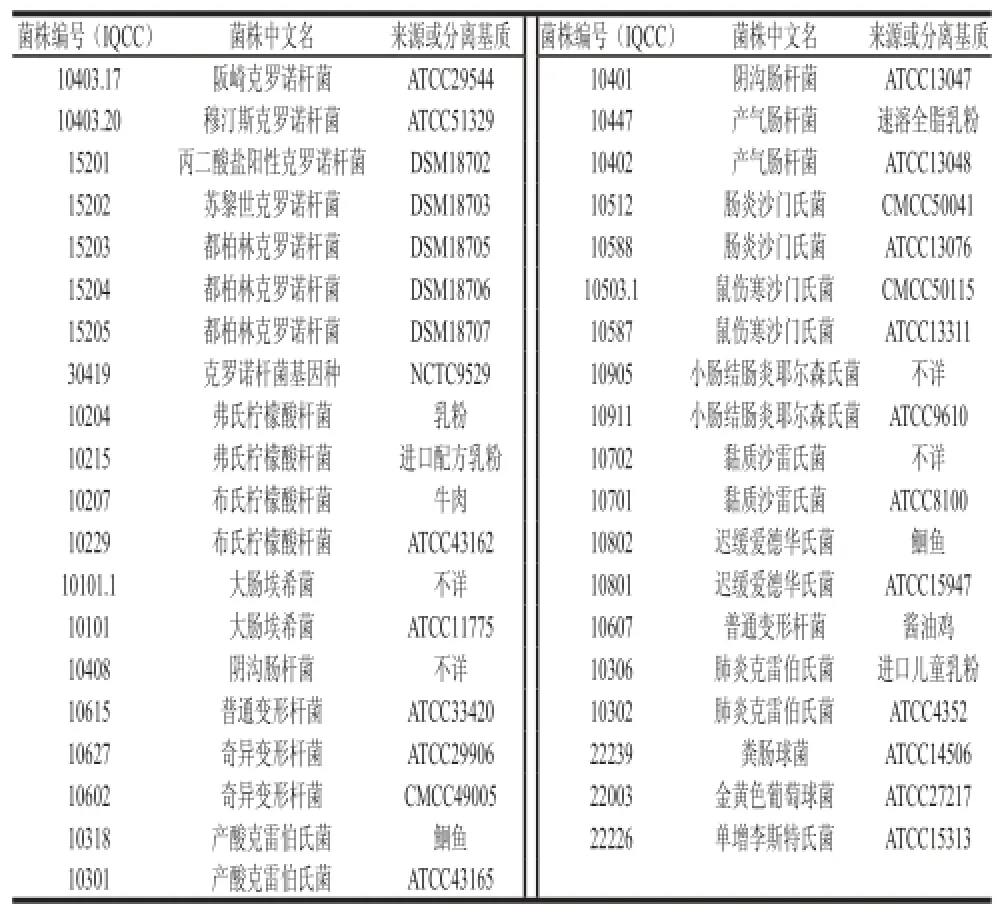
表1 参考菌株Table 1 Reference strains
共174株菌株,均来自中国检验检疫科学研究院微生物菌种保藏管理中心(Inspection & Quarantine Culture Collection,IQCC),参考菌株购自美国模式培养物集存库(American Type Culture Collection,ATCC)、德国微生物和细胞培养物集存库(Deutsche Sammlung von Mikroorganismen und Zellkulturen,DSMZ)和英国国家模式培养物集存库(National Collection of Type Cultures,NCTC),135 株克罗诺杆菌分离菌株均经过SN/T 1632—2013《出口奶粉中阪崎肠杆菌(克罗诺杆菌属)检验方法》第1部分:分离与计数方法、第3部分:荧光PCR方法验证,实验菌株信息见表1和表2。
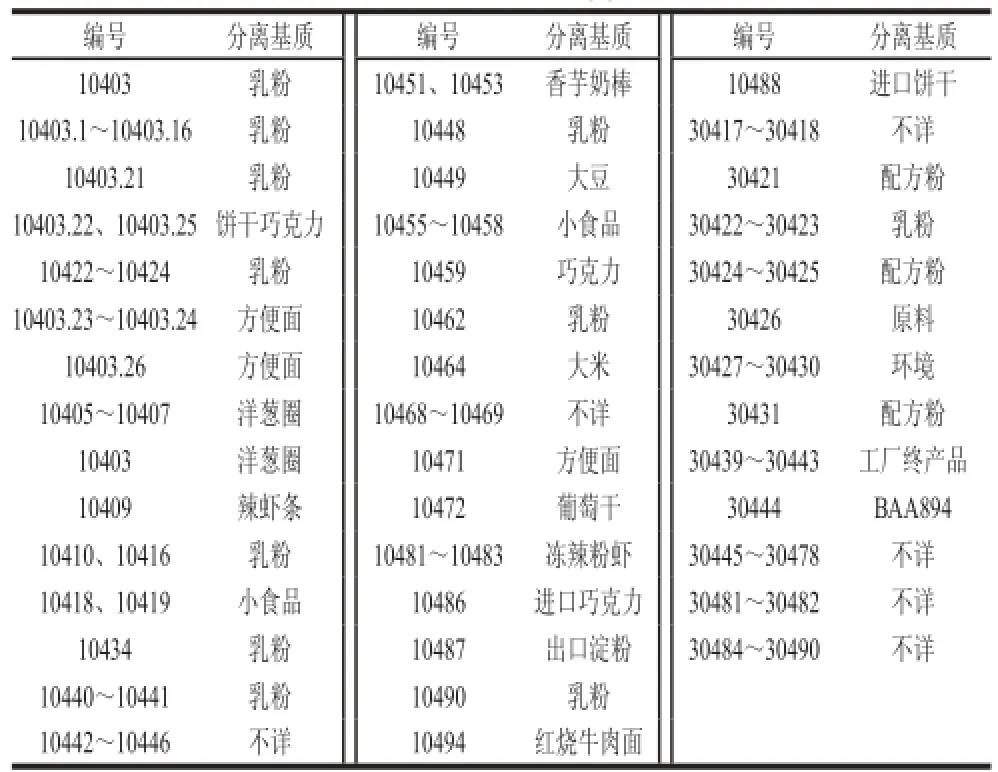
表2 克罗诺杆菌分离菌株Table 2 Cronobacter spp. isolates
1.1.2 试剂
甲酸(formicacid,FA)、乙腈(acetonitrile,ACN)和无水乙醇(色谱纯) 美国Fisher公司;三氟乙酸(trifluoracetic acid,TFA)(色谱纯) 德国Merck公司;标准品和基质α-氰基-4-羟基肉桂酸(α-cyano-4-hydroxy-cinnamic acid,HCCA) 德国Bruker公司;脑心浸液培养基(brain heart infusion medium,BHI)、胰蛋白胨大豆琼脂(tryptic soy agar,TSA)和营养琼脂(nutrient agar,NA) 美 国BD公司。
1.2 仪器与设备
AutoflexIII生物质谱仪 德国Bruker公司。
1.3 方法
1.3.1 培养条件的确定
将克罗诺杆菌的模式菌株阪崎克罗诺杆菌ATCC29544分别接种于TSA、NA两种培养基,36 ℃培养18、24、48 h。将不同培养条件下采集的ATCC29544质谱图与MALDI Biotyper数据库相比对,获得最高匹配度鉴定结果、谱图基线平滑、信噪比高、蛋白质峰多的条件为最佳培养条件。
1.3.2 样品处理方法的确定
直接涂抹法:用无菌棉棒直接挑取单菌落,涂于样品靶板上,室温条件下晾干,再覆盖1.0 μL基质溶液,晾干后进行质谱分析。
甲酸-乙腈提取法:挑取适量(约5~10 mg)菌落样品于1.5 mL离心管中,加入300 μL纯净水,混匀,再加入900 μL无水乙醇,混匀;12 000 r/min离心2 min,弃去上清液;加入50 μL 70%甲酸,仔细混匀,再加入50 μL乙腈,仔细混匀,12 000 r/min离心2 min,吸取1 μL上清液点在靶板上,自然晾干后再点1 μL基质覆盖,晾干后进行质谱分析。
1.3.3 培养基对质谱图背景信号的影响
选取两种培养基、不同培养时间下无微生物生长的区域,用无菌棉拭子接触培养基表面进行取样,后续处理方法相同,然后进行质谱分析。
1.3.4 MALDI-TOF-MS数据采集
仪器参数:Smartbeam激光器;波长355 nm;每个样品谱图累积400 个激光脉冲信号;质量范围m/z 2~20 kD;延迟提取时间200 ns;加速电压20 kV;提取电压18.6 kV;聚焦电压6.5 kV。每个样品采集30 张蛋白质量图谱,以获得可靠鉴定结果。
1.3.5 MALDI-TOF-MS数据库建立
选择最佳培养条件培养8 株参考菌株,每株参考菌株选3 个菌落作为平行试样,每个平行试样重复点样3 次,每个靶点单独采集8 张蛋白质量图谱,每个平行试样选取1 个靶点作为最终采集数据(尽量消除因样品制备可能带来的影响),每个参考菌株共收集24 张质谱图,每张质谱图都与Biotyper软件中的数据库比对,鉴定结果均为克罗诺杆菌,再通过Biotyper软件,将采集的质量图谱汇总生成克罗诺杆菌的标准质谱数据。
1.3.6 MALDI-TOF-MS数据库验证
将28 株肠杆菌科其他菌、1 株粪肠球菌、1 株金黄色葡萄球菌、1 株单增李斯特氏菌和135 株克罗诺杆菌属分离株,采用相同培养条件进行质谱分析,用Biotyper数据库和自建克罗诺杆菌数据库获得鉴定分值,根据分值验证克罗诺杆菌MALDI-TOF-MS数据库的准确性。
1.3.7 MALDI-TOF-MS结果判定
所采集的质谱图与Biotyper软件中标准图谱进行比对,鉴定分值≥2.0表示可鉴定到种水平,其中2.300~3.000之间表示菌种鉴定的可信度较高,在1.700~1.999之间表示可属鉴定到属,在0.000~1.699之间表示不可信的鉴定。
2 结果与分析
2.1 最佳培养条件的确定
微生物在不同的培养基中摄取不同的营养物质,因此生长特性也各异;培养时间的长短也会影响微生物的生长状态[19]。阪崎克罗诺杆菌ATCC29544在NA、TSA两种培养基上生长状态均良好,不同培养时间所获得的质谱图与数据库的匹配值均在2.3以上,但在TSA培养基上培养24 h时获取的蛋白指纹图谱与其他培养条件的相比,相对峰强度高、信噪比高、离子峰多,因此,确定最佳培养条件为在TSA培养基上培养24 h。见图1。
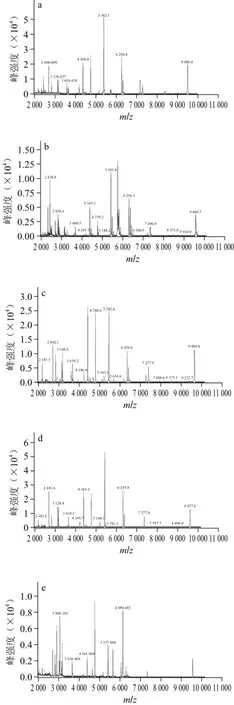
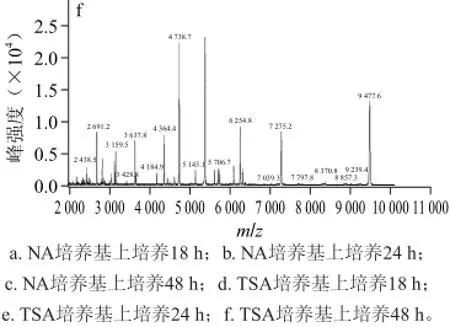
图1 不同培养条件对阪崎克罗诺杆菌(ATCC29544)蛋白指纹图谱的影响Fig.1 Effect of different culture conditions on protein mass spectrometric profiles of C. sakazakii
2.2 样品最适处理方式的确定
仍以阪崎克罗诺杆菌ATCC29544作为实验菌株,在TSA培养基上培养24 h之后分别用直接涂抹法和甲酸-乙腈法对样品进行处理。由图2可知,两种方法所得图谱中蛋白离子峰的强度一致,但甲酸-乙腈法所得图谱的离子峰信噪比较高,系统可标识出更多的离子峰,包含了更多的蛋白标志物,这可能是因为甲酸-乙腈法中水和乙醇能洗掉培养基内的杂质[20],两种溶剂混合作用将菌体表面和细胞内的高峰强度的蛋白提取出来,最终选择甲酸-乙腈法为样品最适处理方式。见图2。
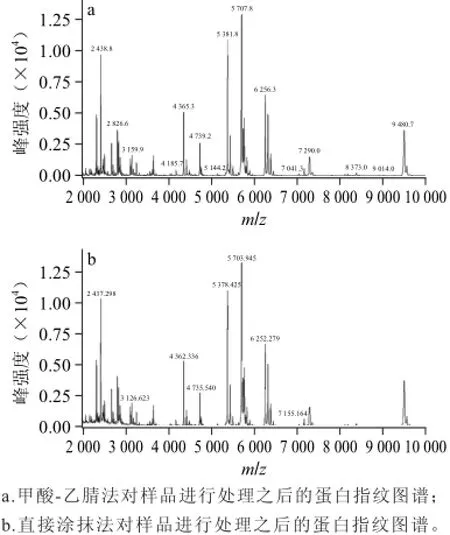
图2 不同处理方法对细菌蛋白指纹图谱的影响Fig.2 Effect of different sample treatments on protein mass spectrometric profiles
2.3 培养基对质谱图信号的影响
选取不同培养条件下未生长微生物的区域进行蛋白质量图谱的采集,单纯的培养基峰信号集中在m/z 2 000~4 000之间,峰强度在100~500之间,与微生物蛋白质峰相比,这些峰信号不在微生物的离子峰中,且相对峰强度低,因此培养基本身不影响MALDI-TOF-MS对微生物蛋白质量图谱的采集(图略)。
2.4 克罗诺杆菌模式菌株MALDI-TOF-MS鉴定数据库的建立
使用Biotyper软件将已采集的8 株克罗诺杆菌参考菌株的蛋白质量图谱汇总,建立克罗诺杆菌参考菌株的标准蛋白指纹图谱。每株克罗诺杆菌参考菌株共采集70个离子峰,以75%出现的离子峰为属特有离子峰,与数据库中阴沟肠杆菌和产气肠杆菌(生物学特性与克罗诺杆菌相似)的离子峰相比对,得到6个不同离子峰:m/z 3 366.3±0.6、3 413.8±1.2、3 537.6±1.7、4 385.9±0.9、4 623.7±3.0、4 697.9±0.7,作为判别克罗诺杆菌属的参考生物标识物。以37.5%出现的离子峰为种特有离子峰,得到23 个种特有的离子峰:m/z 3 064.9、3 156.2、3 243.25±2.0、3 275.7±0.5、3 299.5±1.5、3 352.7±0.5、3 463.4、3 564.3、3 675.7、3 795.3±0.9、4 613.9±0.6、5 553.1、5 799.2、5 855.1、6 535.8、6 548.6、7 022.1±0.7、7 036.2±0.4、7 233.6、8 081.2、9 225.0±0.7、9 234.3±1.2、9 249.8±0.8,可区分克罗诺杆菌属内不同种以及同一种内的不同亚种[21]。克罗诺杆菌参考菌株MALDITOF-MS数据转换出的蛋白质谱条码见图3。
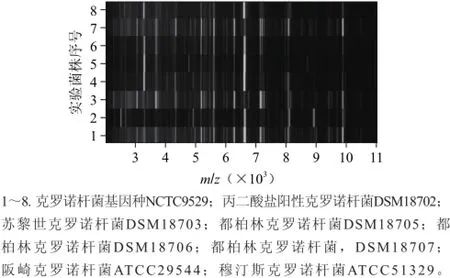
图3 8株克罗诺杆菌参考菌株MALDI-TOF蛋白质谱条码Fig.3 MALDI TOF protein mass spectral barcodes of 8 reference strains of Cronobancter spp.
2.5 MALDI-TOF-MS鉴定数据库的验证
采用所构建的克罗诺杆菌MALDI-TOF-MS鉴定数据库分析31 株肠杆菌科其他菌的蛋白质质量,所得分值均小于1.7,而与Biotper数据库相比较,鉴定分值均不小于2.0,鉴定结果与预期相同(表1)。该结果表明所建立的克罗诺杆菌MALDI-TOF-MS鉴定数据库能够用于克罗诺杆菌属和种水平的鉴定。
2.6 MALDI-TOF-MS鉴定数据库对克罗诺杆菌分离株的鉴定与分型
采用所建立的克罗诺杆菌数据库对135 株克罗诺杆菌分离株进行了鉴定。结果表明所有被测菌株均能鉴定到种的水平,其中阪崎克罗诺杆菌101 株、克罗诺杆菌基因种12 株、丙二酸盐阳性克罗诺杆菌9株、都柏林克罗诺杆菌7 株、苏黎世克罗诺杆菌4 株、穆汀斯克罗诺杆菌2 株。
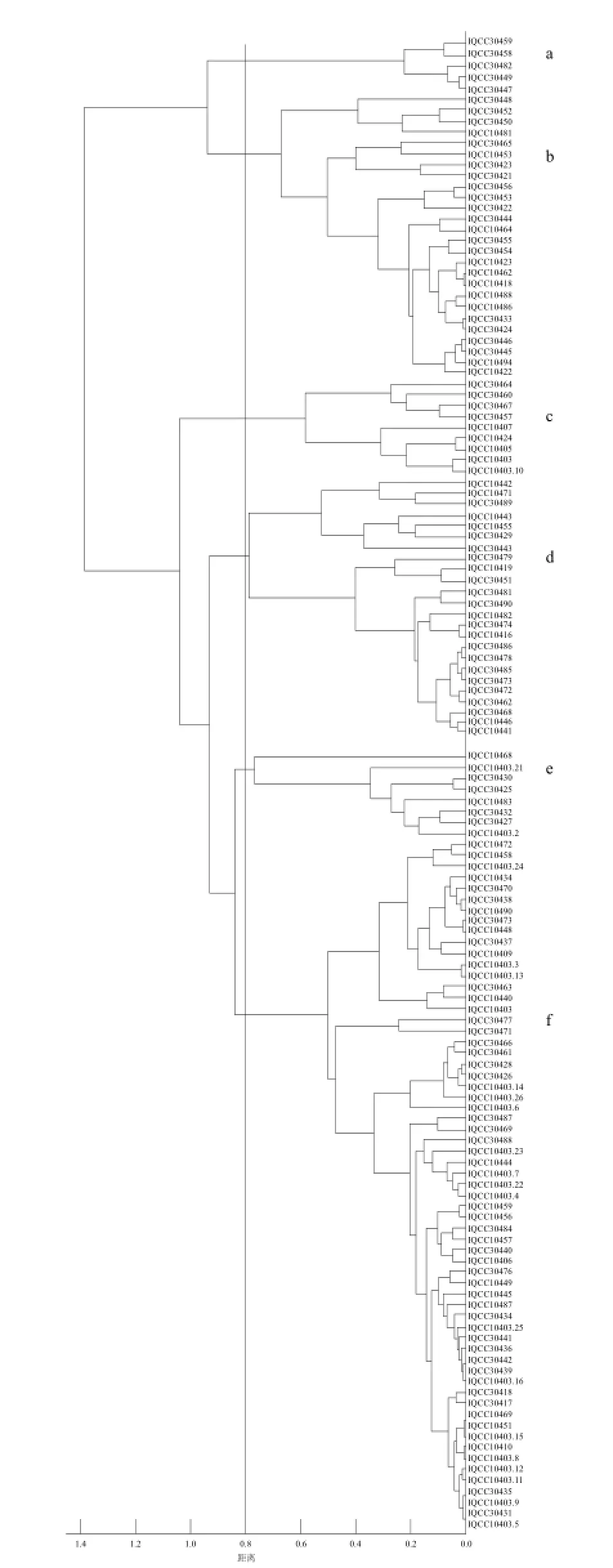
图4 135株克罗诺杆菌的聚类分析图Fig.4 Cluster analysis diagram for 135 strains of Cronobancter spp.
将135 株克罗诺杆菌分离菌株的蛋白质质量图谱进行聚类分析(图4),选择相似度0.8的差异水平时,可被分为6 类,用a~f类群表示,a类有5 株;b类有26 株,其中2 株来源于乳粉,1 株来源于香芋奶棒,1 株来源于冻熟辣粉整肢虾,1 株来源于出口红烧牛肉面;c类有9 株,其中3 株来源于洋葱圈,1 株来源于乳粉;d类有24 株,其中2 株来源于乳粉,1 株来源于洋葱圈,1 株来源于冻熟辣粉整肢虾;e类有8 株,其中4 株来源于乳粉,1 株来源于辣粉整肢虾,1 株来源于环境;f类有63 株,其中25 株来源于乳粉,4 株来源于工厂终产品,3 株来源于方便面。根据以上分析,样品菌株的分型与其对应的来源及种类并无明显对应关系,说明克罗诺杆菌在自然界的分布为随机分布。
3 讨 论
基质、培养条件、菌体处理方法被认为是影响MALDI-TOF-MS分析微生物的3 个主要因素。在培养条件优化方面,NA、TSA两种培养基对克罗诺杆菌属的蛋白质谱鉴定结果没有明显影响,这说明克罗诺杆菌对生长条件的要求较为宽泛,但从谱图中可以看出从两种培养基上生长的克罗诺杆菌获得的质谱图的特征峰强度和数量方面略有差异,因此,要根据待鉴定菌株的实际情况选择最佳的培养条件。在实验样品处理方面,直接涂抹法快速易操作,但易受菌体其他代谢物干扰;甲酸-乙腈提取法可以将细菌内部和表面的蛋白质提取出来,尽管本实验两种方法所得图谱中蛋白离子峰的强度一致,但甲酸-乙腈法所得图谱的离子峰的信噪比较高,包含了更多的蛋白标志物。
赵贵明等[22]曾用MALDI-TOF-MS对32 株克罗诺杆菌进行过与分型实验,鉴定到种、属水平分别为56.2%和37.5%,在实验中提出了利用Biotyper开放型数据库的特点建立克罗诺杆菌MALDI-TOF-MS数据库以提高克罗诺杆菌种鉴定水平的建议[23],本实验将135 株克罗诺杆菌全部鉴定到种水平的结果说明,通过自建数据库是提高鉴定结果准确性的方法之一。通过对鉴定菌株蛋白质质量图谱聚类分析,选择不同差异水平,可以在种以下对菌株进一步分型,这对克罗诺杆菌的快速溯源具有重要参考价值。
MALDI-TOF-MS除用于常规细菌的快速高通量鉴定外,还用于对较难鉴定菌株的快速识别,如对临床标本分离的544 株(79 个种)厌氧菌鉴定中,332 株(61%)可一次获得鉴定结果[24];对高毒性和耐药菌株的准确识别,如应用MALDI-TOF-MS分析了85 株经spa基因分型方法确定的耐甲氧西林金黄色葡萄球菌,结果60 株菌得出15 种质量图谱,经聚类分析后与分子分型方法相比不仅结果一致,而且区分更细[25]。因此,笔者认为在细菌鉴定工作中,如果应用MALDI-TOF-MS先行鉴定,再采用分子生物学技术如16S rRNA依据MALDI-TOF-MS给出的排序结果优先分析,将是现阶段提高细菌鉴定效率的理想组合。
[1] FARMER J J III, ASBURY M A, HICKMAN F W. The enterobacteriaceae study group (USA). Enterobacter sakazakii: a new species of “Enterobacteriaceae” isolated from clinical specimens[J]. International Journal of Systematic Bacteriology, 1980, 30(3): 569-584.
[2] HUNTER C J, PETROSYAN M, FORD H R, et al. Enterobacter sakazakii: an emerging pathogen in infants and neonates[J]. Surgical Infections, 2008, 9(5): 533-539.
[3] NAZAROWEC WHITE M, FARBER J M. Enterobacter sakazakii: a review[J]. International Journal Food Microbiol, 1997, 34(2): 103-113.
[4] YAN Q Q, CONDELL O, POWER K, et al. Cronobacter species (formerly known as Enterobacter sakazakii) in powdered infant formula: a review of our current understanding of the biology of this bacterium[J]. Journal of Applied Microbiology, 2012, 113(1): 1-15.
[5] BARBARA J, STOLL M D, HANSEN N, et al. Enterobacter sakazakii is a rare cause of neonatal septicemia or meningitis in vlbw infants[J]. The Journal of Pediatrics, 2004, 144(6): 821-823.
[6] IVERSEN C, MULLANE N, MCCARDELL B, et al. “Cronobacter gen. nov., a new genus to accommodate the biogroupsof Enterobacter sakazakii, and proposal of Cronobacter sakazakii gen.nov. comb. nov., C. malonaticus sp. nov., C. turicensis sp. nov., C. muytjensii sp. nov., C. dublinensis sp. nov., Cronobacter genomospecies 1, and of three subspecies, C. dublinensis sp. nov. subsp. dublinensis subsp. nov., C. dublinensis sp. nov. subsp. lausannensis subsp. nov., and C. dublinensis sp. nov. subsp. lactaridi subsp. nov. ”[J]. International Journal Systematic and Evolutionary Microbiol, 2008, 58(6): 1442-1447.
[7] 董晓晖, 吴清平, 莫树平, 等. 克罗诺杆菌检测方法研究进展[J]. 华中农业大学学报, 2013, 32(1): 130-136.
[8] CHEN Wanyi, AI Lianzhong, YANG Jielin, et al. Development of a PCR assay for rapid detection of Cronobacter spp. from food[J]. Canadian Journal of Microbiology, 2013, 59(10): 656-661.
[9] FRICKER FEER C, CERNELA N, BOLZAN S, et al. Evaluation of three commercially available real-time PCR based systems for detection of Cronobacter species[J]. International Journal of Food Microbiology, 2011, 146(2): 200-202.
[10] 胡连霞, 张伟, 张先舟, 等. 改良环介导等温扩增技术快速检测婴儿配方奶粉中的阪崎肠杆菌[J]. 微生物学报, 2009, 49(3): 378-382.
[11] 其布勒哈斯, 田世民, 邹明强, 等. MALDI-TOF质谱技术分析与鉴定病原细菌研究[J]. 微生物学通报, 2009, 36(3): 416-426.
[12] 龚艳清, 陈信忠, 杨俊萍, 等. 基质辅助激光解吸电离飞行时间质谱在李斯特菌检测和鉴定中的应用[J]. 食品科学, 2012, 33(6): 209-214.
[13] CARBONNELLE E, MESQUITA C, BILLE E, et al. MALDI-TOF mass spectrometry tools for bacterial identification in clinical microbiology laboratory[J]. Clinical Biochemistry, 2011, 44(1): 104-109.
[14] 赵贵明, 杨海荣, 赵勇胜, 等. MALDI-TOF MS在病原菌鉴定与分型中的应用进展[J]. 检验检疫学刊, 2012, 22(增刊1): 5-9.
[15] ESPINAL P, SEIFERT H, DIJKSHOORN L, et al. Rapid and accurate identification of genomic species from the Acinetobacter baumannii (Ab) group by MALDI-TOF MS[J]. Clinical Microbiology and Infection, 2012, 18(11): 1097-1103.
[16] MARINACH C, ALANIO A, PALOUS M, et al. MALDI-TOF MS-based drug susceptibility testing of pathogens: the example of Candida albicans and fluconazole[J]. Proteomics, 2009, 9(20): 4627-4631.
[17] WIESER A, SCHNEIDER L, JUNG J, et al. MALDI-TOF MS in microbiological diagnostics-identification of microorganisms and beyond (mini review) [J]. Applied Microbiology Biotechnology, 2012, 93(3): 965-974.
[18] BRIGHT J J, CLAYDON M A, SOUFIAN M, et al. Rapid typing of bacteria using matrix-assisted laser desorption ionisation time-of-flight mass spectrometry and pattern recognition software[J]. Journal of Microbiological Methods, 2002, 48(2/3): 127-138.
[19] 吕佳, 卢行安, 刘淑艳, 等. MALDI-TOF-MS技术鉴定食源 性致病菌的影响因素[J]. 分析仪器, 2011(2): 12-17.
[20] 陈秀金, 尹红红, 匡华, 等. 沙门氏菌 MALDI-TOF-MS 蛋白质指纹图谱分析方法的研究[J]. 食品与生物技术学报, 2012, 31(11): 1189-1198.
[21] STEPHAN R, ZIEGLER D, PFLÜGER V, et al. Rapid genus- and species-specific identification of Cronobacter spp. by matrix-assisted laser desorption ionizat ion-time of flight mass spectrometry[J]. Journal of Clinical Microbiology, 2010, 48(8): 2846-2851.
[22] 赵贵明, 杨海荣, 赵勇胜, 等. MALDI-TOF质谱技术对克罗诺杆菌的鉴定与分型[J]. 微生物学通报, 2010, 37(8): 1169-1175.
[23] 赵贵明, 杨海荣, 赵勇胜, 等. MALDI Biotyp er与API20E对克罗诺杆菌(阪崎肠杆菌)的鉴定结果比较[J]. 中国卫生检验杂志, 2010, 20(3): 464-466.
[24] la SCOLA B, FOURNIER P E, RAOULT D. Burden of emerging anaerobes in the MALDI-TOF and 16S rRNA gene sequencing era[J]. Anaerobe, 2011, 17(3): 106-112.
[25] WOLTERS M, ROHDE H, MAIER T, et al. MALDI-TOF MS fingerprinting allows for discrimination of major methi-cillin-resistant Staphylococcus aureus lineages[J]. International Journal of Medical Microbiology, 2011, 301(1): 64-68.
Establishment and Application of an Analytical Database for Cronobacter spp. by Matrix-Assisted Laser Desorption Ionization-Time of Flight Mass Spectrometry
ZHAO Gui-ming, LIU Yang, CHEN Ying*, YANG Hai-rong, ZHAO Yong-sheng, WANG Ping
(Chinese Academy of Inspection and Quarantine, Beijing 100123, China)
The purpose of this study was to establish an analytical database for high throughput identification and subtyping of 8 species of Cronobacter spp. by a matrix-assisted laser desorption ionization time-of-flight mass spectrometry (MALDITOF-MS) method. Under the optimized culture and sample treatment conditions by using the established MALDI-TOFMS protein fingerprint acquisition parameters, the acquired characteristic protein mass spectrometric profiles of 8 reference strains were used to build a MALDI-TOF-MS database with Biotyper software, and the accuracy of the database was validated by comparison with data from related enterobacteriaceae strains and isolates of the genus Cronobacter. The MALDI-TOF-MS database was applied to analyze 135 isolates of Cronobacter spp. with an identification score no smaller than 2.0, which reached the requirement for identification at the species level. Further subtyping was achieved by cluster analysis. This MALDI-TOF-MS database may provide a new approach for high throughput identification and subtyping of Cronobacter strains.
Cronobacter spp.; MALDI-TOF-MS; database; identification; subtyping
TS207.4
A
1002-6630(2014)08-0105-06
10.7506/spkx1002-6630-201408020
2014-03-25
“十二五”国家科技支撑计划项目(2012BAD29B02)
赵贵明(1963—),男,研究员,研究方向为食品微生物。E-mail:zhcaiq@163.com
*通信作者:陈颖(1972—),女,研究员,博士,研究方向为食品安全。E-mail:chenyingcaiq@163.com
