考拉维酸对人血清白蛋白结构的影响
2014-02-18何文英姚小军华英杰黄国雷吴秀丽李小宝韩长日宋小平
何文英 姚小军 华英杰 黄国雷 吴秀丽李小宝 韩长日 宋小平,*
(1海南师范大学化学化工学院,海口571158;2兰州大学化学化工学院,兰州730000;3海南师范大学,热带药用植物化学教育部重点实验室,海口571158)
1 引言
考拉维酸(3-methyl-5-[(1S,2R,4aR,8aR)-1,2,3,4,4a,7,8,8a-octahydro-1,2,4a,5-tetramethyl-1-naphthalenyl]-(2E)-2-pentenoic acid),Kolavenic acid,简称KA,结构式如图1所示)为海南药用植物暗罗根首次分离得到的一种化合物.1研究表明,KA具有抗菌、拒食活性及多种细胞毒活性的药理作用.2作为一种潜在可开发利用的药物小分子,有关它影响人体内生物大分子的结构及其相互作用的报道却稀有.蛋白质是生命科学的研究对象中最重要的一种生物大分子,也是药物的一种非常重要的运输载体,通过研究KA与蛋白质的相互作用及所引起的氨基酸、构象的变化,不仅对于揭示体内KA药物动力学问题、指导其临床合理用药具有一定意义,而且对于设计考拉维酸类新药、筛选相关药物等都具有重要的指导意义.3-6
蛋白质的特定生理活性在很大程度上由其构象决定,这是判断药物小分子与生物大分子是否相互作用的一个重要指标.由于生命体中蛋白质多数以溶液状态存在,而光谱法是研究水溶液中蛋白质分子构象的最常用方法,如圆二色谱法、核磁共振法、荧光光谱法、紫外光谱法、红外光谱法等.7荧光光谱分析法能够提供包括发射光谱、激发光谱以及荧光强度、荧光偏振、荧光寿命、量子产率等多个物理化学参数,可从各个角度反映蛋白质分子的成键和结构情况以及发光特性.紫外光谱法可以考察在物理和化学因素的影响下,蛋白质二、三级结构的变化.8,9本文首次利用多种光谱法结合分子对接技术揭示了KA与HSA的相互作用情况,定性分析了KA对HSA二级结构、微环境及宏观结构的影响,并从分子水平上确定了它们之间的作用力模式及多种键合参数.
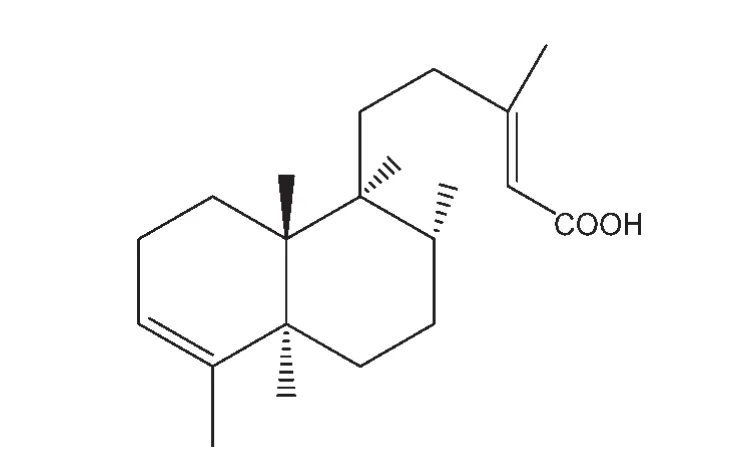
图1 考拉维酸的化学结构Fig.1 Chemical structure of Kolavenic acid
2 实验部分
2.1 仪器与试剂
F-7000荧光光度计(日本日立),RF-5301PC荧光光度计(日本岛津),配套的超级恒温槽控制实验所需温度,TU-1901双光束紫外-可见分光光度计(北京普析通用仪器有限责任公司),BS124S电子天平(德国赛多利斯),PH-3S计(上海精密仪器厂),Eppendorf移液器(德国).
人血清白蛋白(HSA,0.2 g∙mL-1,相对分子量66478,上海伯奥生物科技有限公司),用pH 7.40 Tris-HCl缓冲溶液配制,其储备液(3.0×10-5mol∙L-1)4°C保存于暗处.三羟甲基氨基甲烷(国药集团化学试剂有限公司,分析纯)-盐酸缓冲溶液(Tris-HCl):用超纯水溶解并用盐酸调节pH至7.40,配制成浓度为0.05 mol∙L-1的缓冲液.KA由海南师范大学热带药用植物化学教育部重点实验室提供(白色粉末,纯度>99.9%,相对分子量为304.47),储备液(1.70×10-3mol∙L-1),用二甲基亚砜配制.实验水为超纯水,其他试剂均为分析纯.
2.2 二维及三维荧光光谱
二维荧光光谱在RF-5301PC(Shimadzu)荧光光谱仪上进行测定.移取一定量的HSA溶液于3 mL比色皿中,逐次加入一定量KA溶液.以285 nm为激发波长(λex),激发和发射狭缝宽度均为5 nm,扫描295-500 nm范围内的荧光光谱(1 cm石英池).
三维荧光光谱在F-7000(Hitachi)荧光光谱仪上进行测定:溶液配制方法同上二维荧光光谱的测定.选择荧光激发和发射狭缝宽度均为5 nm(1 cm石英池),分别测定激发波长范围在230-325 nm,发射波长(λem)在230-480 nm范围的三维荧光光谱.
2.3 同步荧光测定
同步荧光测定在RF-5301PC(Shimadzu)荧光光谱仪上进行,以230 nm为激发波长,290 nm为发射波长(Δλ=60 nm),测定加入不同浓度KA后HSA-KA溶液体系的同步荧光光谱(激发和发射狭缝宽度均为5 nm).
2.4 紫外吸收光谱
在若干10 mL比色管中,依次移取一定量的HSA和KA储备液,用pH 7.40的Tris-HCl缓冲溶液定容,以相应试剂的空白为参比,在紫外-可见分光光度计上记录HSA-KA体系在200-350 nm范围内的紫外吸收光谱(1 cm石英池).
2.5 荧光偏振度的测定
依据如下各向异性的定义:10,11
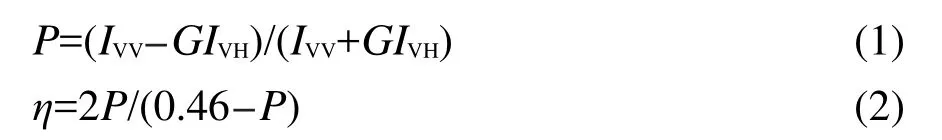
其中,P为荧光偏振度,IVV和IVH分别为垂直偏振光激发后的垂直偏振和水平偏振的发射光强度,η为粘度.G为仪器校正因子,G=IHV/IHH,IHV和IHH分别为水平偏振光激发下的垂直偏振和水平偏振光的发射光强度.选激发与发射波的狭缝宽均为5 nm,激发波长为285 nm,发射波长为348 nm,在RF-5301PC(Shimadzu)荧光光谱仪上进行测定(1 cm石英池).
2.6 键合参数及热力学参数的测定
配制不同浓度比的HSA-KA溶液体系,选择激发波长和发射波长分别为285和348 nm,采用荧光滴定法,测定三种不同温度(298、308和318 K)下的荧光强度,再依据相应的公式计算结合常数.
2.7 分子模拟
从Brookhaven蛋白质数据库中获得HSA的晶体结构(编号为1 h9z),根据Amber 4.0力场,用Kollman-all-atom电荷计算出HSA三维结构的势能;12再用Gasteiger-marsili软件和tripos力场优化HSA分子的几何结构,用AutoDock 3.05程序来确定KA与HSA分子之间的相互作用模式,最后用拉马克(LGA)遗传算法来计算KA分子与HSA结合的可能构象,13,14所有的计算在Silicon Graphics Ocatane 2工作站上完成.
2.8 位点标记的竞争实验
在RF-5301PC(Shimadzu)荧光光谱仪上进行,采用荧光滴定法,固定HSA的浓度,向KA-HSA体系分别加入一定量的竞争试剂苯基丁氮酮(phenylbutazone,PB)、氟芬那酸(flufenamic acid,FA)和洋地黄毒苷(digitoxin,Dig),分别作为位点I、II和III位的标记药物.15选择激发波长和发射波长分别为285和348 nm,测定HSA-KA体系在298 K的荧光强度.
3 结果与讨论
3.1 光谱法定性分析KA影响HSA的二级结构
作为血浆中最为丰富的球状蛋白质,血清白蛋白含有多种可配位基团,可以与生物体内许多内源或外源物质相互作用,因此在药物小分子与蛋白质相互作用的研究中常用作模型蛋白.3,4
利用紫外光谱分析HSA的光谱特征时,一般约210 nm处的峰是肽键的强吸收峰,其最大吸收峰反映了其α-螺旋结构的信息.16图2为HSA、KA及KAHSA体系的紫外光谱图.可看出:KA(5.6×10-6mol∙L-1)具有较强的紫外吸收,其最大吸收波长位于223 nm左右,吸收强度约为2.6;HSA(5.0×10-6mol∙L-1)最大吸收波长位于214 nm左右,吸收强度约为2.3;随KA浓度的增加,HSA-KA体系发生最大吸收峰位置红移现象(214 nm➝225 nm),且体系的吸收强度并非是KA与HSA的加和.以上结果说明:KA确实与HSA发生了相互作用,使得HSA的α-螺旋结构发生一定的变化.
在蛋白质分子中,色氨酸Trp残基的荧光强度最大,对微环境的变化很敏感,故常作为内源荧光探针来研究溶液状态下蛋白质的构象,而同步荧光(Δλ=60 nm)呈现的是Trp残基的光谱特征.7,8图3为采用同步荧光扫描法,固定HSA浓度,依次增大KA浓度所测得的同步荧光光谱图,从图中可看出:KA的存在猝灭了HSA的Trp残基荧光,荧光强度从150降至106左右,其最大荧光发射峰位置基本未变,但在KA浓度较大时,有较明显的双峰出现迹象.说明KA对HSA微环境有一定影响.
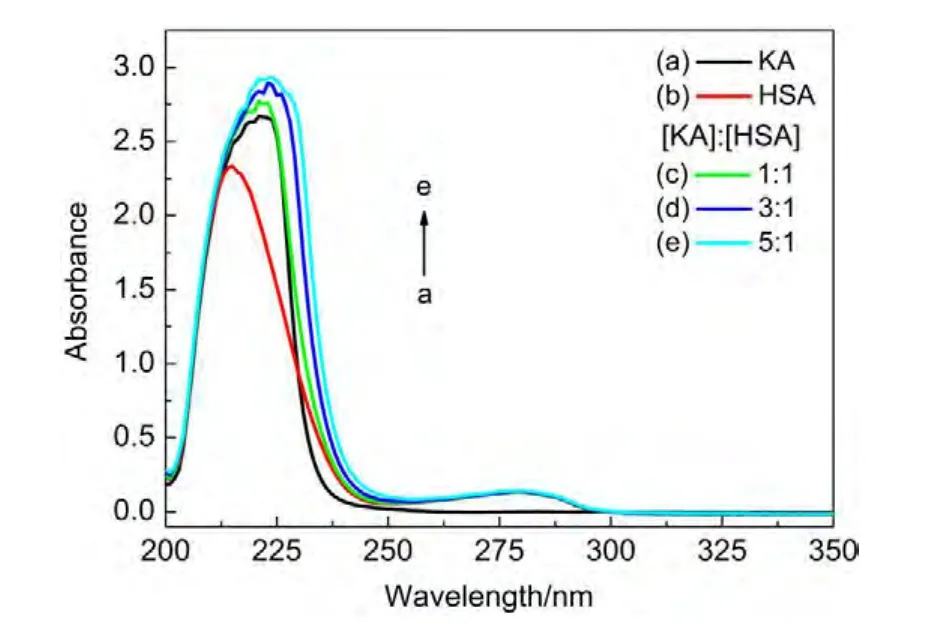
图2 KA-HSA体系的紫外吸收光谱图Fig.2 UV absorption spectra of KA-HSAsystem
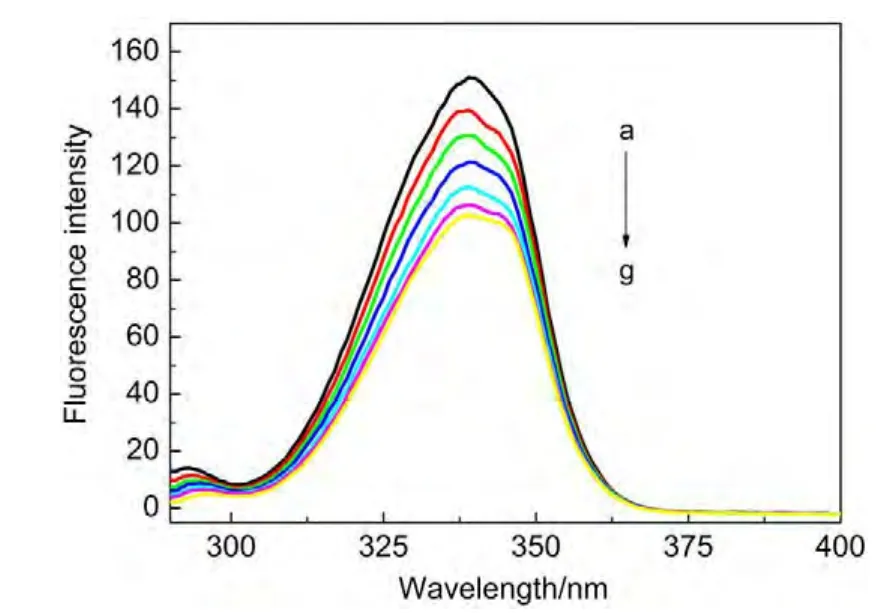
图3 KA-HSA体系的同步荧光光谱图Fig.3 Synchronous fluorescence spectra of KA-HSAsystems
在同一测定条件下,从KA-HSA体系的二维及三维荧光光谱来研究对蛋白结构的影响.图4及图5为固定HSA浓度,变化KA浓度时得到的二维及三维荧光光谱.从图4及图5中可见,随KA浓度的加大,相比不加KA时的HSA(图4中曲线a及图5A),HSA-KA体系的荧光强度逐渐减小(图4中曲线b➝h及图5B),即KA猝灭了HSA的内源色氨酸残基的荧光,且两种荧光谱图的峰型均呈现规则形状,并伴随有红移现象:二维谱(图4A)显示最大发射波长从348 nm轻微红移至350 nm,三维谱(图5B)则从350 nm红移至355 nm.对谱型规则及红移可能归结为两个原因:一是加入KA后,使得HSA的微环境极性增大,从而形成规则的发射光谱;再是由于KA的分子结构中存在的羧基与双键,形成的共轭体系使得双键上的C原子为给电子基团,导致荧光光谱产生位移,及吸收光强度的增大(见图2).17以上均说明了KA的存在对HSA的色氨酸残基所处的微环境引起的差异,也表明了HSA分子中构象的变化.
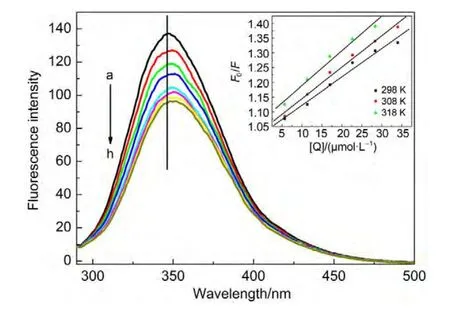
图4 KA-HSA体系的荧光光谱Fig.4 Fluorescence spectra of KA-HSAsystem
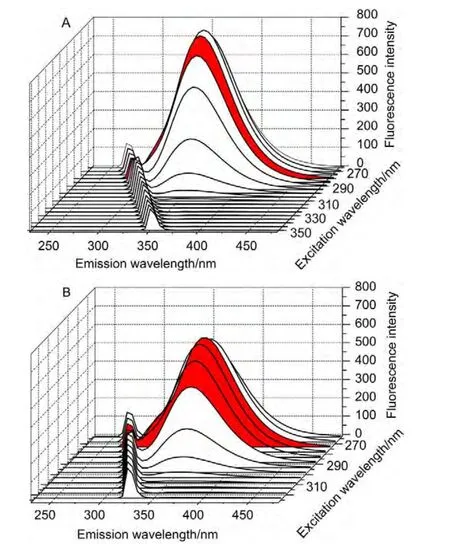
图5 HSA(A)和KA-HSA体系(B)的三维荧光光谱图Fig.5 Three dimensional(3D)fluorescence spectra of HSA(A)and KA-HSAsystem(B)
根据荧光光谱测得的相关数据,确定了KA猝灭与HSA之间的荧光猝灭机理.利用Stern-Volmer方程,将F0/F对[Q]作线性拟合图,通过该直线的斜率可得到猝灭常数(KSV),其中,F0为游离状态下HSA的荧光发射强度,F为加入KA后与HSA形成复合物后的体系荧光发射强度,[Q]为KA的浓度.由于荧光分子与猝灭剂之间的猝灭效率常数都遵循:Kq=KSV/τ0,τ0为没有猝灭剂存在下荧光分子的平均寿命(对大多数的生物分子τ0大约是10-8s),各类猝灭剂对生物大分子的最大扩散碰撞猝灭常数Kq为2.0×1010L∙mol-1∙s-1,如果猝灭速率常数小于这个最大值,则说明猝灭过程主要为动态猝灭机理;反之,猝灭速率常数大于这个最大值,则猝灭过程可能为静态猝灭过程,还需通过测定体系的吸收光谱是否有变化来确定.8,11
在模拟生理条件(pH 7.40的Tris缓冲液)下,选取三个不同温度(298、308及318 K)进行荧光滴定的测定,将所得数据作KA对HSA荧光猝灭的Stern-Volmer图及KSV值计算(图4中的插图及表1).可看出,在选定的浓度范围内,猝灭曲线的斜率即猝灭常数KSV值均为104数量级,猝灭速率常数远远大于2.0×1010L∙mol-1∙s-1,再结合吸收光谱(图2),表明KA对HSA的荧光猝灭机理为静态猝灭.猝灭常数KSV值用于计算热力学参数.
3.2 键合参数及热力学参数的确定
利用荧光猝灭法的有关理论及公式来确定KA与HSA作用的键合常数、键合位点数及作用力形式.依据Scatchard公式求得键合常数及键合位点数,3见表1及图6,可看出KA与HSA的键合常数K值都较大,且随温度的增大,K值呈增大趋势,而键合位点数n呈减小趋势.说明KA对HSA的键合作用较强,且温度的变化会影响键合常数的大小.
不同小分子与HSA结合的作用力类型不同,根据Van′tHoff定律及公式可求出反应的热力学参数,进而大致确定其作用力类型,当温度变化不太大时,反应的焓变可看作一个常数,由lnK对1/T作图(未列出),可求得药物与HSA相互作用的热力学常数(见表1).18
从表1中可看出:正的ΔS值(103.112 J∙mol-1∙K-1)及ΔH值(8.003 kJ∙mol-1)表明KA主要以疏水作用键合HSA,负ΔG值则表明这种键合过程是自发进行的.这也与文中后面的分子模拟的结果相一致,再次证明KA键合在HSA亚结构域的疏水腔.结合热力学计算与分子模拟的结果,可得出:KA主要以疏水作用及氢键键合HSA.
3.3 荧光偏振结果
为进一步从分子水平上探讨KA影响HSA的结构及其键合情况,采用了荧光偏振法.荧光偏振法可阐明溶液状态下生物大分子的构象以及相应的生物活性,通过荧光偏振或各向异性值的变化,可了解药物与蛋白结合的一些结构信息.10,19图7为309与319 K两个不同温度的条件下,改变KA的浓度而测得的HSA的荧光偏振值,其中的插图为改变KA的浓度而测得的HSA的粘度η值.可看出,较高温度(319 K)的偏振值均稍高于较低温度(309 K)的,并且随着KA浓度比的增大(0➝28.30×10-6mol∙L-1),偏振值都逐渐减小,在319 K HSA的偏振值下降幅度(0.041➝0.012)比309 K的大(0.027➝0.0009).插图中的粘度η值也显示了相似的变化趋势.结果说明:一方面,较小的偏振值(均小于0.1)暗示KA与HSA结合比较松散,结合后的大分子弛豫时间短,使HSA在从螺旋到无规卷曲伸展变化时,由于挠性增大,故荧光偏振就小;20另一方面,KA对HSA的偏振值表现为下调作用,使HSA的微粘度减小,流动性增加,并且随温度的增大,微粘度下降幅度逐渐增强,说明HSA的流动性随KA的浓度及温度依赖性的增大,21也证实了KA的存在引起HSA的微结构及二级结构发生了变化.
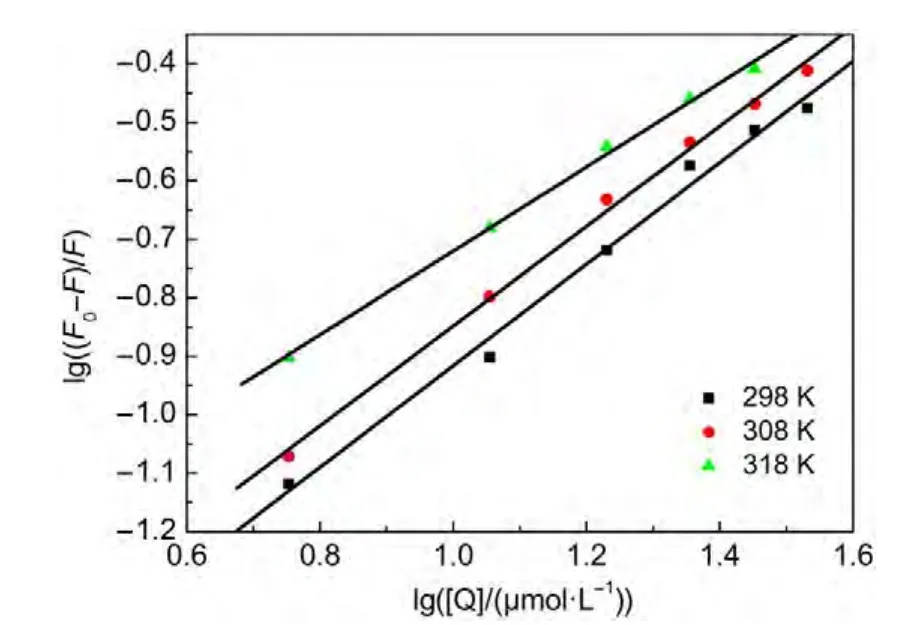
图6 KA-HSA体系的Scatchard图Fig.6 Scatchard plots for KA-HSAsystem
3.4 KA与HSA作用的分子模拟
采用计算机化学的分子对接技术,确定KA在HSA的作用区域及位点,进一步从理论上证实KA与HSA的相互作用,这有利于深入认识它在生物体系中所发挥的重要功能.HSA分子分为三个结构域,它们又分别含有A、B两个亚结构域,并以相对的槽口方式构成圆筒状的一疏水腔,腔内不仅是大多小分子键合HSA的主要区域,也包埋了几乎所有疏水性氨基酸残基.22
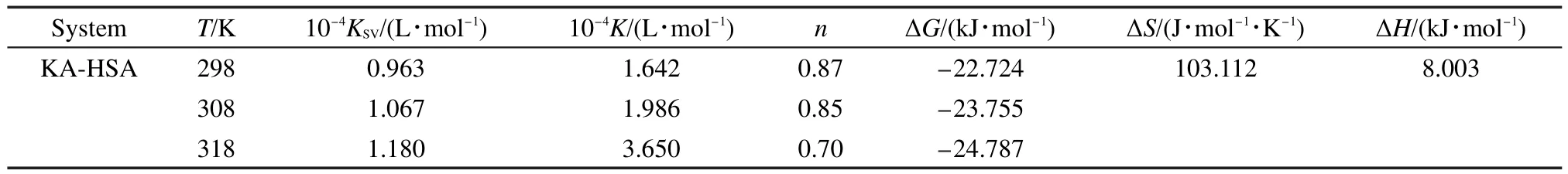
表1 KA-HSA体系键合值和热力学参数比较Table 1 Comparison of the binding values and thermodynamic functions for KA-HSAsystems
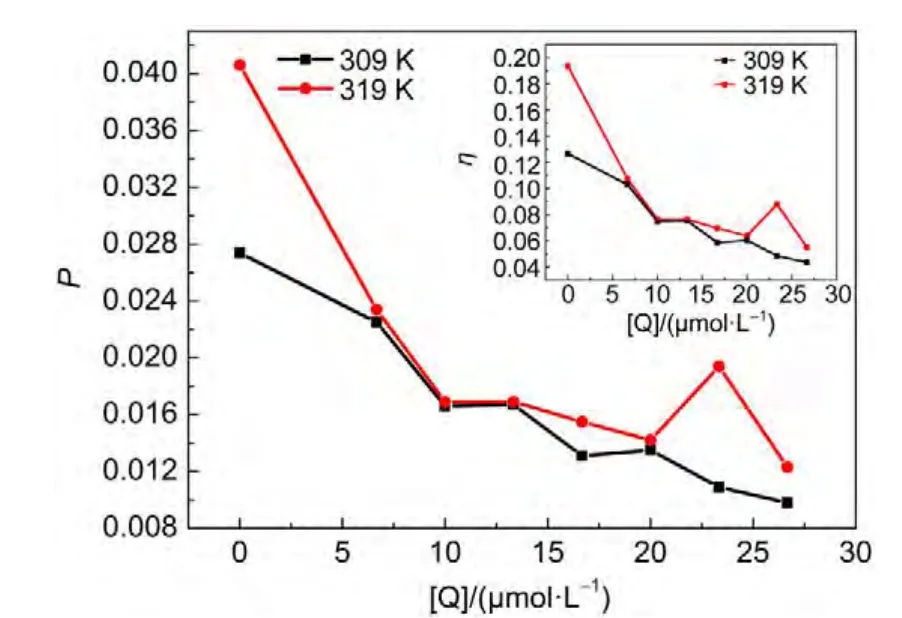
图7 HSA(3.0 μmol∙L-1)的荧光偏振值(P)随KA浓度的变化Fig.7 Variation in the fluorescence polarization(P)of HSA(3.0 μmol∙L-1)with KAconcentration
图8为KA键合HSA的飘带状分子模拟对接图.从图中可看出,KA分子可以键合在HSA分子的疏水腔内,整个分子呈非平面结构,且很靠近HSA分子的苯丙氨酸Phe211和色氨酸Trp214,这不仅是KA与HSA的键合存在疏水作用的一个有力证据,也表明KA能猝灭HSA的內源荧光,这两点在前面的有关荧光光谱法的测定结果中得到很好的证实.另外,KA与HSA氨基酸残基的赖氨酸Lys195位和天冬氨酸Asp451位形成三个氢键.计算得到相应的吉布斯自由能ΔG值为-25.050 kJ∙mol-1,与实验数据(ΔG=-22.724 kJ∙mol-1)较接近.这些结果均说明KA键合HSA的反应是自发进行的,作用力的模式主要是疏水作用,兼有部分氢键作用.

图8 KA与HSA的飘带状模型对接图Fig.8 Zonal binding mode between KAand HSA
3.5 竞争位点的实验结果
为确定KA在HSA上的结合位置,选择对HSA具有特异结合性的三种竞争试剂(苯基丁氮酮(PB)、氟芬那酸(FA)和洋地黄毒苷(Dig))分别作为位点I、位点II、位点III位的标记药物,15,23通过探针标记药物存在时KA与HSA的结合常数的变化来判断其结合位置.在分别含一定浓度的以上三种竞争试剂的存在条件下,用荧光滴定法测定不同KA-HSA体系的荧光强度,用双倒数公式处理所得的数据.24与不加竞争试剂时KA-HSA体系的键合常数相比(2.804×104L∙mol-1),发现键合常数均发生了变化(见图9),分别为:3.542×104L∙mol-1(PB),4.670×104L∙mol-1(FA),3.491×104L∙mol-1(Dig).其中,变化幅度最大的为FA标记物,即说明FA与KA竞争键合HSA的相同位点,或者可以说KA在位点II位与HSA发生作用.
3.6 KA与HSA键合的几种物理化学参数
将光谱法测得的有关数据,用相关的分析化学公式处理.8,25,26得到离解常数、电荷密度及量子产率(以色氨酸在激发波长280 nm的荧光量子产率0.14为标准)几个物理化学参数值,这有助于深入揭示KA与HSA的键合情况及发光特性,结果列于表2.从表2中可看出,在给定的KA浓度范围内(约6-40 μmol∙L-1,体系的离解常数pKa值呈升高趋势,但是在高浓度时又稍微降低,pKa值(Ka数量级10-8)均较小,再次说明KA与HSA有强的键合作用;对体系的电荷密度δ值,随着KA浓度的加入有减小,而且最大的值未大于0.5,说明静电作用并非是稳定KAHSA体系的主要作用力,这与前面所讨论的键合模式的结果相一致;而量子产率Φ值呈现较明显的规律降低趋势,表明KA可以猝灭HSA的荧光强度,也符合文中前的荧光光谱测定结果.
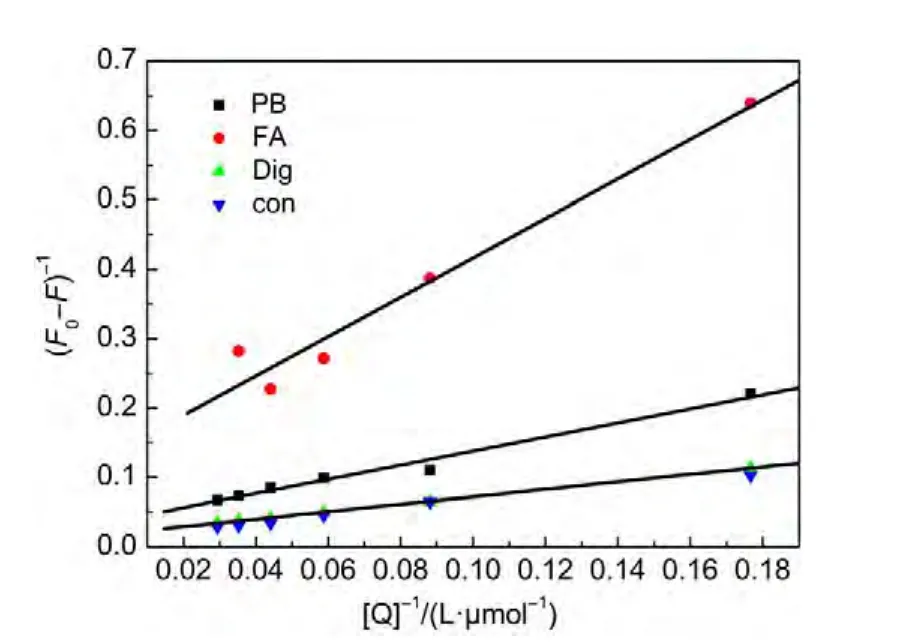
图9 位点标记竞争实验结果图Fig.9 Results of site marker competitive experiments
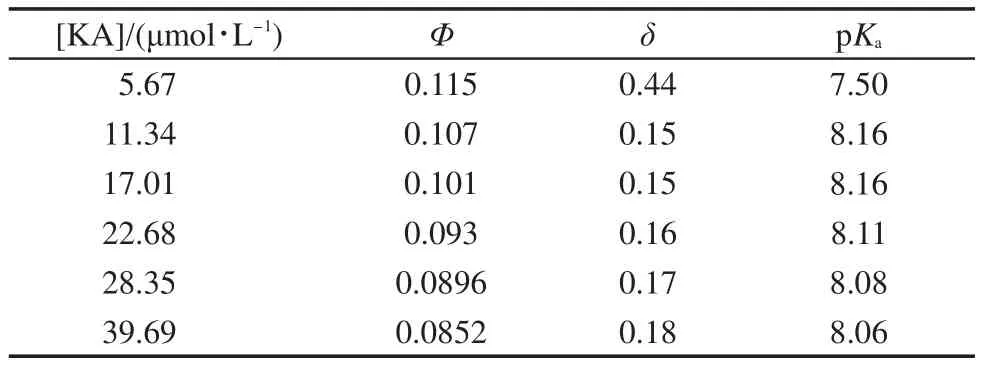
表2 KA-HSA体系的部分物理化学参数Table 2 Some parameters of physical properties for KA-HSAsystem
4 结论
本文利用多种光谱法及分子模拟方法,首次研究了海南暗罗根活性组分考拉维酸影响人血清白蛋白的结构特征.分子模拟及位点竞争实验确定了KA在HSA上的位点II键合.多种荧光光谱法表明KA的存在影响了HSA的微环境及二级结构;紫外光谱图表征了KA对HSA构象的影响.热力学参数表明KA键合HSA的模式主要为疏水作用;获得的不同温度下的键合常数(104数量级)说明KA与HSA有较强的键合作用.并结合KA-HSA体系的几种物理化参数,从分子水平上阐明了KA与模型蛋白的键合反应及作用机制.
(1) Li,X.B.Studies on Chemical Constituents and Pharmacogical Activities of the Roots ofPolyalthia laui.Master Dissertation,Hainan Normal University,Haikou,2012.[李小宝.海南暗罗根化学成分及其药理活性研究[M].海口:海南师范大学,2012.]
(2)http://www.med126.com/pharm/2010/20100123233057_164906.shtml,2010(accessed July 1,2014).
(3) Liu,Y.;Long,M.;Xie,M.X.Acta Phys.-Chim.Sin.2013,29,2647.[刘 媛,龙 梅,谢孟峡.物理化学学报,2013,29,2647.]doi:10.3866/PKU.WHXB201310311
(4)Ning,A.M.;Meng,L.;Zhao,Z.L.;Zheng,X.F.;Wan,X.S.Acta Phys.-Chim.Sin.2013,29,2639.[宁爱民,孟 磊,赵仲麟,郑先福,宛新生.物理化学学报,2013,29,2639.]doi:10.3866/PKU.WHXB201310281
(5) Gowda,J.I.;Nandibewoor,S.T.Spectrochimica Acta Part A2014,124,397.doi:10.1016/j.saa.2014.01.028
(6) Tunc,S.;Duman,O.;Soylu,I.;Bozoğlan,B.J.Lumin.2014,151,22.doi:10.1016/j.jlumin.2014.02.004
(7) Tao,W.S.;Li,W.;Jiang,Y.M.The Basic of Protein Molecules,2nd ed.;Higher Education Press:Beijing,1995;pp 350-355.[陶慰孙,李 惟,姜涌明.蛋白质分子基础.第二版.北京:高等教育出版社,1995:350-355.]
(8) Yang,P.;Gao,F.Principles of Bioinorganic Chemistry;Science Press:Beijing,2002;pp 322-342.[杨 频,高 飞.生物无机化学原理.北京:科学出版社,2002:322-342.]
(9) Zhu,B.;Du,X.L.;Li,R.C.;Wang,K.;Jin,J.;Wang,B.C.Chem.J.Chin.Univ.2001,22,26.[朱 兵,杜秀莲,李荣昌,王 夔,金 坚,王博诚.高等学校化学学报,2001,22,26.]
(10) Liu,G.Q.;Yue,H.Q.;Zhang,Y.Mod.Instruments Med.Treat.2004,6,33.[刘桂琴,岳慧琴,张 勇.现代仪器与医疗,2004,6,33.]
(11) Lakowicz,J.R.Principles of Fluorescence Spectroscopy;Plenum:New York,1983;pp 341-379.
(12) SYBYL Software,Version 6.9;TriposAssociates Inc.:St.Louis,MO,USA,2002.
(13)Morris,G.M.;Goodsell,D.S.;Huey,R.;Olson,A.J.J.Comput-Aided.Mol.Des.1996,10,293.doi:10.1007/BF00124499
(14) Morris,G.M.;Goodsell,D.S.;Halliday,R.S.;Huey,R.;Hart,W.E.;Belew,R.K.;Olson,A.J.J.Comput.Chem.1998,19,1639.
(15)Carter,D.C.;He,X.M.;Munson,S.H.;Twigg,P.D.;Gernert,K.M.;Broom,M.B.;Miller,T.Y.Science1989,244,1195.doi:10.1126/science.2727704
(16)Wolfbeis,O.S.;Leiner,M.;Hochmuth,P.;Geiger,H.;Bunsenges,B.Bunsenges.Phys.Chem.1984,88,759.doi:10.1002/bbpc.19840880817
(17)Valeur,B.Molecular Fluorescence:Principles and Applications;Wiley-VCH Verlag GmbH.ISBNs:3-527-29919-X(Hardcover);3-527-60024-8(Electronic).2001;pp 56-68.
(18) Ross,P.D.;Subramanian,S.Biochemistry1981,20,3096.doi:10.1021/bi00514a017
(19) Hill,J.J.;Royer,C.A.Methods in Enzymology;Academic Press:San Diego,1997;pp 390-416.
(20) Kido,C.;Murano,S.;Tsuruoka,M.Gene2000,259,123.doi:10.1016/S0378-1119(00)00457-1
(21)Yang,Y.;Ye,X.L.;Li,X.G.Negative2009,17,1627.[杨勇,叶小利,李学刚.医学争鸣,2009,17,1627.]
(22) Sudlow,G.;Birkett,D.J.;Wade,D.N.Mol.Pharmacol.1976,12,1052.
(23)Khan,A.M.;Muzammil,S.;Musarrat,J.Int.J.Biol.Macromol.2002,30,243.doi:10.1016/S0141-8130(02)00038-7
(24)Mohammed,H.R.;Toru,M.;Tomoko,O.;Keishi,Y.;Masaki,O.Biochem.Pharm.1993,46,1721.doi:10.1016/0006-2952(93)90576-I
(25) Wuhan University.Analytical Chemistry;People Press:Beijing,1978;pp 131-132.[武汉大学.分析化学.北京:人民出版社,1978:131-132.]
(26)Gao,H.;Lei,L.D.;Liu,J.Q.;Kong,Q.;Chen,X.G.;Hu,Z.D.J.Photochem.Photobiol.A2004,167,213.doi:10.1016/j.jphotochem.2004.05.017
