牦牛雌激素受体ERα和 ERβ 基因分子特征及组织表达研究
2014-02-13李键符梅兰道亮熊显荣
李键, 符梅, 兰道亮, 熊显荣
(1.西南民族大学青藏高原研究院, 四川 成都 610041; 2.西南民族大学生命科学与技术学院, 四川 成都 610041)
【特约专稿】
牦牛雌激素受体ERα和 ERβ 基因分子特征及组织表达研究
李键1, 符梅2, 兰道亮1, 熊显荣2
(1.西南民族大学青藏高原研究院, 四川 成都 610041; 2.西南民族大学生命科学与技术学院, 四川 成都 610041)
为研究牦牛雌激素受体(ERα和ERβ)基因序列特征及其在不同组织中的表达差异.根据NCBI上已公布的普通牛ERα和ERβ基因序列, 设计特异性引物, 利用RT-PCR技术克隆麦洼牦牛ERα和ERβ基因序列, 同时利用荧光定量PCR技术检测ERα和ERβ基因在牦牛不同组织的表达分布.结果表明, 分别获得2 100 bp ERα(GenBanK登录号: KJ011123)和1 791 bp ERβ(GenBanK登录号: KJ011124)基因序列, 其中ERα的ORF为1 658 bp, 编码596个氨基酸, ERβ的ORF为1 584 bp, 编码527个氨基酸.多重序列分析表明, 牦牛ERα和ERβ与普通牛、原鸡、人、小鼠、大鼠、扬子鳄、蟾蜍及斑马鱼的氨基酸序列同源性分别为45.3%-99.5% 和 53.9%-99.1%.荧光定量PCR结果显示, ERα和ERβ基因在牦牛的各种组织中均有表达, 且除睾丸外, ERα在输卵管、子宫及乳腺中的表达高于ERβ.此外, 就ERα而言, ERα在牦牛乳腺中表达最高, 子宫、输卵管及卵巢次之, 心、肝、脾、肺、肾及睾丸相对表达较低.而牦牛ERβ基因在卵巢中表达最高, 子宫和输卵管次之, 心、肝、脾、肺、肾、睾丸及乳腺表达较低.为进一步了解牦牛ERα 和 ERβ基因的生物学功能及其分子繁殖机制提供了基础.
牦牛; ERα基因; ERβ基因; 基因特征
牦牛生活在青藏高原海拔3 000米以上的地区, 其对低温、低氧、低压的高原环境有较强的适应能力, 能充分利用其他家畜难以利用的高山草原, 是高原地区牧民的主要经济来源[1].然而牦牛三年两胎或两年一胎的繁殖能力普遍低于其它普通牛属动物.
雌激素是一种甾体激素, 是人类及其他动物体内最重要的激素之一, 具有广泛的生理功能.其广泛的生理作用是通过特定的细胞表面受体雌激素受体(Estrogen receptor, ER)实现的.雌激素受体是核受体超家族的一种配体依懒性转录因子, 与雌激素专一性的结合, 通过与雌激素的相互作用来调节与生殖机能相关基因的表达[2].至今发现的雌激素受体有两种类型, 即ERα和ERβ.国内外大量的研究显示ERs基因与哺乳动物的卵泡发育和高繁殖力有密切的关系[3-4], 对雌激素受体的研究将有助于了解动物卵泡发育机制及繁殖能力高低机制, 因此雌激素受体的研究受到了广泛的关注.自1962年Jenson等发现子宫、阴道等靶组织存在ERs以后, 学者们便开始了对ERs基因的深入研究.尹玉涛[5]等的研究表明, ERα对腺管伸长和上皮分化起到促进作用.白淑[6]等对济宁青山羊出生后发育过程中雌激素受体在子宫中的作用研究表明雌激素参与调节子宫内膜与肌层的增殖和分化.PAUL等对两种雌激素受体在人和鼠生殖器官中分布的研究表明, 在卵巢、睾丸及前列腺中, ERβ表达占据主导作用[7], 而在子宫和附睾中ERα较ERβ优势表达[8].此外, 内分泌学和生理学研究发现, 雌激素受体广泛分布于消化系统, 能影响胃、小肠和结肠等消化器官的机能[9].目前为止, 已经有多种哺乳类动物[10], 鸟类[11-12], 爬行类[13], 两栖类[14-15]及鱼类[16-17]的ERs基因被克隆.但目前关于牦牛的雌激素受体还未见报道, 因此本研究以麦洼牦牛为例, 利用RT-PCR及荧光定量PCR技术克隆了牦牛ERs基因, 并检测了ERs mRNA在不同组织中表达情况, 为进一步探究牦牛的繁殖机理提供了基础.
1 与方法
1.1 材料
试验用牦牛(品种: 麦洼)来自四川省阿坝州红原县屠宰场, 屠宰后分别采集3头公牦牛与3头母牦牛心、肝、脾、肺、肾、胃、子宫、输卵管、睾丸、乳腺和卵巢10种组织, 用锡箔纸将各组织包好后立即投入液氮中速冻,之后-80 ℃保存备用.
1.2 设备和试剂
Trizol试剂购自Invitrogen公司; PrimeScriptTMRT reagent Kit反转录试剂盒、SYBR®Premix Ex TaqTMII试剂盒和pMD-19T载体均购自TaKaRa公司.DNA胶回收试剂盒、感受态、DEPC和Marker等均购自北京天根公司.荧光定量PCR仪(Bio-Rad)、PCR仪(Eppendorf)、Doc 2000凝胶成像系统(Bio-Rad).
1.3 方法
1.3.1 的设计与合成
根据NCBI上公布的普通牛ERα基因mRNA序列(GenBank登录号: NM_001001443.1)和ERβ基因mRNA序列(GenBank登录: NM_174051.3), 采用Prime5软件分别设计四对引物(ERα1、ERα2、ERβ1及ERβ2)分别扩增牦牛ERα基因和ERβ基因的编码区序列.参照扩增出的牦牛ERα和ERβ基因系列设计ERs基因定量引物, 选用GADPH基因(GenBank登录号: AC_000162.1)作为看家基因.引物送由上海Invitrogen公司合成.引物扩增长度、登录号及引物序列见(表1).1.3.2 总RNA的提取和cDNA第一链的合成

表1 隆和定量的引物序列Table 1 Primes used for molecular cloning and Q-PCR in this study
液氮中取出牦牛各组织, 按照Trizol试剂盒说明书提取组织总RNA, 1%甲醛变性凝胶电泳检测RNA的质量,并用紫外分光光度计测定总RNA的浓度及纯度.按照PrimeScriptTMreagent Kit(TakaRa,Dalian,China)说明书进行反转录.体系为10 μL: 包括 5×PrimeScript Buffer 2 μL, PrimeScript RT Enzyme Mix I 0.5 μL, Oligo dT Primer 0.5 μL, Random 6 mers 0.5 μL, total RNA 6.5 μL.反应条件为: 37 ℃ 15min, 85 ℃ 5s.反转录产物臵于-20 ℃保存备用.
1.3.3 RT-PCR扩增、克隆及测序
以反转录的cDNA做模板进行PCR反应.PCR扩增体系25 μL: 2×taq Master Mix 12.5 μL, 上、下游引物(10μmol·L-1)各1 μL, 模板1μL, 加ddH2O补足25 μL.PCR反应条件: 94 ℃ 5 min; 94 ℃ 30 s, 60 ℃ 30 s, 72 ℃ 1 min, 35个循环; 72 ℃ 再延伸5 min, 4 ℃ 保存.1%的琼脂糖凝胶电泳检测PCR扩增产物.用DNA胶回收试剂盒回收琼脂糖凝胶电泳检测到的目的条带, 然后将胶回收产物连于pMD-19T载体, 转化大肠杆菌(DH5α), 37 ℃ LB平板上培养过夜.经蓝白斑筛选, 挑取单个白色菌落于液体LB培养基中37 ℃ 200 r/min 振荡6 h.PCR鉴定后将阳性克隆送由上海Invitrogen公司测序.
1.3.4 序列分析与结构预测
测序结果用DNAMAN7.0 软件进行序列对比和拼接.用NCBI的blastn程序在GenBank数据库中对比鉴定,用Protparam软件(http // web.expasy.org/protparam)分析其蛋白质的基本理化性质; 用DNAstar将其编码区翻译成氨基酸序列; 采用在线软件(http://www.ebi.ac.uk/interpro/scan.html)预测功能结构域; 分子进化树构建采用MEGA5.0软件.
1.3.5 实时荧光定量PCR反应
参照SYBR®Premix EX TaqTM II试剂盒进行荧光定量PCR.反应体系为SYBR®Premix EX TaqTM II 10 μL,上、下游引物(10μmol·L-1)各0.5 μL, cDNA 1 μL, 加ddH2O补足20 μL.PCR反应条件为95 ℃ 预变性30 s; 95 ℃变性5 s, 60 ℃退火30 s, 72 ℃延伸30 s, 40个循环.溶解曲线分析: 95 ℃ 10 s, 60 ℃ 1 min, 然后以0.5 ℃·10s-1的速率从60 ℃缓慢升温到95 ℃.每个样品重复3次.荧光定量结果采用2-△△Ct法进行分析[18].
1.3.6 数据统计与分析
数据运用SPSS17.0软件进行分析, 结果用“平均值±标准误(mean±SE)”表示, 采用ANOVA和T检验进行显著性差异分析.结果判定: 当P<0.05时, 认为差异显著.
2 结果与分析
2.1 牦牛ERα和ERβ基因克隆测序结果分析
与预期结果一样, 我们获得了820 bp和1 280 bp的ERα扩增产物(图1), 其中ORF为1 791 bp, 编码596个氨基酸残基, 蛋白质分子质量为66.5 kDa.测序结果提交GenBank已获得基因登录号(KJ011123).通过与普通牛(NM001001443.1)、人(NM000125.3)、小鼠(NM007956.4)、大鼠(NM012689.1)、原鸡(NM205183.2)、扬子鳄(XM006024833.1)、蟾蜍(AF383157.1)及斑马鱼(NM152959.1)的氨基酸同源性对比发现, 麦洼牦牛ERa基因氨基酸序列与普通牛及人的同源性最高, 分别为99.5%和91.1%.然后仅与斑马鱼有45.3%的相似性(表2).ERβ的PCR扩增产物分别为783 bp 和875 bp(图1), 其中包括1 584 bp 的ORF, 编码527个氨基酸残基, 预测的蛋白质的分子质量为59.0 kDa.测序结果提交GenBank已获得基因登录号(KJ011124).与普通牛(NM174051.3)、原鸡(NM204794.2)、人(NM001437.2)、小鼠(NM207707.1)、大鼠(NM012754.1)、扬子鳄(XM_006018673.1)、蟾蜍(NM001130954.1)及斑马鱼(NP851297.1)的同源性分别为9.0%, 70.5%, 78.8%, 80.9%, 96.5%, 87.8%, 79.6% and 91.9%.与很多核受体一样, ERα和ERβ都包含了六个基本的功能结构域(图2).而牦牛ERα基因和ERβ基因的C和E结构域从鱼类到两栖类都有很高的相似性(C domain: 94%-99% 氨基酸相似性; E domain: 58%-94%氨基酸相似性)(图3, 图4).ERα和ERβ都包含了8个半胱氨酸残基(图5).

图1 麦洼牦牛ERα和ERβ基因的RT-PCR扩增产物Fig.1 RT-PCR amplification of ERαand ERβgene of yak

表2 麦洼牦牛ERα和ERβ氨基酸序列与其他物种相似性比较Table 2 Similarities of amino acid of the ERα and ERβ of MaiWa yak to other species

图2 牦牛ERα和ERβ结构域的比较Fig.2 The comparison of ERα and ERβ of yak
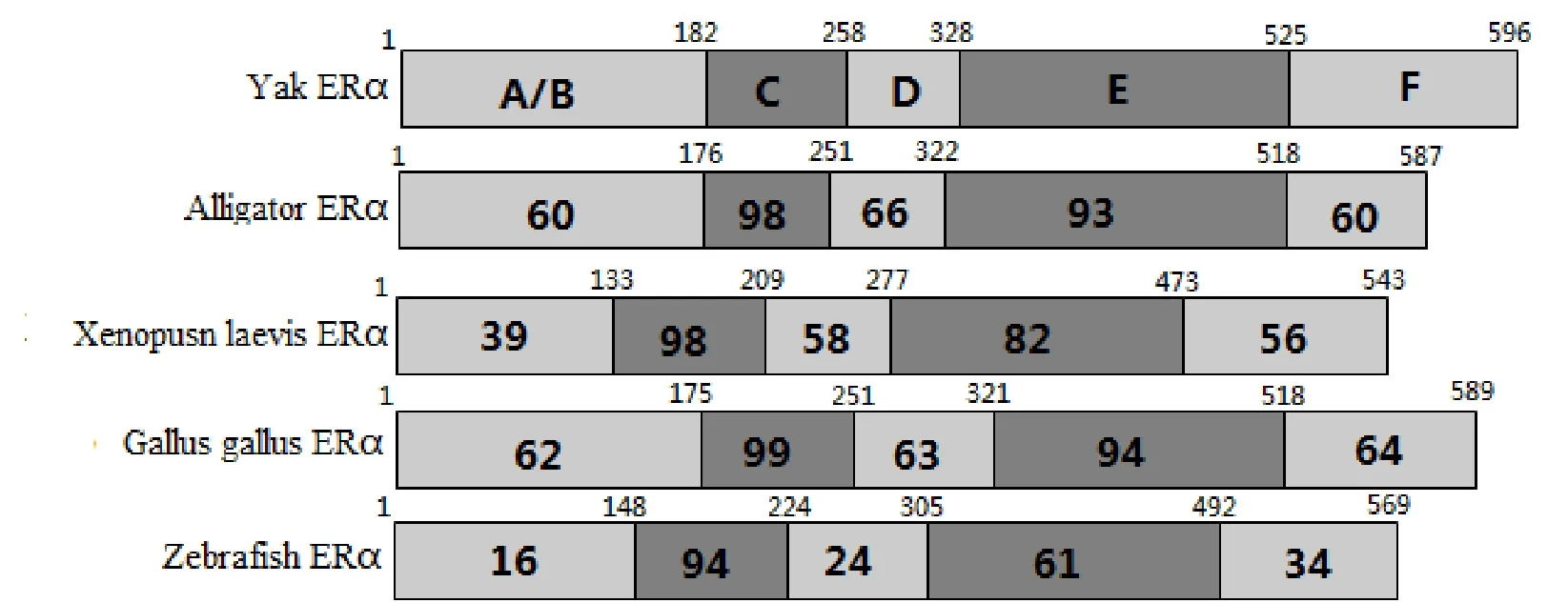
图3 牦牛ERα结构域与 鳄鱼、蟾蜍、原鸡及斑马鱼的比较(方框里的数字代表ERα相对应结构域的相似性)Fig.3 Domain structure of the yak ERα, and identity with alligator, xenopus laevis, gallus gallus, zebrafish ERα.The numbers within each box indicated the percentage identity of the domain relative to the yak ERα.

图4 牦牛ERβ结构域与 鳄鱼, 蟾蜍, 原鸡, 斑马鱼的比较(方框里的数字代表ERβ相对应结构域的相似性)Fig.4 Domain structure of the yak ERβ, and identity with alligator, xenopus laevis, gallus gallus, zebrafish ERβ.The numbers within each box indicated the percentage identity of the domain relative to the yak ERβ.
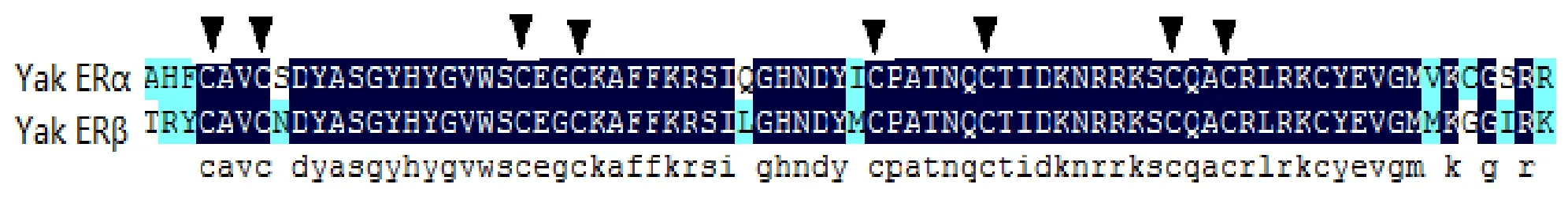
图5 牦牛ERα和ERβ中八个半胱氨酸的位置Fig.5 The position of eight cysteine of yak ERα and ERβ
2.2 进化树分析
根据不同物种的ERα和ERβ的氨基酸序列, 用MEGA5.0软件, 用NJ法构制系统进化树如(图6)所示.由图可见, ERα和ERβ各聚成一支.
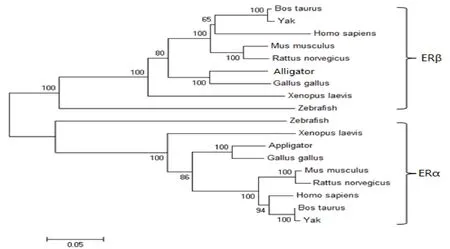
图6 牦牛ERα和ERβ蛋白质进化树分析(NJ法)Fig.6 Phylogenetic tree of the deduced amino acid of ERα and ERβ
2.3 麦洼牦牛ERα基因和ERβ基因的组织表达谱
以GADPH为内参基因, 采用定光定量PCR技术对牦牛ERα和ERβ基因在牦牛心、肝、脾、肺、肾、胃、乳腺、睾丸、输卵管和子宫组织中的表达情况进行了研究.结果表明, ERα和ERβ两种可变剪切体在牦牛各组织中广泛表达, 但表达在各组织中存在较大差异(图7).ERα在牦牛乳腺中表达最高, 子宫、输卵管及卵巢次之,心、肝、脾、肺、肾及睾丸相对表达较低.牦牛ERβ基因在卵巢中表达最高, 子宫和输卵管次之, 而心、肝、脾、肺、肾、睾丸及乳腺表达较低.就牦牛同一组织中ERα和ERβ而言, 牦牛ERα的mRNA的表达水平普遍高于ERβ, 除了睾丸.
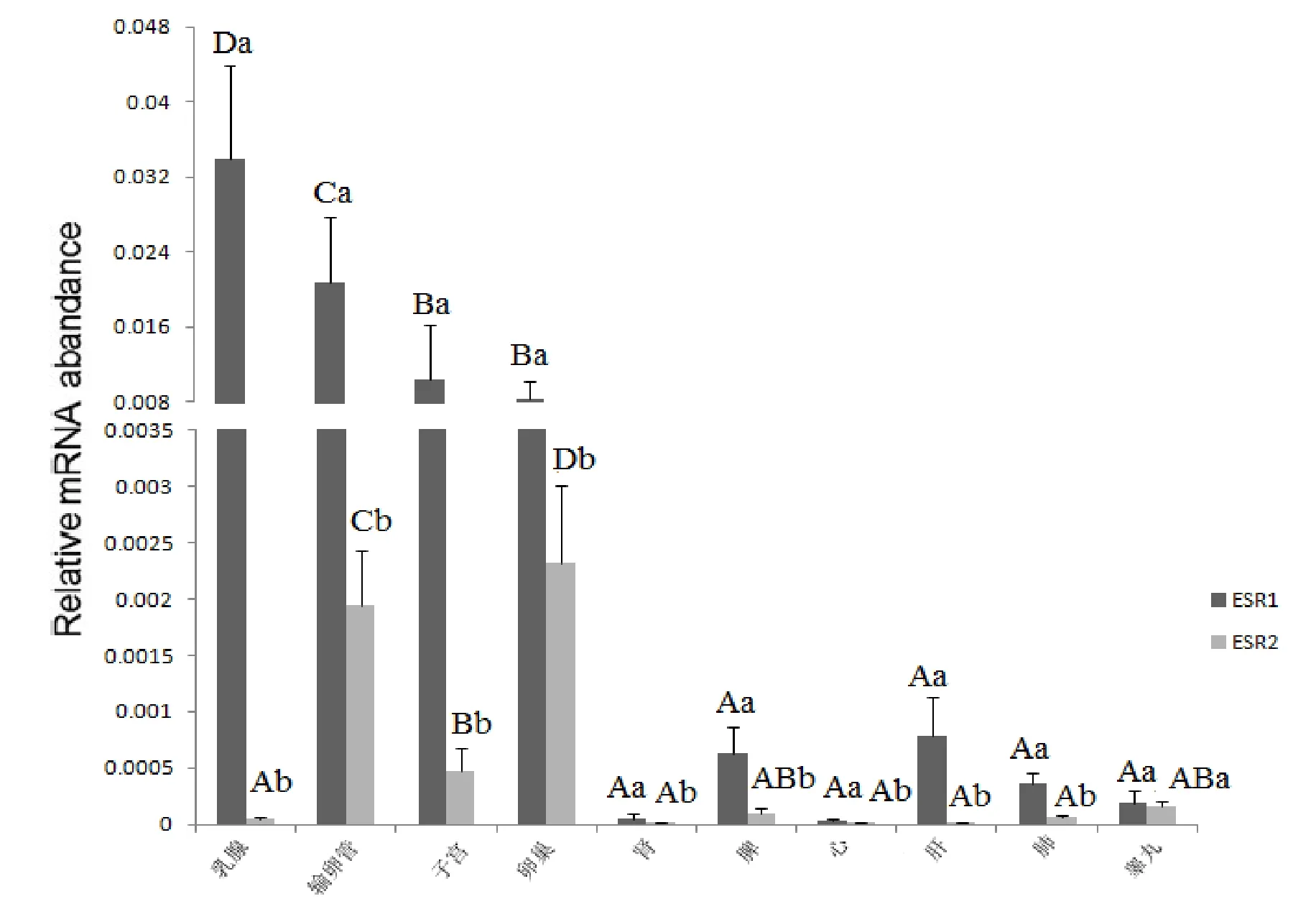
图7 Rα和ERβ基因在麦洼牦牛不同组织中的荧光定量表达Fig.7 The realtime quantification of ERα and ERβ gene expression in various tissues in MaiWa yak
3 讨论
雌激素是人类及其他动物体内最重要的激素之一, 具有广泛的生理功能.而雌激素受体是介导其实现功能的受体[19].本研究首次克隆得到牦牛ERα基因cDNA共2100bp, 其中ORF序列1791bp, 理论编码596个氨基酸.另外, 获得牦牛ERβ基因cDNA共1 658 bp, 其中ORF序列1 584 bp, 编码个527氨基酸.系统进化树分析表明, 牦牛ERα是雌激素受体α集群的成员, 而牦牛ERβ属于ERβ集群.与此同时, 牦牛ERα和牦牛ERβ氨基酸同源性与其他物种具有很高的相似性, 说明了ERα和ERβ基因在进化上具有很高的保守性.目前通过缺失突变与点突变技术已证明ERα和ERβ基因从编码区的5' 端到3' 端可分为6个功能区: N末端的A/B区, 具有转录激活作用; C区含有两个II型锌指结构, 与DNA结合有关; D区为绞链区, 蛋白质在此改变构象; E区是激素结合区; F区的特异性功能域还未有人研究[20].ERα与ERβ结构域的比对结果表明, ERα和ERβ的A/B结构域长度存在很大的差异(ERα为182个氨基酸, ERβ为120个氨基酸), 这与Filby等对其他物种的报道相似[21].在A/B区有一个非配基依赖性转录激活功能区AF1, 基础转录因子或其他转录因子与其相互作用活化目的基因[19].而有的研究表明ERβ可能缺少AF1.A/B区域的长度不规则的高度可变, 调节ERs与DNA的相互作用.在ERα和ERβ中, 8个半胱氨酸残基的分布是相同的, 都由两个锌指结构(C I和C II)组成, 这些结构是与目标基因相结合所必须的.C I的功能是确定结合的特异性, 控制ERs对靶基因的专一性选择, C I的作用是由锌指根部C端侧Cys邻近的3个保守性残基, 称为P-盒(P-box)实现的.C II的功能是提供非专一性选择, C II的功能是有其基部的被称为D-P盒(D-box)的5个氨基酸残基实现的.此外, 牦牛的ERs基因的C(C domain: 94-99%)和E(E domain: 58-94%)结构域从鱼类到哺乳类都具有很高的相似性, 这与前人的研究结果一致[22].C结构域和E结构域是雌激素的核心结构域, 且是雌激素功能发挥必不可少的部分.从鱼类到哺乳类, 雌激素的主要功能都未发生变化, 因此雌激素基因在进化上保持了相对高的保守性.与其他结构域相比较, D结构域的相似性要低一些, D区可与热休克蛋白(hotskock protdn, Hsp)结合, 使ERs基因可适当折叠, 从而保护疏水的配基结合区(ligandblnding domain, LBD).
此外, 本研究结果表明, 两种雌激素受体ERα和ERβ在牦牛的各种组织中均有表达.说明雌激素可能参与了动物生殖系统、神经系统、消化系统、骨骼及内分泌系统等多种系统的基本生理活动.雌激素受体在输卵管、子宫、乳腺及卵巢等生殖器官中高表达, 在心、肝、脾、肺、肾及睾丸中也可检测到表达.这说明雌激素的主要作用还是调控生殖系统的生长与发育.mRNA可变剪接在很大程度上增加了真核生物基因表达的复杂程度并相应的增加了蛋白质功能的多样性[23].实验结果还显示, 在子宫、乳腺、卵巢及输卵管中ERα的表达高于ERβ.这与前人的研究结果有很多相似之处.WEIHUA[24]等报道, 子宫中雌激素是通过ERα和ERβ共同作用的.在未成熟子宫中, ERβ对ERα起着调节的作用.Hiroi[25]等研究表明, 在大鼠子宫中, 均可检测到ERα和ERβ的mRNA的表达, 但ERα的表达占优势.Couse[26]等指出, ERα基因敲除小鼠表现为不孕, 而ERβ基因敲除小鼠可孕, 但受孕的胎数显著减少.因此Couse等猜想ERα和ERβ共同参与调节子宫的发育, 而ERβ的表达量可能与动物的繁殖率有关.根据本研究得出的结果, 笔者推测, 两种雌激素受体在动物体内的同一器官可能发挥着不同的作用.ERα对生殖、乳腺的发育和泌乳起重要作用, 因此在这些组织中, ERα的表达占优势, ERβ基因可能与排卵有重要的关系而与泌乳和生殖没有很大关系.牦牛与其他牛相比, 表现为低繁, 但是否与子宫中ERβ的低表达有关系是下一步拟研究的问题.综上所述, 本研究首次克隆了牦牛ERα和ERβ基因, 并发现ERα和ERβ基因在牦牛多种组织中均有不同程度的表达, 且在生殖器官中优势表达; 同时牦牛ERα和ERβ基因mRNA在多种组织中都存在两种类型的可变性剪切.该研究结果为进一步探究牦牛的特殊繁殖机理提供了基础.
[1]熊显荣, 高川, 符梅, 等.供体细胞来源和TSA处理对牦牛iSCNT胚胎重编程的影响[J].西北农林科技大学: 自然科学版, 2012, 40(10): 9-14.
[2]丁利军, 吴井生, 王建武, 等.猪雌激素受体α基因E区部分cDNA克隆与原核表达[J].畜牧兽医学报, 2006, 37(7): 635-639.
[3]WANG P H.Role of sex hormone receptors in ovulation[J].Taiwanese J Obstet Gynecol, 2005, 44: 16-25.
[4]董文艳, 陈阿琴, 王争光, 等.湖羊高繁殖力候选基因ESR的研究[J].浙江农业学报, 2009, 21(6): 561-564.
[5]尹玉涛, 王树迎, 刘宵, 等.出生后发育时期济宁青山羊乳腺中ERα和PR的分布及其mRNA表达[J]. 畜牧兽医学报, 2012, 43(12): 1984-1990.
[6]白淑, 同海妮, 石运芝, 等.济宁青山羊子宫内ERα、ERβ和PR的分布及其mRNA表达的增龄性变化[J].畜牧兽医学报, 2013, 44(10): 1539-1546.
[7]PAUL J S, MALCOLM V L, PAUL J S, et al.Comparative distribution of estrogen receptor(ERα) and (ERβ) mRNA in the rat pituitary, gonad and reproductive tract[J].Steroids, 1998, 63: 498-504.
[8]SAKAGUCHI H, FUJIMOTO J, AOKI I, et al.Expression of estrogen receptor alpha and beta in myometrium of premenopausal and postmenopausal women[J].Steroids, 2003, 68: 11-19.
[9]COSKUN T, SEVINE A, TEVETOGLU I, et al.Delayed gastric emptying in conscious male rats following chronic estrogen and progesterone treatment[J].Res Exp Med, 1995, 195: 49-54.
[10]GREEN S, WALTER P, KUMER V, et al.Human oestrogen receptor cDNA: sequence expression and homology to v-erb-A[J].Nature, 1986, 320: 134–139.
[11]MOSSELMAN S, POLMAN J, DIJKEMA R.ERβ: identification and characterization of a novel human estrogen receptor[J].FEBS Letter, 1996, 392: 49-53.
[12]KRUST A, GREEN S, ARGOS P, et al.The chicken oestrogen receptor sequence: homology with v-erbA and the human oestrogen and glucocoticoid receptors[J].EMBO J, 1986, 5: 891-897.
[13]SUMIDA K, OOE N, SAITO K, et al.Molecular cloning and characterization of reptilian estrogen receptor cDNAs [J].Mol Cell Endocrinol, 2001, 183: 33-39.
[14]WEILER I J, LEW D, SHAPIRO D J.The Xenopus laevis estrogen receptor: sequence identity with human and avian receptors and identification of multiple estrogen receptor messenger ribonucleic acids [J].Mol Endocrinol, 1987, 1:355-362.
[15]KLEIN S L, STRAUSBERG R L, WAGNER L.Genetic and genomic tools for Xenopus research: The NIH Xenopus initiative [J].Dev Dyn, 225(4): 384-391.
[16]PAKDEL F, LEGAC F, LEGOFF P, et al.Full-length sequence and in vitro expression of rainbow trout estrogen receptor cDNA [J].Mol Cell Endocrinol, 1990, 71: 195-204.
[17]CHANG X T, KOBAYASHI T, TODA T, et al.Molecular cloning of estrogen receptors alpha and beta in the ovary of a teleost fish, the tilapia (Oreochromis niloticus) [J].Zool Sci, 1999, 16: 653–658.
[18]LIVAK K J, SCHMITTGEN T D.Analysis of relative gene expression data using real-time quantitative PCR and the 2 −ΔΔCT method [J].Methods, 2001, 25: 402-408.
[19]赵晓民, 徐小明.雌激素受体及其作用机制[J].西北农林科技大学: 自然科学版, 2004, 32(12): 154-158.
[20]TAKASHI T, SHINJI A, KOHEI Y.Molecular cloning and characterization of Japanese eel estrogen receptor cDNA [J].Molecular and Cellular Endocrinology, 1996, 119: 37-45.
[21]FILBY A L, Tyler C R.Molecular characterization of Estrogen receptors 1, 2a, and 2b and their tissue and ontogenic expression profiles in fathead minnow (Pimephales promelas) [J].Biology of reproduction , 2005, 73: 648-662.
[22]YOSHINAO K, ENA T, HIROSHI U, et al.Molecular cloning and characterization of ligand-and species-specificity of amphibian estrogen receptors [J].General and comparative endocrinology, 2010, 168:220-230.
[23]BLENCOWE B J.Alternative splicing: new insights from global nanlyses [J].Cell, 2006, 126(1): 37-47.
[24]WEIHUA Z, SAJI S, MKINEN S, et al.Estrogen receptor(ER) beta, a modulator of ER alpha in the uterus [J].Proc Natl Acad Sci USA, 2000, 97(11): 5936-5941.
[25]HIROI H, INOUE S, WATANABE T, et al.Differential immunolocalization of estrogen receptor α and β in rat ovary and uterus [J].J Mol Endocrinol, 1999, 22(1): 37-44.
[26]COUSE J F, KORACH K S.Contrasting phenotypes in reproductive tissues of female estrogen receptor null mice [J].Ann N Y Acad Sci, 2001, 948(1): 1-8.
Astract:The objective of this study is to explore the molecular characteristics and expression pattern of ERα and ERβ gene in yak(Bos grunniens).A pair of special primes were designed according to released sequence of bovine ERα and ERβ in GenBank.Then reverse transcription-polymerse chain reation (RT-PCR) was used to clone ERα and ERβ genes of MaiWa yak, and Real-time PCR was used to identify the mRNA expression of ERα and ERβ.The result showed that MaiWa yak ERα gene cDNA was 2 100 bp, with an ORF of 1 658 bp encoding 596 amino acids. ERβ gene cDNA was 1 791 bp, with an ORF of 1 584 bp encoding 527 amino acids. The sequence multialign results showed that yak ERα and ERβ gene shared 45.3%-99.5% and 53.9%-99.1% of similar amino sequence, with that of Bos Taurus, gallus gallus, Homo sapiens, Mus musculus, Rayyus norvegicus, alligator, xenopus laevis, and zebrafish. Real-time PCR analysis revealed that the ERα and ERβ were expressed in a variety of tissues, but the expression of ERα was higher than ERβ in productive organs. Additionally, as far as ERα, the mRNA expression was the highest in mammary gland, followed by uterus, oviduct and ovary, and lowest in liver, kidney, lung, testis, spleen and heart. The ERβ mRNA level was highest in ovary, intermediary in uterus and oviduct, and lowest in heart, liver, spleen, lung, kidney, mammary gland and testis. Identification and tissue distribution of yak ERα and ERβ genes provided foundation for the further study on their biological roles in yak.
Molecular characterization and tissue distribution of ERαand ERβ genes in domestic Yak
LI Jian1, FU Mei2, LAN Dao-liang1, XIONG Xian-rong2
(1.Institute of Qinghai-Tibetan Plateau Research, Southwest University for Nationalities, Chengdu 610041, P.R.C.; 2.School of Life Science and Technology, Southwest University for Nationalities, Chengdu 610041, P.R.C.)
yak ; ERα gene; ERβ gene; molecular characterization; gene expression
S814.1, S823
A
1003-4271(2014)06-0801-08
10.3969/j.issn.1003-4271.2014.06.01
2014-10-10
李键(1967-), 男, 藏族, 四川理县人, 教授, 博士, 研究方向: 动物生殖生理与胚胎工程; E-mail: lijian@swun.cn.
国家科技支撑计划课题(2012BAD13B06).
