Hsp70核苷酸结合域突变体影响酶活的分子动力学模拟研究
2013-11-14周小红徐利楠薛友林宋有涛
周小红,徐利楠,薛友林,宋有涛,2*
(1.辽宁大学生命科学院,辽宁沈阳110036;2.辽宁大学环境学院,辽宁沈阳110036;3.辽宁大学轻型产业学院,辽宁沈阳110036)
Hsp70家族是细胞内一类高度保守的分子伴侣蛋白,可以与其它分子伴侣共同参与多种重要的生命活动,如非天然多肽的折叠、多聚体的组装、细胞器蛋白的转运及错误折叠蛋白的降解等[1]。所有的Hsp70家族都由一个氮端44 kDa的核苷酸结合域(NBD,包括 NBD-I,NBD-II)和一个碳端25 kDa的底物结合域(SBD,Substrate-binding domain,包括SBD-α,SBD-β)组成[2]。
此前,McKay等人的研究发现牛Hsc70(Heat shock cognate 70 protein)的ATP酶活性与金属离子及活性位点 T13、T204的自身磷酸化有重要关系[3],其中,T13侧链的羟基对 ATP酶活性具有重要作用[4]。此外,Chiappori等人研究指出,loop1的柔性对ATP酶活性具有重要作用,柔性越强ATP酶活性越高[5]。这些结果暗示着loop1的构象改变可能引起Hsp70氮端ATP酶活的变化。随后,Jones等人的研究指出酵母Hsp70氮端NBD存在大量影响ATP酶活性的位点[6-7],Chang等人在 DnaK(大肠杆菌Hsp70)中得出相同结果[8]。但是,Jones和Chang等人都没有从分子水平去探讨loop1的构象改变和ATP酶活的关系。
为了从分子水平上探讨loop1构象的改变与ATP酶活的关系,在已有研究表明同源的突变体功能相似的基础上[9],我们选用同源性很高的DnaK进行动力学模拟(酵母Hsp70晶体结构未知),研究了点突变可能通过loop1的构象改变而引起ATP酶活变化——酵母 Hsp70氮端 NBD中 A17V(DnaKA17V)、R23H(DnaKT23H)、G32S(DnaKG32S)增强ATP酶活、R167H(DnaKR167H)对 ATP酶活无影响[6]。该研究不仅是对Jones等人生化试验数据的分子解释,而且对后续Hsp70氮端NBD的点突变研究具有重要的借鉴意义。
1 实验过程
1.1 蛋白质的来源及模型构建
酵母Hsp70氮端NBD初始三维结构模型来自同源蛋白 E.coli DnaK(RCSB数据库,PDB编号1DKG:D)[10]。将已知的野生型DnaK NBD晶体结构模型与对应的17位、23位、32位、167位突变体一级序列提交至 Swiss Model服务器上(http://swissmodel.Expasy.org)[11],根据同源建模算法得到所有突变型蛋白的三维结构,将该模型作为后续动力学模拟突变型A17V、R23H、G32S、R167H NBD的初始模型。
1.2 动力学模拟过程
本模拟利用由动力学模拟软件Gromacs 4.5.5[12]和三维结构分析软件 PyMOL 进行。各模型的模拟步骤及参数设定均相同。具体步骤及参数如下:
(1)力场选择:根据不同力场偏向于不同的结构状态模拟,该模拟各体系均采用NPT系综,计算力场选用GROMOS96 43a1。
(2)溶解蛋白:将结构模型放进一个立方体的周期性边界盒子中并置于该盒子的中心,设定模型中任一原子距盒子边缘的距离大于1.0 nm,以保证其不会和另一个盒子中的镜像相互作用。水溶液环境采用SPC模型水分子(约含25 169个水分子)。
(3)平衡电荷:正常的生理条件下,体系的pH值应接近中性,总电荷为0。向各溶液体系中加入不同数量的Na+或Cl-,保持体系 pH=7,总电荷为0。
(4)能量最小化:为了消除可能的原子碰撞,根据蛋白质折叠的热力学,先将各模型在真空中以最陡下降法(steep)优化2 000步的能量使其达到结构稳定状态。
(5)限制性模拟:根据固定主链,优化侧链的原则,为了使侧链找到一个较稳定的状态避免出现溶剂分子分布不均的问题,进行了80 ps的限制性模拟。
(6)最终的动力学模拟:达到上述平衡之后,各体系在恒定的温度和压力下进行10 ns的分子动力学模拟。分子的运动轨迹每4 ps收集一次,以便用于后续的数据分析。
此外,为了保持整个模拟过程中体系温度稳定在300 K、压力稳定在 1.0×105Pa,分别采用“V-rescale”和“Parrinello-Rahman”方法对体系温度和压力进行控制,耦合常数分别为0.1 ps和0.5 ps。模拟的时间积分步长为2fs,整个体系使用周期性边界条件,采用LINCS(linear constraint solver)算法对体系中所有键的键长进行约束,静电相互作用采用PME(particle mesh Ewald)算法进行估算,截断半径为1.0 nm。范德华力通过“cut-off”方法进行估算,截断半径为1.0 nm。
2 结果与讨论
2.1 结构稳定性
RMSD(Root mean square deviation,均方根偏差)可用来测量进行比对的两个蛋白质相同位点的氨基酸残基碳原子之间的距离,是衡量模拟过程中蛋白质稳定程度的重要参数,间接反应蛋白质灵活性。通过比较整体RMSD曲线的变化趋势得出在7 ns时各模型均达到平衡状态(见图1a),结构稳定。对loop1稳定性分析发现,各模型在7 ns时达到平衡(见图1b),平均值分别为:WT(0.51 nm)、R167H(0.69 nm)、G32S(0.81 nm)、A17V(0.84 nm)、T23H(0.75 nm)。从这些数据可以明显看出突变模型A17V、T23H、G32S中loop1的RMSD平均值均比WT、R167H高。表明突变模型 A17V、T23H、G32S的柔性比 WT、R167H 强,暗示 A17、T23、G32三个位点可能通过loop1构象变化,引起酵母Hsp70氮端NBD的ATP酶活性变化。
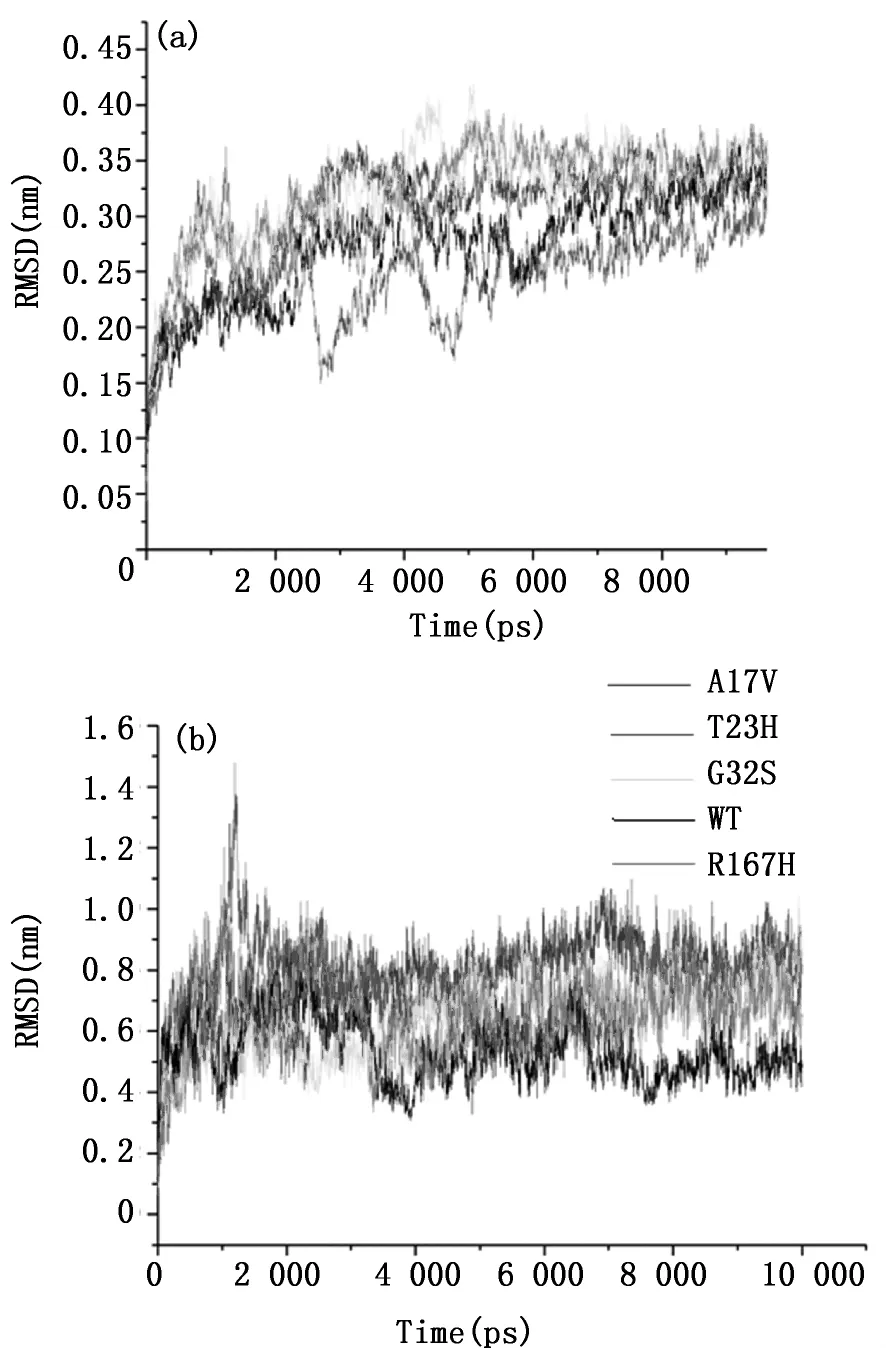
图1 (a)蛋白RMSD随时间变化(b)loop1RMSD随时间变化Fig.1 (a)The protein RMSD as a function of simulation time(b)The loop1 RMSD as a function of simulation time
2.2 Hsp70氮端ATP酶活性区域结构对比
通过三维可视软件PyMOL,标示出loop1和重要残基T11在E.coli DnaK氮端NBD的位置(见图2)。

图2 DnaK NBD三维结构Fig.2 DnaK NBD three-dimensional structural
为进一步分析loop1怎样的构象改变而引起Hsp70氮端NBD的ATP酶活性变化,我们对A17V、T23H、G32S、R167H、WT 进行了 10 ns时整体蛋白三维结构对比分析。结果显示,突变模型R167H与WT在loop1处具有相似的构象,重要残基T11侧链未向内发生移动(见图 3a)。突变模型 A17V、T23H、G32S与WT结构对比发现,T11的侧链均向内发生不同程度移动(见图3b,图c,图d),预示着在酵母Hsp70 ATP水解过程中,A17V、T23H、G32S三个突变模型与WT和R167H相比能够更快速的通过T11侧链的羟基与ATP进行相互作用,攻击ATPγ-磷酸基团,引起 ATP快速水解,从而增强ATP酶活性。

图3 模拟后loop1结构对比:WT(T11:Sticks)(a)R167H(T11:lines)(b)A17V(T11:lines)(c)T23H(T11:lines)(d)G32S(T11:lines)Fig.3 loop1 structural alignment after MD simulation:WT(T11:Sticks)(a)R167H(T11:lines)(b)A17V(T11:lines)(c)T23H(T11:lines)(d)G32S(T11:lines)
2.3 Hsp70 ATP酶活性区域距离分析
为了深入探讨前面提到的构象改变与功能变化关系,我们引入了一个ATP分子(RCSB数据库,PDB编号:3LDL)进行距离分析。McKay等人研究指出,Hsc70 T13(DnaKT11)残基对ATP水解的重要作用,是通过T13侧链的羟基对ATPγ-磷酸基团作用,引起ATP酶水解速率变化[5]。数据显示,10 ns时T11侧链的羟基与γ-磷酸基团距离(见图4a,b,c,d,e)WT(5.5 Å),R167H(6.0 Å)均大于突变模型 A17V(1.3 Å)、T23H(4.9 Å) 和 G32S(4.6 Å)。数据表明,T11侧链的羟基与γ-磷酸基团距离变近,其相互作用增强,可能引起ATP酶活性增强,与前面结论相一致。从分子水平初步解释了生化试验酵母Hsp70 A17V,R23H,G32S点突变增强ATP酶活结论。
2.4 loop1 氢键分析
为了探讨引起loop1发生向内移动的原因,我们对loop1与周围氨基酸的氢键交互作用进行了分析,通过计算得到各突变体模型10ns模拟过程中氢键数量的平均值分别为:WT(1.2个)、R167H(1.2个)、A17V(2.6 个)、T23H(2.4 个)、G32S(0.4个)。结果表明,A17V、T23H突变体模型在10ns模拟过程中的氢键数量显著高于WT、R167H,然而有趣的是G32S突变体模型在10ns模拟过程中的氢键数量低于WT、R167H(见图5a)。随后氢键存活时间分析发现(见图5b),在10ns模拟过程中稳定氢键数量分别为WT(1个)、R167H(1个)、A17V(3个)、T23H(3个)、G32S(1个)。以上数据表明A17V、T23H点突变后,引起loop1中与T12相互作用的氢键数量显著增加,增强了loop1附近的氢键网络,从而间接调控loop1中T11侧链向内移动,引起ATP酶活性的增强;值得注意的是,G32S点突变后尽管减少了loop1氢键数量,但是氢键存活时间表明,在没有与T12相互作用的氢键存在时,每隔一段时间会重复出现T11-S38氢键,有规律的影响T11残基的状态,可能直接引起T11侧链向内移动,从而增强ATP酶活性。

图4 模拟后T11残基与γPi距离(单位:Å)(a)WT(b)R167H(c)A17V(d)T23H(e)G32SFig.4 the distance of T11 and γPi after MD simulation(Å)(a)WT(b)R167H(c)A17V(d)T23H(e)G32S

图5 (a)模拟过程中loop1氢键数量(b)模拟过程中loop1氢键存活时间Fig.5 (a)the number of hydrogen bond during the MD simulation(b)Hydrogen bond existence map of loop1 during the MD simulation
3 结论
Hsp70分子伴侣SBD和NBD的功能既相互联系又相互独立,分析其中一个结构域的构象功能变化,能够间接反应出整个Hsp70分子伴侣的功能变化。在本研究中,A17V,T23H,G32S点突变引起Hsp70氮端NBD ATP酶活性区域的ATP结合口袋袋口Loop1柔性增强,重要残基T11侧链向内移动,接近ATPγ-磷酸基团,增强其与ATP的作用,提高ATP水解速率,从而增强ATP酶活性,引起Hsp70分子伴侣功能变化。
本研究为我们从分子水平上解释Jones等人的生化试验结果(A17V,R23H和G32S点增强ATP酶活,R167H与WT对 ATP酶活没有影响[6])提供了数据支持。同时也从分子水平对 Hsc70 T13(DnaKT11)侧链的羟基对ATP酶具有重要作用[4]进行了一定验证,对后续Hsp70氮端NBD的突变研究具有重要的借鉴意义。
References)
[1] Sharma D,Masison DC.Hsp70 Structure,Function,Regulation and Influence on Yeast Prions[J].Protein Peptide Letters,2009,16(6):571-581.
[3] Johnson ER,McKay DB.Mapping the role of active site residues for transducing an ATP-induced conformational change in the bovine 70-kDa heat shock cognate protein[J].Biochemistry,1999,38(33):10823-10830.
[4] Sousa MC,McKay DB.The hydroxyl of threonine 13 of the bovine 70-kDa heat shock cognate protein is essential for transducing the ATP-induced conformational change[J].Biochemistry,1998,37(44):15392-15399.
[5] Chiappori F,Merelli I,Colombo G,et al.Molecular Mechanism of AllostericCommunication in Hsp70 Revealed by Molecular Dynamics Simulations[J].PLoS computational biology,2012,8(12):e1002844.
[6] Jones GW,Masison DC.Saccharomyces cerevisiae Hsp70 Mutations Affect[PSI+]Prion Propagation and Cell Growth Differently and Implicate Hsp40 and Tetratricopeptide Repeat Cochaperones in Impairment of[PSI+][J].Genetics,2003,163(2):495-506.
[7] Loovers HM,Guinan E,Jones GW.Importance of the Hsp70 ATPase Domain in Yeast Prion Propagation[J].Genetics,2007,175(2):621-630.
[8] Chang L,Thompson AD,Ung P,et al.Mutagenesis reveals the complex relationships between ATPase rate and the chaperone activities of Escherichia coli heat shock protein 70(Hsp70/DnaK)[J].Journal of Biological Chemistry,2010,285(28):21282-21291.
[9] Xu L,Hasin N,Shen M,et al.Using Steered Molecular Dynamics to Predict and Assess Hsp70 Substrate-Binding Domain Mutants that Alter Prion Propagation[J].PLoS computational biology,2013,9(1):e1002896.
[10] Harrison CJ,Hayer-Hartl M,Di Liberto M,et al.Crystal structure of the nucleotide exchange factor GrpE bound to the ATPase domain of the molecular chaperone DnaK[J].Science,1997,276(5311):431-435.
[11] Kiefer F,Arnold K,Künzli M,et al.The SWISS-MODEL Repository and associated resources[J].Nucleic acids research,2009,37(suppl 1):D387-D392.
[12] Pronk S,Páll S,Schulz R,et al.GROMACS 4.5:a highthroughput and highly parallel open source molecular simulation toolkit[J].Bioinformatics,2013,29(7):845-854.
