基于双功能荧光标签8-氨基1,3,6-萘三磺酸的N-连接糖基化富集方法
2013-10-09乔晓强王蕊马宝伟
乔晓强,王蕊,马宝伟
(河北大学药学院,河北省药物质量研究重点实验室,河北保定 071002)
糖基化是蛋白质最重要的翻译后修饰之一,与生物体中多种的分子细胞机制,包括发展、生长、通信、特别是癌症的发生息息相关[1].许多糖基化蛋白质已被成功用于医学的治疗和诊断,例如her2/neu是乳腺癌的生物标记物;CA125是卵巢癌的标记物;前列腺特异抗原是前列腺癌的标记物[2-3].因此,对蛋白质糖基化的研究具有重要的意义.
然而,许多具有重要生物学功能的糖基化蛋白质往往具有低丰度,如果进一步采用蛋白酶酶切,糖基化肽段的比例仅占到总肽段数目的2%~5%[4],大大过量的非糖基化肽段会严重干扰后续的质谱(MS)鉴定.因此,发展高效、高选择性的富集技术已成为糖基化蛋白质研究必不可少的要务之一.近年来,包括亲水作用色谱法[5-6]、凝集素亲和色谱法[7-8]、硼亲和色谱法[9-10]以及肼化学法[11-12]在内的多种技术得到发展,并成功地实现了糖基化蛋白质/肽段的选择性富集[13].然而,蛋白质糖基化的复杂性和异质性本质使得目前发展的技术均不能实现复杂生物样品中所有糖基化蛋白质的富集[14-16].因此,发展新颖的糖基化富集技术对于深入的糖基化蛋白质组学研究具有重要的意义.
荧光检测具有灵敏度高、选择性好等特点,广泛应用于蛋白质分析[17].在糖基化蛋白质研究中,为了增强糖基化蛋白质释放的低聚糖的分析,通常需要对低聚糖进行荧光衍生后,再进行MS或光学分析[18].常用的荧光试剂包括8-氨基萘-1,3,6-三磺酸二钠盐(ANTS)和氨基芘-1,3,6-三磺酸(APTS)等,据笔者所知,该类试剂尚未应用于糖基化肽段的选择性富集研究.
本文发展了基于荧光标签ANTS的糖基化蛋白质选择性衍生和二氧化钛(TiO2)亲和选择性富集的N-连接糖基化富集策略(ANTS-TiO2),并将其用于标准糖蛋白质混合物和人血清样品中N-连接糖基化肽段的富集.与TiO2法直接富集和肼化学富集法的结果进行比较,实现了互补的N-连接糖基化肽段鉴定.
1 实验部分
1.1 材料与试剂
ANTS,马细胞色素c,马肌红蛋白,牛核糖核酸酶b,鸡抗生素蛋白,鸡伴清蛋白,人α-1-抗胰蛋白酶以及TPCK处理的胰蛋白酶均购自Sigma(St.Louis,MO,USA).TiO2购自GL(Tokyo,Japan).二硫苏糖醇(DTT)、碘代乙酰胺(IAA)和三氟乙酸(TFA)均购于Acros Organics(Geel,Belgium).肽N-糖苷酶F(PNGase F)购自New England Biolabs(Ipswich,MA).碱性磷酸酶(1U/μL)购自上海生工生物工程有限公司.色谱纯乙腈(ACN)购自Merck(Darmstadt,Germany).所有无机试剂均为分析纯而其他试剂均为色谱纯.实验用水采用Milli-Q系统(Millipore,Bedford,USA)纯化制得.
1.2 蛋白质衍生
蛋白质衍生过程如下.首先将20μL的人血清与30μL的氧化缓冲液(100mmol/L乙酸钠,150mmol/L氯化钠,pH 5.0)均匀混合,然后向其中加入高碘酸钠使其终浓度为10mmol/L,于室温避光振荡反应30min.反应完毕,向其中加入终浓度为20mmol/L的亚硫酸钠,继续于室温避光振荡反应10min,以中和过量的高碘酸钠.最后,向其中顺序加入100μL 40mg/mL ANTS(100mmol/L乙酸钠,pH 6.0)和20μL新鲜配制的1.0mol/L氰基硼氢化钠溶液,继续于室温振荡反应2h,反应完毕,蛋白质采用C8固相萃取柱除盐、冷冻干燥后待进一步实验.
1.3 蛋白质酶解
在典型的实验过程中,首先将20μL的人血清样品溶解于8mol/L的尿素,蛋白质经DTT还原和IAA烷基化后,样品用100mmol/L的碳酸氢铵(pH 8.0)稀释至尿素浓度低于1mol/L,最后向其中加入胰蛋白酶使得酶与蛋白质的质量比为1∶20,然后于37℃酶解过夜.为了避免磷酸化肽段的共富集,向上述溶液中加入1μL的碱性磷酸后继续于37℃孵育过夜.反应完毕,加入甲酸终止反应.
1.4 N-连接糖基化肽段富集
N-连接糖基化肽段富集分别采用3种方法:ANTS-TiO2法,TiO2法和肼化学法.ANTS-TiO2法富集过程如下:首先将15mg的TiO2微球和1mg肽段酶解产物以及500μL上样缓冲(1mol/L乙醇酸,体积分数5%TFA,体积分数85%ACN)均匀混合,并于室温孵化1h,TiO2微球采用500μL的上样缓冲清洗2次,以去除未结合肽段,而糖基化肽段采用500μL体积分数2%的氨水(pH>11.5)洗脱、除盐、冷冻干燥后重溶于20μL新鲜配制的100mmol/L碳酸氢铵(pH 8.0)溶液.上述样品中加入2.5μL的PNGase F并于37℃孵浴过夜,反相除盐、冷冻干燥后重溶于20μL含体积分数0.1%甲酸水溶液后进样μRPLC-ESI-MS/MS分析.TiO2法富集过程参考ANTS-TiO2富集法,只是实验过程中不加入荧光标签ANTS.肼化学富集法参考文献[12].
1.5 HPLC分析
HPLC分析在由进样阀(Rheodyne,Cotati,CA,USA)、四元脱气机、配备四元梯度单元的智能色谱泵、智能荧光检测器(Jasco,Tokyo,Japan)和C8柱(大连依利特分析仪器有限公司)组成的系统中进行.流动相A为纯水(含体积分数0.1%TFA),流动相B为体积分数95%的ACN(含体积分数0.1%TFA).分离梯度如下(体积分数):0,20%B;25min,80%B;30min,90%B;流速,1.0mL/min.荧光检测激发和发射波长分别为360nm和515nm.
1.6 μRPLC-ESI-MS/MS分析
Paradigm GM4μHPLC(Michrom Bioresources,Auburn,CA,USA)结合了LCQDUO四级离子阱(LCQ-IT MS,Thermo Fisher,San Jose,CA,USA)的系统用于N-连接糖基化肽段的分析.流动相A为体积分数98%水(含体积分数0.1%甲酸),流动相B为体积分数98%的ACN(含体积分数0.1%甲酸).5cm长的C18柱(5μm,20nm)用于标准糖基化肽段的分析,其分离梯度如下(体积分数):0,0B;40min,40%B;45min,80%B;50min,80%B;流速,5.0μL/min.人血清富集糖基化肽段采用15cm长的C18柱分析,其分离梯度如下:0,0B;5min,5%B;105min,40%B,110min,80%B;125min,80%B;流速,5.0μL/min.
LCQ仪器采用正离子模式,加热毛细管温度为150°C,喷雾电压为2.5kV,MS/MS扫描碰撞能量为35%.一级质谱全扫描质量为m/z400~2 000.MS/MS谱图采用数据依赖模式(data-dependent mode),并且针对全扫描中前两个强度最高峰进行子离子碎片扫描.
1.7 数据分析
采用基于SEQUEST算法的BioWorks软件(版本号3.1)对获得的MS/MS数据进行检索,检索参数设置如下:数据库,ipi.HUMAN.V3.31;固定修饰,半胱氨酸残基+57.0215Ku;可变修饰,天冬酰胺脱乙酰化,甲硫氨酸氧化;母离子和碎片离子的质量容忍度分别为2Ku和1Ku;胰蛋白酶完全酶切,允许最大2个漏切位点;数据筛选标准设定为Xcorr≥1.9,2.2,3.75(分别对应于+1,+2和+3价离子),ΔCn≥0.26.
2 结果与讨论
2.1 ANTS-TiO2法N-连接糖基化富集
ANTS-TiO2糖基化富集过程包括6步,如图1所示.1)糖基化蛋白氧化:糖基化蛋白质中多糖的顺式邻二醇经高碘酸氧化成醛基;2)ANTS衍生:氧化醛基与ANTS的氨基反应后再经氰基硼氢化钠还原形成共价的碳氮键;3)胰蛋白酶酶解:蛋白质混合物经胰蛋白酶酶解成肽段;4)糖基化肽段选择性富集:TiO2选择性亲和标记有荧光标签试剂ANTS的糖基化肽段;5)PNGase F处理:N-连接糖基化肽段通过PNGase F酶切后去除糖链;6)分析:去糖肽经μRPLC-ESI-MS/MS进行分离鉴定.
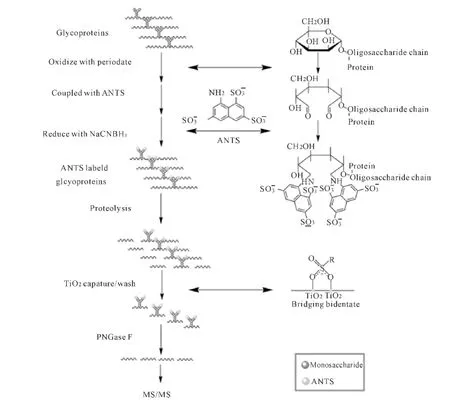
图1 ANTS-TiO2糖基化富集过程示意Fig.1 Schematic diagram of glycosylation enrichment by ANTS-TiO2strategy
2.2 标准蛋白质分析
5种蛋白质,细胞色素c、肌红蛋白、核糖核酸酶b、抗生素和α-1-抗胰蛋白酶经ANTS标记后其荧光光谱如图2所示.细胞色素c和肌红蛋白为典型的非糖基化蛋白质,而核糖核酸酶b、抗生素和α-1-抗胰蛋白酶为典型的糖基化蛋白质.从图2中可以看出所有的糖基化蛋白质经ANTS标记后均发出亮绿色荧光,而非糖基化蛋白质则不能观测到荧光,这主要是由于ANTS的活性氨基和糖基化蛋白质中氧化多糖的醛基反应形成共价化合物从而使得糖基化蛋白质选择性标识有荧光标签,因而发出很强的荧光.上述结果表明,在优化的条件下,ANTS可以实现糖基化蛋白质的选择性标记.
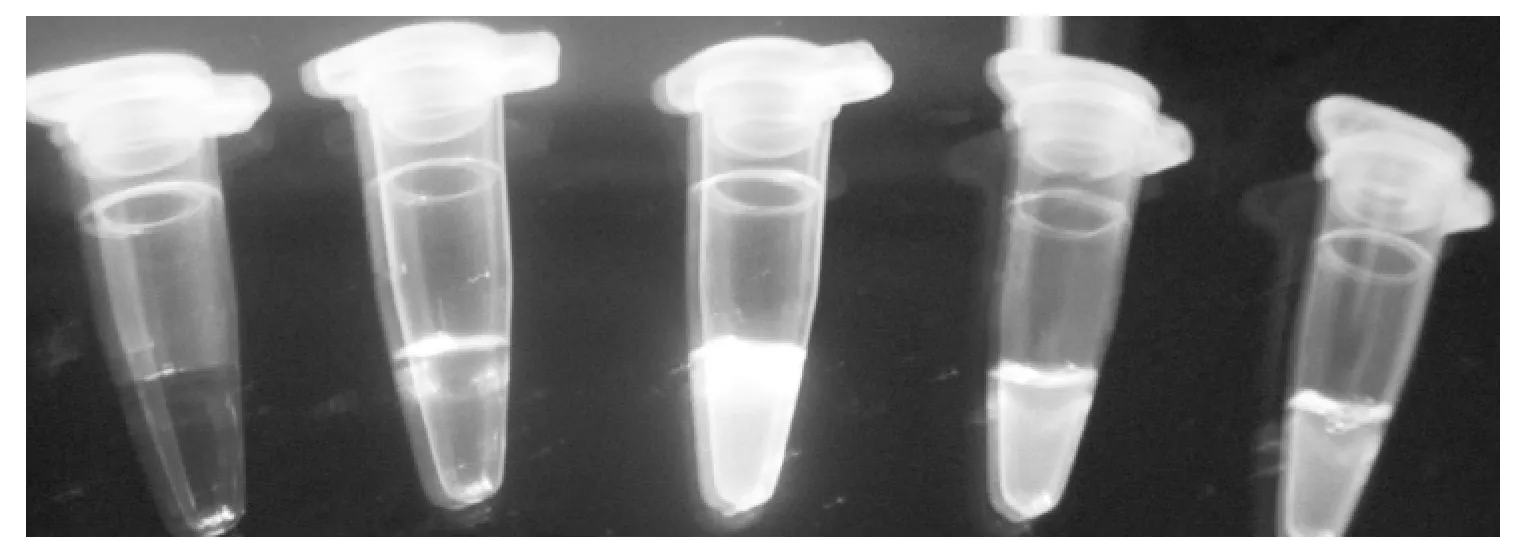
图2 5种蛋白质经ANTS标记后的荧光光谱Fig.2 Fluorescence image of five standard proteins respectively labeled by ANTS
进一步考察了3种糖基化蛋白质的最低检测限,通过结合HPLC分析和荧光检测,核糖核酸酶b、抗生素和α-1-抗胰蛋白酶的检测限分别达到59nmol/L(1.2pmol),105nmol/L(2.1pmol)和2.7μmol/L(54pmol).此外,ANTS衍生的核糖核酸酶b的稳定性结果如图3所示,其衍生物于室温放置1周后其荧光强度仅降低了约10%,显示了ANTS衍生产物具有高稳定性,从而利于后续的富集分析过程.
2.3 蛋白质混合物分析
文献[19]报道TiO2可用于唾液酸糖肽的专一富集.基于此,α-1-抗胰蛋白酶(唾液酸糖蛋白)、抗生素和伴清蛋白(均为中性糖蛋白)组成的糖蛋白质混合物用于评价了发展的ANTS-TiO2富集法.
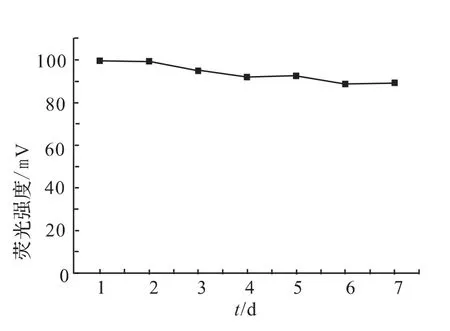
图3 核糖核酸酶b的ANTS衍生物稳定性Fig.3 Stability of ribounuclease b derivatized by ANTS
蛋白质混合物经ANTS-TiO2法处理后鉴定结果如表1所示.抗生素蛋白和伴清蛋白的2条中性糖肽均实现了有效鉴定.α-1-抗胰蛋白酶含有3个已知的N-连接糖基化位点,其中糖基化肽段K.YLGNATAIFFLPDEGK.L成功地实现了鉴定;而另外2个糖基化位点没有得到鉴定,这可能是由于LCQ的低灵敏度造成的.上述结果表明ANTS-TiO2法可以同时实现中性糖肽和唾液酸糖肽的选择性富集.

表1 糖基化蛋白质混合物经LCQ分析鉴定的糖肽Tab.1 Results of LCQ analysis of N-linked glycopeptides isolated from the three glycoprotein mixture
2.4 人血清N-连接糖基化蛋白质分析
进一步采用人血清样品评价了ANTS-TiO2富集法并与TiO2富集法进行了比较.如表2所示,20μL人血清采用TiO2法直接富集共鉴定到35个唯一的N-连接糖基化蛋白质,其中包括53个唯一的N-连接糖基化位点.而上述样品采用ANTS-TiO2法处理后,共鉴定到39个唯一的N-连接糖基化蛋白质,包括了53个唯一的N-连接糖基化位点.进一步比较了糖基化蛋白质和糖基化位点的组成,结合ANTS-TiO2和TiO22种富集方法共鉴定到78个唯一的N-连接糖基化位点,如图4a所示,其中仅有28(35.9%)个位点为2种方法共同所鉴定的,显示这2种方法部分的互补性;其共同鉴定的糖基化蛋白质占42.3%(22/52)(Fig.4b),进一步说明上述2种方法部分的互补性.互补性可能的原因是ANTS-TiO2法针对磺酸化的糖肽,而TiO2法则针对唾液酸糖肽.此外,ANTS-TiO2法的选择性为42.1%,这与TiO2法直接富集的选择性相当(45.2%).

表2 比较ANTS-TiO2法、TiO2法以及肼化学法分析人血清N-连接糖基化蛋白质Tab.2 Comparison of N-linked glycosylation identification by ANTS-TiO2,TiO2,and hydrazide chemistry strategies for human serum proteins
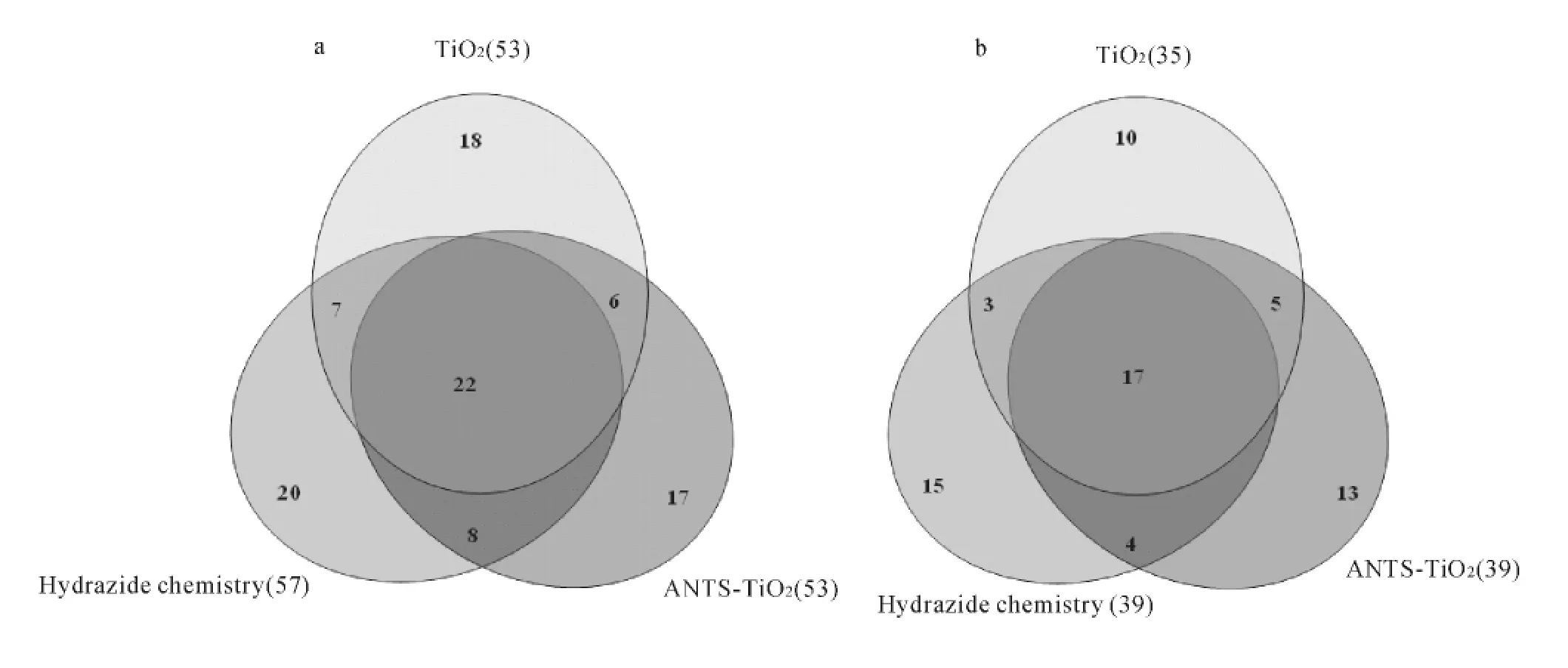
图4 比较ANTS-TiO2法、TiO2法以及肼化学富集法分析人血清糖基化蛋白质.Fig.4 Comparison of ANTS-TiO2,TiO2,and hydrazide chemistry strategies for glycoprotein analysis of human serum.
肼化学法由于具有高效、高选择性等特点而成为广泛采用的技术,目前已应用于多种生物样品的大规模N-连接糖基化分析[20-22].因此,进一步将实验中发展的ANTS-TiO2富集法和传统的肼化学富集法进行比较.尽管其选择性(42.1%)略低于肼化学法(60.8%),但是其共同鉴定的糖基化位点和糖基化蛋白质分别仅为37.5%和36.8%(图4),同样显示了部分的互补性.由于ANTS-TiO2富集法与TiO2直接富集和传统的肼化学富集法均显示了部分的互补性,因此,通过结合多种富集技术,有望实现复杂样品中N-连接糖基化的全面分析.
3 结论
发展了基于双功能团荧光标签ANTS选择性标记和TiO2选择性富集的N-连接糖基化富集新方法.结果表明,采用该方法可同时实现中性和唾液酸糖基化蛋白质的选择性衍生和糖基化肽段的选择性富集.进一步应用于人血清N-连接糖基化分析,该方法与采用TiO2法直接富集以及肼化学法富集鉴定到的交叠糖基化位点均小于40%,说明这一新方法与传统方法在糖基化富集方面具有互补性,有望用于复杂蛋白质样品N-连接糖基化的高效富集.
[1] PARODI A J.Protein glycosylation and its role in protein folding[J].Annual Review of Biochemistry,2000,69:69-93.
[2] DUBE D H,BERTOZZI C R.Glycans in cancer and inflammation-potential for therapeutics and diagnostics[J].Nature Reviews Drug Discovery,2005,4(6):477-488.
[3] SHEPARD H M,LEWIS G D,SARUP J C.Monoclonal antibody therapy of human cancer:taking the HER2protooncogene to the clinic[J].Journal of Clinical Immunology,1991,11(3):117-127.
[4] LI Xin,MA Li,LI Jianjun.Recent developments in the enrichment of glycopeptides for glycoproteomics[J].Analytical Letters,2008,41(2):268-277.
[5] 梁鑫淼.基于亲水作用色谱的寡糖色谱分离新进展[J].色谱,2011,29(3):191-192.
[6] ZHU Jun,WANG Fangjun,CHEN Rui,et al.Centrifugation assisted microreactor enables facile integration of trypsin digestion,hydrophilic interaction chromatography enrichment,and on-column deglycosylation for rapid and sensitive N-glycoproteome analysis[J].Analytical Chemistry,2012,84(11):5146-5153.
[7] 卢庄,王兰,贾伟,等.麦胚凝集素亲和色谱富集糖肽过程中的肽键裂解[J].色谱,2009,27(1):19-23.LU Zhuang,WANG Lan,JIA Wei,et al.Cleavage of amide bond during glycopeptide enrichment using wheat germ agglutinin affinity chromatography[J].Chinese Journal of Chromatography,2009,27(1):19-23.
[8] FANATAB S,HINCAPIE M,HANCOCK W S.Using lectins to harvest the plasma/serum glycoproteome[J].Electrophoresis,2012,33(12):1746-1754.
[9] LI Hengye,WANG Heye,LIU Yunchun,et al.A benzoboroxole-functionalized monolithic column for the selective enrichment and separation of cis-diol containing biomolecules[J].Chemical Communications,2012,48(34):4115-4117.
[10] QU Yanyan,LIU Jianxi,YANG Kaiguang,et al.Boronic acid functionalized core-shell polymer nanoparticles prepared by distillation precipitation polymerization for glycopeptide enrichment[J].Chemistry-A European Journal,2012,18(29):9056-9062.
[11] KIM Y J,ZAIDI AINOUCH Z,GALLIEN S,et al.Mass spectrometry-based detection and quantification of plasma glycoproteins using selective reaction monitoring[J].Nature Protocols,2012,7(5):859-871.
[12] SUN Bingyan,RANISH J A,UTLEG A G,et al.Shotgun glycopeptide capture approach coupled with mass spectrometry for comprehensive glycoproteomics[J].Molecular &Cellular Proteomics,2007,6(1):141-149.
[13] PAN Sheng,CHEN Ru,AEBERSOLD R,et al.Mass spectrometry based glycoproteomics-from a proteomics perspective[J].Molecular &Cellular Proteomics,2010,doi:10.1074/mcp.R110.003251.
[14] CAO Jing,SHEN Chengping,WANG Hong,et al.Identification of N-Glycosylation sites on secreted proteins of human hepatocellular carcinoma cells with a complementary proteomics approach[J].Journal of Proteome Research,2009,8(2):662-672.
[15] LEE A,KOLARICH D,HAYNES P A,et al.Rat liver membrane glycoproteome:enrichment by phase partitioning and glycoprotein capture[J].Journal of Proteome Research,2009,8(2):770-781.
[16] PAN Sheng,WANG Yang,QUINN J F,et al.Identification of glycoproteins in human cerebrospinal fluid with a complementary proteomic approach[J].Journal of Proteome Research,2006,5(10):2769-2779.
[17] QIAO Xiaoqiang,SUN Liangliang,WANG Li,et al.High sensitive protein detection by hollow fiber membrane interface based protein enrichment and in situ fluorescence derivatization[J].Journal of Chromatography B,2011,879(17-18):1439-1443.
[18] 马海宁,华玉娟,屠春燕,等.毛细管电泳法分析藏红花植物细胞多糖中单糖组成[J].色谱,2012,30(3):304-308.MA Haining,HUA Yujuan,TU Chunyan,et al.Analysis of monosaccharides in the saffron corm glycoconjugate by capplilary electrophoresis[J].Chinese Journal of Chromatography,2012,30(3):304-308.
[19] LARSEN M R,JENSEN S S,JAKOBSEN L A,et al.Exploring the sialiome using titanium dioxide chromatography and mass spectrometry[J].Molecular &Cellular Proteomics,2007,6(10):1778-1787.
[20] ZHANG Hui,LI Xiaojun,MARTIN D B,et al.Identification and quantification of N-linked glycoproteins using hydrazide chemistry,stable isotope labeling and mass spectrometry[J].Nature Biotechnology,2003,21(6):660-666.
[21] QIAO Xiaoqiang,TAO Dingyin,QU Yanyan,et al.Large-scale N-glycoproteome map of rat brain tissue:Simultaneous characterization of insoluble and soluble protein fractions[J].Proteomics,2011,11(21):4274-4278.
[22] CHEN Rui,JIANG Xinning,SUN Deguang,et al.Glycoproteomics analysis of human liver tissue by combination of multiple enzyme digestion and hydrazide chemistry[J].Journal of Proteome Research,2009,8(2):651-661.
