模型多肽Trp-Cage 折叠形成机制的研究进展
2013-09-21吴晓敏袁晓辉薛书蕾查岭生王光利张海军
吴晓敏 袁晓辉 薛书蕾 查岭生 王光利 张海军,*
(1淮北师范大学生命科学学院,资源植物生物学安徽省重点实验室,安徽淮北235000;2东北林业大学生物资源生态利用国家地方联合工程实验室,哈尔滨150040;3暨南大学生物医药研究院,广州510632)
1 引 言
蛋白质不仅是生命体的重要结构组成部分,而且也是生物体内多种功能的实现者;1其结构的动力学行为,特别是天然态结构的折叠形成过程对其生物功能的实现是至关重要的.2−5许多疾病都是由蛋白质折叠异常或结构破坏引起的,如老年痴呆症(蛋白质构象变化)、囊性纤维病变(蛋白质不能折叠)、家族性高胆固醇症(蛋白质错误折叠)、家族性淀粉样蛋白症(蛋白质沉淀)等等.6−9因此,理解蛋白质结构折叠机制及其与生物功能之间的相互关系一直是蛋白质科学领域中非常重要的研究内容,并且该研究也受到越来越多不同学科领域研究工作者的高度重视.蛋白质结构折叠转变机制的研究不但可以解释生物体中各种功能的实现等基础理论问题,而且还具有重要的现实意义.10,11它可以为寻找更有效的药物靶标位置,改善药物结构以及提高药物效能等提供重要的理论依据.深入研究蛋白质的折叠与解折叠机制,重构构象跃迁过渡路径,发现折叠过程中的中间态、过渡态,对于理解它的结构特征和折叠动力学行为,特别是认识它天然态结构的折叠与解折叠转变过程具有重要的生物学意义,并且对医学、药学等相关领域都将产生重大影响.
本文首先概述了蛋白质折叠研究目前在实验和理论模拟方面存在的问题,然后以结构典型且可快速折叠的人工设计多肽Trp-cage为例,概述Trpcage的折叠动力学行为分别在实验和理论模拟计算方面的研究进展,并对其折叠过渡温度、折叠形成模型及其肽链上关键氨基酸残基在折叠过程中的作用三个方面进行了详细讨论与总结,最后就如何有效化解蛋白质残基间相互作用网络进而降低其折叠机制的复杂性提出了一些新的建议.
2 蛋白质折叠机制研究中存在的问题
2.1 折叠机制实验研究方面
目前适用于生物大分子结构研究的生物物理实验技术(如核磁共振、X射线衍射、电子衍射、单分子荧光技术、时间分辨荧光技术、氢氘交换质谱技术和温度跳跃技术等)发展迅猛,可以通过实验解析构建原子分辨率水平的三维结构模型,也能够清楚地测定蛋白质构象变化及其折叠动力学等特征,但缺点是现有实验手段测定的动力学结构特征也仅是时间和空间上的平均结果,难以真正反映出蛋白质的折叠转变的微观细节过程以及与功能实现紧密相关的动力学过程.12,13尽管目前用于测定蛋白质构象变化动力学等特征的实验技术分辨率已达到10−10m,但缺点是生物分子体系中含氢基团极多,1H核磁共振波谱线易重叠而不易解析;14,15尽管目前的氢氘交换质谱技术也能利用蛋白质与溶剂分子之间的氢氘交换反应对蛋白质结构进行解析,但蛋白骨架中与α-碳原子共价结合的氢原子无法参与氢氘交换反应,而蛋白侧链上的氢原子参与的氢氘交换反应速率又极快,难以进行有效检测;16,17其次是由于蛋白质完成自我折叠转变过程的时间尺度往往在10−3−10−6s范围内,如此短的时间尺度是目前仪器分析手段所难以进行直接观测的,再加上时间分辨率的提高要比空间分辨率的提高困难得多,而目前能观测到微秒级、纳秒级时间尺度内分子运动的仪器恐怕现有技术条件较难达到.另外实验条件往往不易精确控制,得到的结果可能会产生较大的偏差,上述问题都给蛋白质结构折叠机制的实验研究带来很大的困扰.
2.2 折叠机制理论计算研究方面
理论计算模拟在空间和时间分辨率上分别可以达到纳米级和飞秒级,在时空分辨率精度上可以突破这个局限性,能够排除其它因素的干扰,并可获得任意微观量的时空分布和演化轨迹,在一定程度上弥补了目前实验所遇到的这些制约瓶颈,但在模拟时间尺度上也存在一定的缺陷.18−20生物大分子中的氢原子波动频率较快,导致时间步长设定不能太长,通常模拟设定在飞秒级,而这又导致模拟计算耗时较长,对计算机性能要求较高.尽管在模拟过程中可以通过选择Shake或者Rattle算法将模拟体系中氢原子的振动自由度消除,进而选择较大的步长,但会增加额外计算求解约束方程,导致模拟计算效率并不高.另外,蛋白质折叠都是发生在细胞内的溶液中,这也就要求模拟采用更为真实的显式溶剂水分子,而处理大量水分子的运动将消耗大量机时,而有效肽链的运动又很少.这也导致我们目前模拟计算时间尺度受限,其模拟计算结果与其实验研究的大时间尺度仍未达到完全匹配,无法真正观察清楚蛋白质在生物体内的折叠细节与全局过程.12,13为了提高现今模拟计算的时间尺度,一方面随着计算机硬件水平的快速发展,一些理论计算课题组率先开发出超级计算机(Anton),并将全原子分子动力学(MD)的模拟时间尺度提高到微秒级、甚至毫秒级,并能成功地模拟计算出不同类型结构特征模型蛋白的构象转变机制和折叠形成路径;21,22另一方面,很多理论工作者通过对生物大分子骨架和侧链结构的运动规律的研究,提出并发展出很多粗粒化模型(如 HP model,lattice model,gõ-like model和native-centric model等)和相关的模型理论(如hybrid theory,nonexplicit-chain theory等),达到准确描述生物体系(尤其是大分子蛋白质、本质无序蛋白质(IDP))分子结构运动规律进而提高计算效率.23这些研究工作进展都大大提高了目前折叠机制理论计算的研究水平.
由于蛋白质具有很高的自由度及其过渡态呈现的不稳定性,实验上很难直接捕捉到蛋白质过渡态的结构特征,而理论计算方法能够通过主成分分析(PCA)或直接测定回旋半径(radius of gyration,Rg)、均方根偏差(RMSD)、天然接触形成率(content of native contacts,ρNC)、折叠/解折叠速率(folding/unfolding rate,rF/U)等重要结构参数进而构建出折叠的自由能曲面(free energy landscape),清晰勾勒出蛋白质结构折叠的热力学特征,同时通过获取折叠过程中各个中间态以及过渡态等详细的结构特征并推断其折叠动力学行为,在一定程度上弥补了实验方面的不足.目前分子动力学模拟己经成为在原子水平上研究蛋白质等生物大分子的结构和动力学性质的一个重要工具.20,24,25迄今为止,从早期的格点模型到现今更为精确的粗粒化模型以及全原子模型,人们提出并发展了很多理论.在这些折叠理论中,被广泛接受的是蛋白质折叠的漏斗理论,26−29即蛋白质从伸展的非天然态开始,折叠过程沿着能量表面下坡方向运动,整个过程受热力学和动力学的控制,最终可以达到动力学上可及并且热力学上最稳定的态(即蛋白质结构的天然态).该天然态也对应着自由能最小态,但自由能的极小并不意味着势能的极小,其中还包含着熵的作用.此外,蛋白质能够通过多种折叠路径最终形成天然态,而折叠路径的选择又取决于环境和初始结构等条件的影响,30由于折叠问题的复杂性,蛋白质结构在细胞体内如何快速准确地折叠到其功能结构至今仍未完全解决.
相对于蛋白质折叠的微秒级、毫秒级甚至秒级时间尺度来说,目前分子动力学模拟计算能力仍然存在技术上的巨大挑战,这也导致目前的折叠理论计算研究通常应用在折叠迅速且结构典型的模型多肽(如β-hairpin、31−34Amyloid、35−40Melittin41−43和Trp-cage43−46等),小分子量蛋白质或蛋白结构域(如chymotrypsin inhibitor 2、47,48Lysozyme、49−51zinc-finer motif、52,53B domain of protein A/G21,54−58和WW-domain22,59等)等典型折叠机制的研究上,而一些具有重要生物功能的大蛋白质体系构象折叠转变过程还是无法直接通过理论计算得到.其次,目前对生物大分子结构及其体系相互作用的描述依然停留在经典的牛顿力学基础上,各种流行理论计算模拟软件主要是采用OPLS/AA、60,61GROMACS、62,63CVFF、33,53AMBER、64CHARMM65等常用力场,尽管已有很多模拟计算结果表明这些分子力场能够很好地应用于蛋白质结构折叠转变的研究.但这些力场对于蛋白质结构分子间非键相互作用的描述无法随着模拟环境(如pH、温度、离子浓度、溶剂极化效应等)的变化而调整,目前还达不到准确描述不同环境下蛋白质体系相互作用的复杂性.尽管近年来极化力场的提出和发展弥补了过去传统分子力场描述蛋白质结构体系相互作用的不准确性,66,67但由于极化力场的参数繁多且复杂,导致其对蛋白质这样的生物大分子体系折叠研究可行性不高.68因此在未来的折叠理论计算方面会进一步发展更简单更准确的力场或者设计更加有效研究折叠过程的新方法.69,70
3 模型多肽Trp-cage折叠形成机制的研究进展
以模型多肽Trp-cage为例,Trp-cage是一个由20个氨基酸组成的人工设计蛋白,它包含一段α-螺旋(Leu2−Asp9)和一段短的310-螺旋二级结构(Gly10−Ser14)及其一段C端的多脯氨酸II螺旋结构(Pro17−Pro19).模型多肽Trp-cage上N端螺旋上残基Tyr3、Trp6、Leu7、Pro12与C端中连续的三个脯氨酸(Pro17,Pro18和Pro19)一起形成了一个以Trp6为中心的疏水核,具有紧凑、明确的三级结构(图1A).模型多肽Trp-cage分子量小,结构典型且可快速折叠(约4 μs),一直被看作是研究蛋白质折叠转变机制的理想模型.71,72
3.1 Trp-cage的折叠过渡温度
目前国内外科研工作者通过核磁共振(NMR)、45,71圆二色(CD)光谱、72−74荧光猝灭光谱(FQCTS)、75−77紫外共振拉曼光谱(URRS)78等实验手段对模型多肽Trp-cage的折叠机制都已进行过深入的研究,结果表明其折叠过程可在1.0−4.1 μs范围内完成,该结果也得到了理论模拟计算的验证.79−84然而,目前对于模型多肽Trp-cage的一些具体的折叠细节过程仍然存在很大争议和疑问.例如Qiu等72通过跳温波谱技术(TJS)研究表明模型多肽Trp-cage通过两态协同的方式完成折叠过程;但Neuweiler73和Ahmed78课题组发现模型多肽Trp-cage并非只是一个简单的两态折叠蛋白;还有一些课题组利用分子动力学模拟方法对Trp-cage的折叠路径和机理进行研究发现:Trp-cage折叠转变过程中过渡态形成的温度约为400 K80或更高温度,79,81未能重复得出实验所得到的过渡态的温度(315 K);72而Day、85Duan86和Zhang87等对Trp-cage折叠过程进行的副本交换分子动力学(REMD)模拟发现,溶解温度可以和实验值相吻合.通过分析比较可以推测,这些模拟计算结果的差异性主要是由于动力学模拟所采用分子力场与溶剂模型的不同.基于这种模拟计算和实验研究结果的不一致,最近Ołdziej等45采用二维核磁共振技术与分子动力学模拟相结合的方法对Trp-cage的折叠过渡温度进行了研究,结果表明:由于模型多肽Trpcage折叠复杂性(关键氨基酸Trp6和Pro12的疏水长程协同作用与N端α-helix解旋过程同时发生),其折叠过渡温度并非在某一温度时刻,而是在311−317 K范围内.
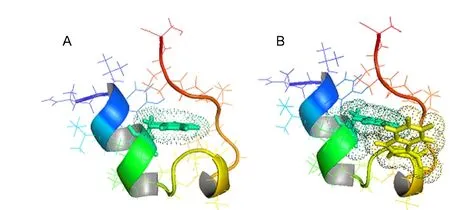
图1 模型多肽Trp-cage71(A)及其突变体P12W(Trp2-cage)98(B)的结构示意图Fig.1 Schematic structures of the Trp-cage mini-protein71(A)and its mutant P12W(Trp2-cage)98(B)
3.2 Trp-cage的折叠形成机制
目前对于模型多肽Trp-cage折叠过程中各个折叠细节发生的顺序一直都未达成共识.例如国内外一些研究蛋白折叠课题组发现盐桥Asp9/Arg16的率先形成对模型多肽Trp-cage的折叠发挥着至关重要的作用,70,71,81然而这恰恰与实验得到的结果相反.通过圆二色谱实验研究发现,将盐桥Asp9/Arg16去掉后,模型多肽Trp-cage的折叠时间未发生明显的变化,73并且最近研究也发现模拟计算起始构象中的盐桥Asp9/Arg16甚至并不能稳定存在,在模拟过程中会发生断裂消失,并且N端α-helix的稳定存在与其形成具有紧密联系.45Chowdhury82和Hu83等运用全原子分子动力学模拟发现疏水核心Trp6的包埋掩藏是模型多肽Trp-cage折叠过程的关键限速步骤,而Juraszek等84采用副本交换分子动力学模拟发现Trp-cage折叠过程的限速步骤并非绝对为疏水核心吲哚环的包埋,其研究结果表明模型多肽Trpcage N-端上的α-螺旋结构形成于折叠过程的早期,之后疏水残基逐渐向疏水核心(Trp6)靠拢,最终疏水核中的Trp6侧链上的吲哚环完整包埋而结束整个折叠过程.Mok等88通过脉冲光谱实验研究发现在非折叠态结构的Trp-cage中存在疏水核结构,说明疏水核的塌陷推动蛋白结构的整个折叠进程,即所谓的疏水塌陷模型.89−92Trp-cage这种疏水塌陷折叠模型的提出也得到了其他课题组的支持,而Ahmed课题组78发现结构中N端的α-螺旋结构的形成先于疏水核的塌陷,Juraszek课题组84却认为Trpcage的折叠形成遵循成核-聚集和扩散-碰撞模型.可见,虽然模型多肽Trp-cage只有20个氨基酸组成,且其结构简单紧凑且折叠迅速,但其折叠机制仍然存在争议.
为了探究该模型多肽Trp-cage复杂的折叠过程,我们曾采用温控分子动力学(TCMD)方法93对模型多肽Trp-cage在真空和水溶剂条件下的折叠与解折叠过程进行了深入的研究,得到了Trp-cage折叠过程中的重要中间态结构,为其折叠与解折叠机制的揭示提供了直接、可靠的依据;70结果发现溶剂水分子可通过疏水作用诱导Trp-cage形成一个亚稳态(MSOL)和一个非天然态疏水核的过渡态(TS)结构,推动肽链疏水残基间的相互作用,进而促进肽链结构疏水核的快速收缩和正确折叠.为深入理解Trpcage天然折叠态构型的稳定形成机制,我们采用分子动力学结合残基点突变(甘氨酸替换)的方法研究Trp-cage中各个氨基酸侧链基团在其结构稳定性中的作用,通过与野生天然态蛋白结构稳定性特征数据对比,确定了每个残基侧链在其整个结构稳定性中的作用.如残基Tyr3与Pro19间的疏水作用、残基Ser14和Arg16间的氢键作用以及Pro17所形成的三级天然接触等稳定作用对Trp-cage整体结构的稳定性起着特殊重要的作用(图2).另外,具有相同侧链的氨基酸残基(Ser13和Ser14)由于其侧链所处位置的不同导致其对结构的稳定性的贡献程度也不同;盐桥在模型多肽结构中本身稳定性非常高,不易被突变所破坏.这些结果为理解模型多肽Trpcage折叠构型的稳定形成机制提供了重要的理论依据.94
3.3 Trp-cage折叠过程中发挥关键作用的氨基酸
正是由于蛋白质折叠问题的复杂性,很多研究者采用残基定点突变的方法化解折叠问题的复杂性,对模型多肽Trp-cage结构中一些关键残基所形成的重要作用对其折叠动力学行为的影响进行了大量的研究.即将每个点突变看作是探测蛋白质结构及其动力学行为的探针,通过与野生天然态蛋白体系平衡状态下的结构稳定性和动力学特征数据对比,确定哪些残基及其侧链的相互作用对蛋白质结构折叠转变产生重要影响.例如Andersen等71,92通过对Trp-cage的N9D和R16K突变体的研究发现,Asp9侧链上的羧基与Arg16侧链上胍基结合所形成的盐桥对Trp-cage折叠态的稳定性起到至关重要的作用;也有人通过残基点突变的方法发现Trpcage结构内部残基Ser14侧链所形成的氢键作用对Trp-cage结构的折叠形成具有重要的作用.92最近,She等95−97通过分子动力学方法发现四个疏水氨基酸残基(Tyr3,Trp6,Leu7和Pro19)(如图2所示)与Trp-cage的折叠动力学行为密切相关,并认为这四个关键疏水残基的相互作用主导着Trp-cage的整个折叠过程.基于这种疏水残基在折叠过程中的重要作用,Gai等98设计了一个全新的多肽模型“Trp2-cage”(即Trp-cage的突变体P12W)(如图1B所示),表明Trp6/Trp12侧链的叠合作用可极大地加速蛋白质的折叠速率,使Trp2-cage的折叠过程可在1 μs范围内完成.

图2 模型多肽Trp-cage的重要氨基酸95,96和“非重要氨基酸”94Fig.2 Four key residues95,96and other“non-key residues”94 in the Trp-cage mini-protein
通过上述研究结果发现,尽管目前有很多实验采用残基点突变的方法研究蛋白质折叠路径,但实验方面得到的数据并不能给出折叠路径及其过渡态结构的细节特征,71−78而现有模拟计算方面的残基点突变几乎都是针对模型多肽Trp-cage结构中个别重要残基位点,且仅是其它残基的替换,通过分析比较不同残基侧链在同一位点上对其折叠过程的影响,得到很多关于模型多肽Trp-cage折叠机理方面的重要成果,同时进一步验证了构成Trp-cage疏水笼结构氨基酸对其整个结构稳定性和折叠动力学行为的主导重要性.79−84,95−98然而,除了已有文献报道关注的关键氨基酸外,模型多肽中那些其它的“非重要”氨基酸残基在我们以前残基点突变对Trpcage折叠天然态构型稳定机制的研究中发现它们对于结构的稳定形成仍具有一定的特殊作用,并且其作用程度与它们侧链位置及其稳定性都存在紧密联系(如图2所示).94这些“非重要”氨基酸侧链在模型多肽折叠过程中又是如何相互作用,对肽链的折叠完成发挥怎样的效用等问题并没有太多具体的报道,尤其是各个侧链分子之间相互作用的协同效应在折叠与解折叠过程中的作用依然不明确.或许这些问题的阐述对于全面理解Trp-cage折叠机制能够提供重要理论依据.相对于实验技术条件来讲,通过理论模拟计算研究在一定程度上能够克服实验上难以获得结构细节特征和实验条件限制因素的制约.尽管现有的全原子水平分子动力学方法在模拟的时间尺度上仍然受到一定条件的限制,一些研究者努力通过调节温度采用副本交换分子动力学(REMD),12,99−102改变压强,103−105添加变性剂,102,106,107施加额外机械力的拉伸分子动力学(SMD)108−112等方法以加速蛋白质的折叠过程,进而缩短模拟时间.但因这些方法是在非平衡状态下模拟蛋白质的动力学行为,有可能得不到真实的运动轨迹,因此这严重限制了对蛋白质动力学行为的理解.为此,我们在研究模型多肽Trp-cage、70,94微管活性肽段93,113,114和G蛋白结构域(GB1)69的折叠形成过程中,基于不改变多肽和蛋白质的折叠路径,但能加快构象转化速率和跨域较高构象能垒的思想,提出的一些新的模拟计算方法(例如温控分子动力学方法根据肽链构象转变行为适时调温可快速有效得到各个温度梯度下构象的稳定的平衡态,清晰显示肽链折叠转变路径;70,93距离限制性分子动力学方法结合原子力显微镜(AFM)拉伸实验原理,通过逐步调控肽链两端距离并固定肽链两端原子分别进行长程分子动力学模拟得到各个稳定平衡态,清晰给出肽链从完全伸展态到天然折叠态转变过程中的每个关键中间态与过渡态,并详尽刻画出整个折叠细节过程,69)不仅能够有效研究蛋白质或多肽在平衡状态下的折叠动力学行为,而且可快速获取有效、稳定的中间态构象以及过渡态结构,有效分解蛋白结构内部复杂的相互作用网络,给出一些实验与常规模拟方法无法得到的折叠细节,为折叠机制提供直接、可靠的依据.相信在未来计算硬件资源迅猛变革发展的同时,还会设计出更好的新算法加速蛋白质折叠难题的破解.
4 结束语
蛋白质天然态折叠结构是其行使生物功能的生物物理基础,搞清楚蛋白质结构折叠机理对于理清结构与功能的关系至关重要.蛋白质肽链的折叠形成过程的驱动力主要取决于肽链上各个残基之间的相互作用.因此,蛋白质折叠机制的研究问题中首要考虑的就是氨基酸的侧链特性(如静电、疏水、盐桥、氢键、位阻等).迷你蛋白Trp-cage分子量小且结构典型,目前是研究蛋白质结构折叠机理的良好模型多肽,哪些侧链特性对其模型多肽Trpcage折叠过程有影响?对于具有影响肽链折叠的侧链中,它们的影响比重又是如何分配?另外侧链间所形成相互作用的协同性对折叠完成的影响又是怎样?目前这些折叠细节的问题迫切要求我们能在未来的研究工作中提供能够解决上述问题的思路,同时该问题解决的思路也能够对我们理解蛋白质肽链折叠形成过程具有极大的促进作用.
(1) Yan,L.F.;Sun,Z.R.Molecular Structure of Protein;Tsinghua University Press:Beijing,1999. [阎隆飞,孙之荣.蛋白质分子结构.北京:清华大学出版社,1999.]
(2) Vendruscolo,M.Curr.Opin.Struct.Biol.2007,17,15.doi:10.1016/j.sbi.2007.01.002
(3) Fink,A.L.Curr.Opin.Struct.Biol.2005,15,35.doi:10.1016/j.sbi.2005.01.002
(4) Karplus,M.;McCammon,J.A.Nat.Struct.Biol.2002,9,646.doi:10.1038/nsb0902-646
(5) Parak,F.G.Rep.Prog.Phys.2003,66,103.doi:10.1088/0034-4885/66/2/201
(6) Thomas,P.J.;Qu,B.H.;Pedersen,P.L.Trends Biochem.Sci.1995,20,456.doi:10.1016/S0968-0004(00)89100-8
(7)Gellman,S.H.;Woolfson,D.N.Nat.Struct.Biol.2002,9,408.doi:10.1038/nsb0602-408
(8) Chellgren,B.W.;Creamer,T.P.Biochemistry 2004,43,5864.doi:10.1021/bi049922v
(9) Woody,R.Adv.Biophys.Chem.1992,2,37.
(10) Zhang,Z.Q.Acta Phys.-Chim.Sin.2012,28,2381.[张竹青.物理化学学报,2012,28,2381.]doi:10.3866/PKU.WHXB201209144
(11) Chen,K.X.;Jiang,H.L.;Ji,R.Y.Computer Aided Drug Design——Principle,Methods and Application;Shanghai Scientific Technology Press:Shanghai,2000.[陈凯先,蒋华良,嵇汝运.计算机辅助药物设计——原理、方法及应用.上海:上海科学技术出版社,2000.]
(12) Thirumalai,D.;Liu,Z.X.;O'Brien,E.P.;Reddy,G.Curr.Opin.Struct.Biol.2013,23,22.doi:10.1016/j.sbi.2012.11.010
(13) Cai,W.S.;Chipot,C.Acta Chim.Sin.2013,71,159.[蔡文生,Chipot,C.化学学报,2013,71,159.]doi:10.6023/A12110930
(14) Fuentes,G.;Nederveen,A.J.;Kaptein,R.;Boelens,R.;Bonvin,A.M.J.Biomol.NMR 2005,33,175.doi:10.1007/s10858-005-3207-9
(15) Wong,K.B.;Clarke,J.;Bond,C.J.;Neira,J.L.;Freund,S.M.;Fersht,A.R.;Daggett,V.J.Mol.Biol.2000,296,1257.doi:10.1006/jmbi.2000.3523
(16) Engen,J.R.Anal.Chem.2009,81,7870.doi:10.1021/ac901154s
(17) Iacob,R.E;Engen,J.R.J.Am.Soc.Mass Spectrom.2012,23,1003.doi:10.1007/s13361-012-0377-z
(18) Dill,K.A.;Ozkan,B.S.;Shell,M.;Weikl,T.R.Ann.Rev.Biophys.2008,37,289.doi:10.1146/annurev.biophys.37.092707.153558
(19) Onuchic,J.N.;Wolyness,P.G.Curr.Opin.Struct.Biol.2004,14,70.doi:10.1016/j.sbi.2004.01.009
(20) Rizzuti,B.;Daggett,V.Arch.Biochem.Biophys.2013,531,128.doi:10.1016/j.abb.2012.12.015
(21) Lindorff-Larsen,K.;Piana,S.;Dror,R.O.;Shaw,D.E.Science 2011,334,517.doi:10.1126/science.1208351
(22) Shaw,D.E.;Maragakis,P.;Lindorff-Larsen,K.;Piana,S.;Dror,R.O.;Eastwood,M.P.;Bank,J.A.;Jumper,J.M.;Salmon,J.K.;Shan,Y.;Wriggers,W.Science 2010,330,341.doi:10.1126/science.1187409
(23) Chan,H.S.;Zhang,Z.;Wallin,S.;Liu,Z.Annu.Rev.Phys.Chem.2011,62,301.doi:10.1146/annurev-physchem-032210-103405
(24) Goodfellow,J.M.;Moss,D.S.Computer Modeling of Biomolecular Process;Bllis Horwood:New York,1992.
(25) Warshel,A.Computer Modeling of Chemical Reactions in Enzymes and Solutions;Jonh Wilev&Sons:New York,1991.
(26) Leopold,P.;Montal,M.;Onuchic,J.Proc.Natl.Acad.Sci.U.S.A.1992,89,8721.doi:10.1073/pnas.89.18.8721
(27) Bryngelson,J.D.;Onuchic,J.N.;Socci,N.D.;Wolynes,P.G.Proteins 1995,21,167.
(28) Mirny,L.A.;Shakhnovich,E.I.Annu.Rev.Biophys.Biomol.Struct.2001,30,361.doi:10.1146/annurev.biophys.30.1.361
(29) Dill,K.A.;Chan,H.S.Nat.Struct.Biol.1997,4,10.doi:10.1038/nsb0197-10
(30) Anfinsen,C.B.Science 1973,181,223.doi:10.1126/science.181.4096.223
(31) Thukral,L.;Smith,J.C.;Daidone,I.J.Am.Chem.Soc.2009,131,18147.doi:10.1021/ja9064365
(32) Ma,B.;Nussinov,R.J.Mol.Biol.2000,296,1091.doi:10.1006/jmbi.2000.3518
(33) Wu,X.M.;Yang,G.;Zu,Y.G.;Zhou,L.J.Comput.Biol.Chem.2012,38,1.doi:10.1016/j.compbiolchem.2012.02.003
(34) Liu,F.F.;Dong,X.Y.;Sun,Y.J.Mol.Graph.Model.2008,27,421.doi:10.1016/j.jmgm.2008.07.002
(35) Li,W.;Zhang,J.;Su,Y.;Wang,J.;Qin,M.;Wang,W.J.Phys.Chem.B 2007,111,13814.doi:10.1021/jp076213t
(36) Lazo,N.D.;Grant,M.A.;Condron,M.C.;Rigby,A.C.;Teplow,D.B.Protein Sci.2005,14,1581.
(37) Guarnera,E.;Pellarin,R.;Caflisch,A.Biophys.J.2009,97,1737.doi:10.1016/j.bpj.2009.06.047
(38) Cecchini,M.;Curcio,R.;Pappalardo,M.;Melki,R.;Caflisch,A.J.Mol.Biol.2006,357,1306.doi:10.1016/j.jmb.2006.01.009
(39) Convertino,M.;Pellarin,R.;Catto,M.;Carotti,A.;Caflisch,A.Protein Sci.2009,18,792.
(40) Scherzer-Attali,R.;Pellarin,R.;Convertino,M.;Frydman-Marom,A.;Egoz-Matia,N.;Peled,S.;Levy-Sakin,M.;Shalev,D.E.;Caflisch,A.;Gazit,E.;Segal,D.PloS One 2010,5,e11101.
(41) Terwilliger,T.C.;Eisenberg,D.J.Biol.Chem.1982,257,6016.
(42) Tanizaki,S.;Clifford,J.;Connelly,B.D.;Feig,M.Biophys.J.2008,94,747.doi:10.1529/biophysj.107.116236
(43) Predeus,A.V.;Gul,S.;Gopal,S.M.;Feig,M.J.Phys.Chem.B 2012,116,8610.doi:10.1021/jp300129u
(44) Shao,Q.;Zhu,W.L.;Gao,Y.Q.J.Phys.Chem.B 2012,116,13848.doi:10.1021/jp307684h
(45) Halabis,A.;Zmudzinska,W.;Liwo,A.;Ołdziej,S.J.Phys.Chem.B 2012,116,6898.doi:10.1021/jp212630y
(46)Adams,C.M.;Kjeldsen,F.;Zubarev,R.A.;Budnik,B.A.;Haselmann,K.F.J.Am.Soc.Mass Spectrom.2004,15,1087.doi:10.1016/j.jasms.2004.04.026
(47) Miklos,A.C.;Sarkar,M.;Wang,Y.;Pielak,G.J.J.Am.Chem.Soc.2011,133,7116.doi:10.1021/ja200067p
(48) Feig,M.;Sugita,Y.J.Phys.Chem.B 2012,116,599.doi:10.1021/jp209302e
(49) Klein-Seetharaman,J.;Oikawa,M.;Grimshaw,S.B.;Wirmer,J.;Duchardt,E.;Ueda,T.;Imoto,T.;Smith,L.J.;Dobson,C.M.;Schwalbe,H.Science 2002,295,1719.doi:10.1126/science.1067680
(50) Radford,S.E.;Dobson,C.M.;Evans,P.A.Nature 1992,358,302.doi:10.1038/358302a0
(51) Xu,J.;Baase,W.A.;Baldwin,E.;Matthews,B.W.Protein Sci.1998,7,158.
(52) Li,W.;Zhang,J.;Wang,J.;Wang,W.J.Am.Chem.Soc.2008,130,892.doi:10.1021/ja075302g
(53) Palmer,A.G.,III;Rance,M.;Wright,P.E.J.Am.Chem.Soc.1991,113,4371.doi:10.1021/ja00012a001
(54) Gronenborn,A.M.;Filpula,D.R.;Essig,N.Z.;Achari,A.;Whitlow,M.;Wingfield,P.T.;Clore,G.M.Science 1991,253,657.doi:10.1126/science.1871600
(55) Odaert,B.;Jean,F.;Boutillon,C.;Buisine,E.;Melnyk,O.;Tartar,A.;Lippens,G.Protein Sci.1999,8,2773.
(56) Dahiyat,B.I.;Mayo,S.L.Science 1997,278,82.doi:10.1126/science.278.5335.82
(57) McCallister,E.L.;Alm,E.;Baker,D.Nat.Struct.Biol.2000,7,669.doi:10.1038/77971
(58) Kmiecik,S.;Kolinski,A.Biophys.J.2008,94,726.doi:10.1529/biophysj.107.116095
(59) Hu,J.P.;He,H.Q.;Jiao,X.;Chang,S.Mol.Simulat.2013,doi:10.1080/08927022.2013.773431
(60) Jorgensen,W.L.;Tirado-Rives,J.J.Am.Chem.Soc.1988,110,1657.doi:10.1021/ja00214a001
(61) Jorgensen,W.L.;Maxwell,D.S.;Tirado-Rives,J.J.Am.Chem.Soc.1996,118,11225.doi:10.1021/ja9621760
(62) Christen,M.;Hunenberger,P.H.;Bakowies,D.;Baron,R.;Bürgi,R.;Geerke,D.P.;Heinz,T.N.;Kastenholz,M.A.;Kräutler,V.;Oostenbrink,C.;Peter,C.;Trzesniak,D.;van Gunsteren,W.F.J.Comput.Chem.2005,26,1719.
(63) Hess,B.;Kutzner,C.;van der Spoel,D.;Lindahl,E.J.Chem.Theory Comput.2008,4,435.doi:10.1021/ct700301q
(64) Weiner,S.J.;Kollman,P.A.;Case,D.A.;Singh,C.;Ghio,C.;Alagona,G.;Profeta,S.;Weiner,P.J.Am.Chem.Soc.1984,106,765.doi:10.1021/ja00315a051
(65) Brooks,B.R.;Bruccoleri,R.E.;Olafson,B.D.;States,D.J.;Swaminathan,S.;Karplus,M.J.Comput.Chem.1983,4,187.
(66) Halgren,T.A.;Damm,W.Curr.Opin.Struct.Biol.2001,11,236.doi:10.1016/S0959-440X(00)00196-2
(67) Kaminski,G.A.;Stern,H.A.;Berne,B.J.;Friesner,R.A.;Cao,Y.X.;Murphy,R.B.;Zhou,R.;Halgren,T.A.J.Comput.Chem.2002,23,1515.doi:10.1002/jcc.10125
(68) Jorgensen,W.L.J.Chem.Theory Comput.2007,3,1877.doi:10.1021/ct700252g
(69)Wu,X.M.;Yang,G.;Zhou,L.J.Theor.Chem.Acc.2012,131,1229.doi:10.1007/s00214-012-1229-4
(70)Wu,X.M.;Yang,G.;Zu,Y.G.;Fu,Y.J.;Zhou,L.J.;Yuan,X.H.Mol.Simulat.2012,38,161.doi:10.1080/08927022.2011.610795
(71) Neidigh,J.W.;Fesinmeyer,R.M.;Andersen,N.H.Nat.Struct.Biol.2002,9,425.doi:10.1038/nsb798
(72) Qiu,L.;Pabit,S.A.;Roitberg,A.E.;Hagen,S.J.J.Am.Chem.Soc.2002,124,12952.doi:10.1021/ja0279141
(73) Neuweiler,H.;Doose,S.;Sauer,M.Proc.Natl.Acad.Sci.U.S.A.2005,102,16650.doi:10.1073/pnas.0507351102
(74) Streicher,W.W.;Makhatadze,G.I.Biochemistry 2007,46,2876.doi:10.1021/bi602424x
(75) Iavarone,A.T.;Parks,J.H.J.Am.Chem.Soc.2005,127,8606.doi:10.1021/ja051788u
(76) Qiu,L.L.;Hagen,S.J.Chem.Phys.2004,307,243.doi:10.1016/j.chemphys.2004.04.030
(77) Qiu,L.L.;Hagen,S.J.J.Am.Chem.Soc.2004,126,3398.doi:10.1021/ja049966r
(78)Ahmed,Z.;Beta,I.A.;Mikhonin,A.V.;Asher,S.A.J.Am.Chem.Soc.2005,127,10943.doi:10.1021/ja050664e
(79) Paschek,D.;Nymeyer,H.;Garcia,A.E.J.Struct.Biol.2007,157,524.doi:10.1016/j.jsb.2006.10.031
(80) Pitera,J.W.;Swope,W.Proc.Natl.Acad.Sci.U.S.A.2003,100,7587.doi:10.1073/pnas.1330954100
(81) Zhou,R.Proc.Natl.Acad.Sci.U.S.A.2003,100,13280.doi:10.1073/pnas.2233312100
(82)Chowdhury,S.;Lee,M.C.;Duan,Y.J.Phys.Chem.B 2004,108,13855.doi:10.1021/jp0478920
(83)Hu,Z.;Tang,Y.;Wang,H.;Zhang,X.;Lei,M.Arch.Biochem.Biophys.2008,475,140.doi:10.1016/j.abb.2008.04.024
(84) Juraszek,J.;Bolhuis,P.G.Proc.Natl.Acad.Sci.U.S.A.2006,103,15859.doi:10.1073/pnas.0606692103
(85) Day,R.;Paschek,D.;García,A.E.Proteins 2010,78,1889.
(86) Duan,L.L.;Mei,Y.;Li,Y.L.;Zhang,Q.G.;Zhang,D.W.;Zhang,J.Z.H.Sci.China Ser.B 2010,53,196.doi:10.1007/s11426-009-0196-7
(87) Mei,Y.;Wei,C.Y.;Yip,Y.M.;Ho,C.Y.;Zhang,J.Z.H.;Zhang,D.W.Theor.Chem.Acc.2012,131,1168.doi:10.1007/s00214-012-1168-0
(88) Mok,K.H.;Kuhn,L.T.;Goez,M.;Day,I.J.;Lin,J.C.;Andersen,N.H.;Hore,P.J.Nature 2007,447,106.doi:10.1038/nature05728
(89) Brylinski,M.;Konieczny,L.;Roterman,I.Comput.Biol.Chem.2006,30,255.doi:10.1016/j.compbiolchem.2006.04.007
(90)Arai,M.;Kondrashkina,E.;Kayatekin,C.;Matthews,C.R.;Iwakura,M.;Bilsel,O.J.Mol.Biol.2007,368,219.doi:10.1016/j.jmb.2007.01.085
(91) Dill,K.A.;Fiebig,K.M.;Chan,H.S.Proc.Natl.Acad.Sci.U.S.A.1993,90,1942.doi:10.1073/pnas.90.5.1942
(92) Barua,B.;Lin,J.C.;Williams,V.D.;Kummler,P.;Neidigh,J.W.;Andersen,N.H.Protein Eng.Des.Sel.2008,21,171.doi:10.1093/protein/gzm082
(93)Wu,X.M.;Zu,Y.G.;Yang,Z.W.;Fu,Y.J.;Zhou,L.J.;Yang,G.Acta Phys.-Chim.Sin.2009,25,773.[吴晓敏,祖元刚,杨志伟,付玉杰,周丽君,杨 刚.物理化学学报,2009,25,773.]doi:10.3866/PKU.WHXB20090333
(94)Wu,X.M.;Yang,G.;Zu,Y.G.;Fu,Y.J.;Yuan,X.H.Comput.Theor.Chem.2011,973(1−3),1.
(95) Yao,X.Q.;She,Z.S.Biochem.Biophys.Res.Commun.2008,373,64.doi:10.1016/j.bbrc.2008.05.179
(96) Gao,M.;Zhu,H.Q.;Yao,X.Q.;She,Z.S.Biochem.Biophys.Res.Commun.2010,392,95.doi:10.1016/j.bbrc.2010.01.003
(97) Gao,M.;Yao,X.Q.;She,Z.S.;Liu,Z.R.;Zhu,H.Q.Acta Phys.-Chim.Sin.2010,26,1998.[高 萌,姚新秋,佘振苏,刘志荣,朱怀球.物理化学学报,2010,26,1998.]doi:10.3866/PKU.WHXB20100733
(98) Bunagan,M.R.;Yang,X.;Saven,J.G.;Gai,F.J.Phys.Chem.B 2006,110,3759.
(99) Day,R.;Bennion,B.J.;Ham,S.;Daggett,V.J.Mol.Biol.2002,322,189.doi:10.1016/S0022-2836(02)00672-1
(100) Zhou,R.H.;Berne,B.J.;Germain,R.Proc.Nat.Acad.Sci.U.S.A.2001,98,14931.doi:10.1073/pnas.201543998
(101) Settanni,G.;Fersht,A.R.Biophys.J.2008,94,4444.doi:10.1529/biophysj.107.122606
(102) Kony,D.B.;Hünenberger,P.H.;van Gunsteren,W.F.Protein Sci.2007,16,1101.
(103) Wroblowski,B.;Diaz,J.F.;Heremans,K.;Engelborghs,Y.Proteins 1996,25,446.
(104) Wang,J.H.;Zhang,Z.Y.;Liu,H.Y.;Shi,Y.Y.Acta Biophys.Sin.2004,20,315.[王吉华,张志勇,刘海燕,施蕴渝.生物物理学报,2004,20,315.]
(105) Hillson,N.;Onuchic,J.N.;García,A.E.Proc.Natl.Acad.Sci.U.S.A.1999,96,14848.doi:10.1073/pnas.96.26.14848
(106) Bennion,B.J.;Daggett,V.Proc.Natl.Acad.Sci.U.S.A.2003,100,5142.doi:10.1073/pnas.0930122100
(107) Rogne,P.;Ozdowy,P.;Richter,C.;Saxena,K.;Schwalbe,H.;Kuhn,L.T.PloS One 2012,7,e41301.
(108) Rief,M.;Gautel,M.;Oesterhelt,F.;Fernandez,J.M.;Gaub,H.E.Science 1997,276,1109.doi:10.1126/science.276.5315.1109
(109) Fernandez,J.M.;Li,H.Science 2004,303,1674.doi:10.1126/science.1092497
(110) Karsai,Á.;Kellermayer,M.S.;Harris,S.P.Biophys.J.2011,101,1968.doi:10.1016/j.bpj.2011.08.030
(111) Borgia,A.;Steward,A.;Clarke,J.Angew.Chem.Int.Edit.2008,47,6900.doi:10.1002/anie.v47:36
(112) Garcia-Manyes,S.;Dougan,L.;Badilla,C.L.;Brujic,J.;Fernandez,J.M.Proc.Natl.Acad.Sci.U.S.A.2009,106,10534.doi:10.1073/pnas.0901213106
(113)Wu,X.M.;Yang,G.;Zu,Y.G.;Yang,Z.W.;Zhou,L.J.In Silico Biol.2009,9,271.
(114)Yang,G.;Wu,X.M.;Zu,Y.G.;Yang,Z.W.;Zhou,L.J.J.Theor.Comput.Chem.2009,8,317.
