1株喜盐Actinopolyspora菌属新种的系统分类
2013-09-12关统伟赵辉平车振明
关统伟,赵辉平,车振明
(西华大学生物工程学院,四川成都 610039)
大英盐湖位于四川省中部地区的遂宁市大英县境内,1.5亿年前地球的两次造山运动在四川大英县形成了地下古盐湖盆地,含盐量超过22%,其盐卤储量达42亿 t[1]。2011年自大英盐湖中分离获得1株极端嗜盐产孢放线菌,菌株编号为TRM4064,经16S rRNA系统发育分析初步确定为1株潜在的新种,经形态学观察、生理生化检验、细胞化学和分子遗传学等多相分类手段的测定,确认菌株TRM4064为多孢放线菌属(Actinopolyspora)的一个新种。研究目的是通过多相分类技术确定该菌种的系统分类地位。多相分类是采用现代分类的多种方法,综合表现型和遗传型信息进行分类鉴定和系统发育研究的过程,它是当前研究微生物各级分类单位最有效的方法。
1 材料与方法
1.1 材料
1.1.1 菌株 2011年12月自大英盐湖样品中分离获得的1株极端嗜盐放线菌,菌株编号为TRM4064。
1.1.2 主要试剂及仪器 QIAquik PCR纯化试剂盒(QIAGEN)、蛋白酶 K(Merck)、Taq酶(TaKa-Ra)、dNTPs(TaKaRa)、DNA Marker(Sangon),其他生化试剂均为国产分析纯。凝胶成像仪(Biorad)、电泳仪(Bio-rad)、PCR 扩增仪(Bio-rad)。
1.2 方法
1.2.1 菌种形态特征观察和生理生化实验 用光学显微镜和扫描电子显微镜观察菌株的形态和细胞的运动性。碳、氮源生长实验采用Mata等[2]建议的培养基(NaCl 100 g,KCl 2 g,MgSO4.7H2O 0.2 g,KNO31 g,(NH4)2HPO41 g,KH2PO40.5 g);抗生素敏感性实验和其他生理生化实验参照文献[2-3]描述的方法进行。
1.2.2 菌种醌、脂肪酸及细胞全水解糖组分分析微生物细胞醌组分的提取和纯化参照Collins[4]的方法,然后根据Groth[5]的方法,利用高压液相色谱(HPLC)测定醌组分。细胞脂肪酸采用Sasser[6]的方法测定。放线菌全细胞水解糖分析主要参照Hasegawa等[7]描述的方法。
1.2.3 DNA(G+C)mol% 的测定与 DNA-DNA分子杂交 根据文献[8]的程序对菌株TRM4064进行(G+C)mol%的测定,采用高压液相色谱(HPLC)进行分析。DNA-DNA分子杂交方法主要依照 Ezaki[9]和徐丽华等[3]所描述的方法进行。
1.2.4 16S rRNA基因测定与系统进化分析DNA的提取和 PCR扩增程序采用 Hezayen[10]的方法,然后将PCR产物送上海生物工程公司测序。用Blast搜索程序从GenBank等公共数据库中调出相似性较高的相关菌株的16S rRNA基因序列,用 CLUSTAL_X进行多序列比对,使用MEGA4.0 软件采用邻接法[11]聚类分析,并构建出系统进化树。同时,重复取样1 000次进行自展值分析来评估系统进化树的拓扑结构的稳定性。
2 结果与分析
2.1 表型特征
菌株TRM4064是好养性微生物,具有很强的嗜盐性,革兰染色阳性,产生发达的孢子。气生菌丝持续生长形成孢子链,孢子链上含有10个以上的孢子,孢子链呈直线型,孢子有椭圆形和圆柱形,表面光滑,不运动,不产生孢子囊,见图1。

图1 TRM4064菌株的电镜照片Fig.1 Transmission electron micrograph of strain TRM4064
菌株TRM4064能够在28~55℃范围内生长,能够耐受10% ~20%的NaCl盐浓度,可以在pH值为6~8的培养基上生长。氧化酶实验为阴性,过氧化氢酶实验结果为阳性,吲哚实验和甲基红实验结果为阴性。硝酸盐不能够被还原,牛奶凝固不被胨化,H2S实验为阳性。Tween 20、Tween 40、Tween 60和 Tween 80水解,纤维素分解,明胶被液化,酪蛋白被水解,但淀粉和尿酶水解为阴性。药敏实验结果表明该菌对ampicillin(10 μg)、azithromycin(15 μg)、carbenicillin(100 μg)、chloramphenicol(30 μg)、gentamicin(120 μg)、erythromycin(15 μg)、lincomycin(2 μg)、nitrofurantoin(300 μg)、tobramycin(10 μg)、novobiocin(30 μg)、rifampicin(30 μg)、tetracycline(30 μg)、streptomycin(10 μg)和 vancomycin(30 μg)敏感,但对 kanamycin(30 μg)、polymyxin B(300 UI)和 nalidixic acid(30 μg)不敏感。菌株TRM4064另外的一些表型特征及其与最近的同源菌株的差异性见表1。在表型特征上实验数据支持该菌为一个新的物种。
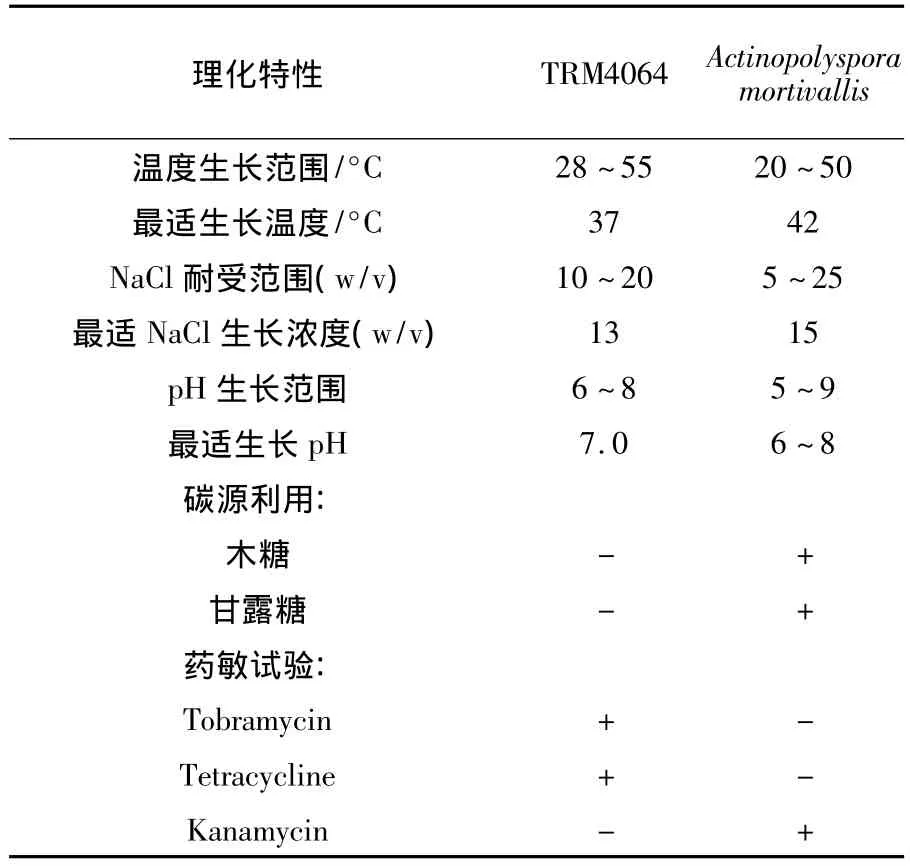
表1 菌株TRM4064与最近同源菌株的表型差异Table 1 Differential characteristics of strain TRM4064 and Actinopolyspora mortivallis
2.2 全细胞水解糖、醌、脂肪酸组分及其细胞壁磷脂分析
根据实验结果分析,菌株TRM4064的主要的泛醌为 MK-10(H4)(38.2%)、MK-9(H4)(25.1%)、MK-9(H2)(28.6%)和 MK-8(H4)(7.3%),TRM4064的主要泛醌与多孢放线菌属的醌型基本一致,只是在含量上有一定的差异。如Actinopolyspora erythraea的优势醌为MK-9(H4)(51.9%),Actinopolyspora alba 的优势醌为 MK-9(H4)(44.9%)和 MK-10(H4)(43.9%)[12]。菌株TRM4064细胞内主要的脂肪酸组分及其含量是 anteiso-C17:0(36.9%)和 iso-C17:0(19.3%),这与亲缘菌株Actinopolyspora mortivallis基本一致,在脂肪酸组分及其含量上与Actinopolyspora mortivallis有一定的差异性,具体的脂肪酸信息见表2。菌株TRM4064的全细胞水解糖分组成是木糖、葡萄糖、核糖和阿拉伯糖,与Actinopolyspora菌属的细胞水解糖糖型一致。薄板层析法实验结果表明菌株TRM4064的细胞磷脂是心磷脂、磷脂酰甘油、卵磷脂、磷脂酰肌醇和2个未知的磷脂类型,与该属的磷脂组成相一致。
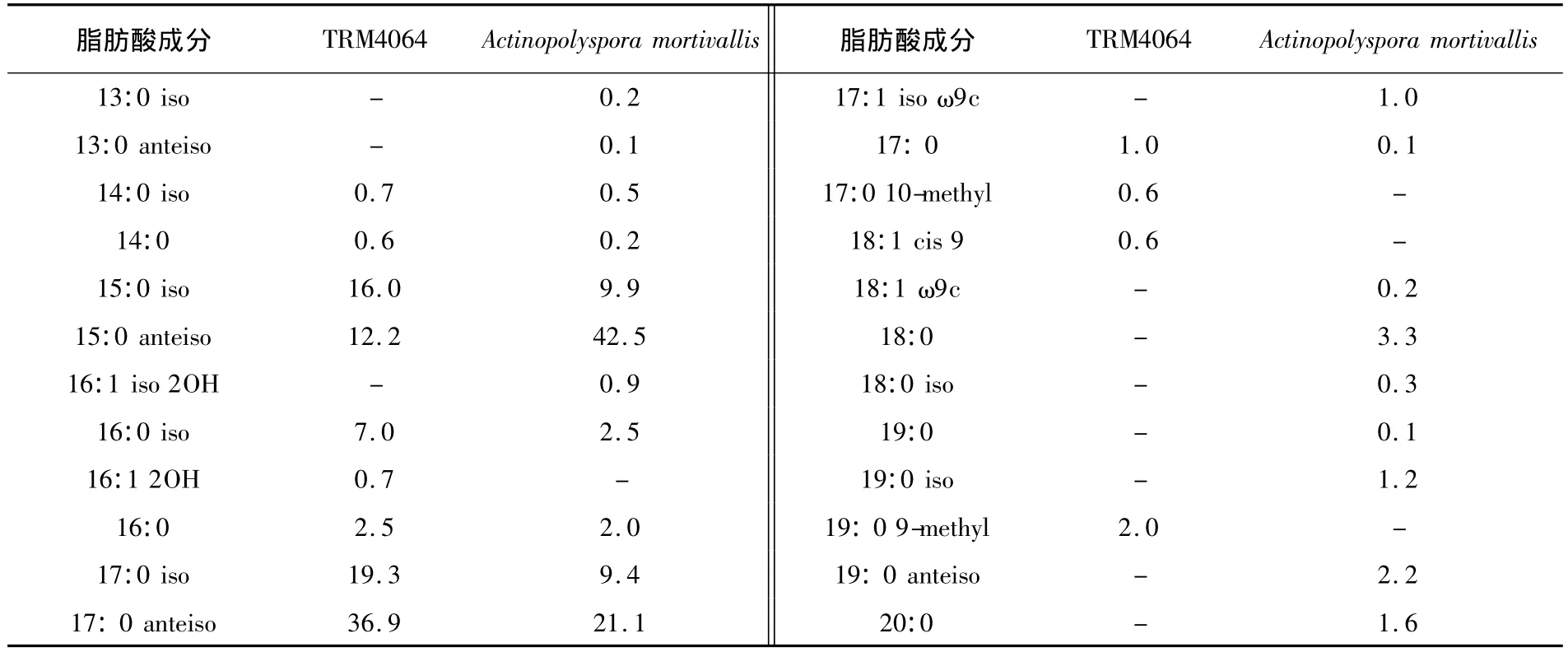
表2 菌株TRM4064与Actinopolyspora mortivallis的脂肪酸组成成分比较Table 2 Fatty acid profiles of strain TRM4064 and Actinopolyspora mortivallis
菌株TRM4064在全细胞水解糖、醌型、磷脂和脂肪酸组分分析上具有Actinopolyspora菌属的典型特征,且存在一定的差异。因此,在细胞化学水平上支持菌株TRM4064为Actinopolyspora菌属的一个新种。
2.3 16S rRNA系统发育分析与 DNA-DNA杂交
菌株TRM4064的16S rRNA基因序列测定结果表明,序列长度大约1 500 bp。采用EzTaxon server 2.1工具比较其菌株的16S rRNA基因与有效命名物种的序列进行序列比对,菌株TRM4064与Actinopolyspora mortivallis的同源性为98.8%,同Actinopolyspora菌属另外有效命名的物种的相似性小于97%。因此,需要进行核酸杂交来验证其是否为新的物种。根据Wayne等[13]的描述,DNA杂交值低于70%可作为不同的种。根据杂交结果菌株TRM4064同Actinopolyspora mortivallis的杂交值为23.2%,表明菌株TRM4064是在Actinopolyspora属之内且不同于已知种的新物种。采用CLUSTALX软件与已知亲缘关系较近的物种进行序列比对,MEGA4.0软件进行相似性计算、进化距离矩阵计算、聚类分析和系统进化树的构建等系统发育分析,结果见图2。
菌株 TRM4064系统发育表明,菌株TRM4064同亲缘关系最近的Actinopolyspora菌属的物种以极高的自展值(>98%)聚类在一起,形成独立的大分支单元,这与EzTaxon server 2.1工具分析的结果相一致。因此,系统发育分析表明菌株 TRM4064是 Actinopolyspora菌属的一个成员。
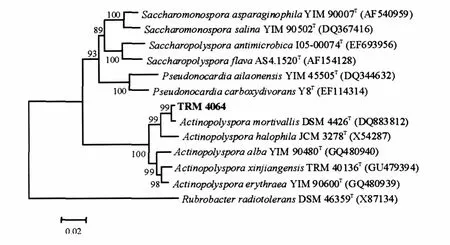
图2 菌株TRM4064同已知同源关系较近菌株的16S rRNA基因系统发育树Fig.2 Neighbour-joining tree based on 16S rRNA gene sequences,showing the phylogenetic relationships of the novel isolate TRM4064 and related taxa
2.4 (G+C)mol%测定
DNA的碱基组成具有种特异性,不受菌龄和外界因素的影响,是细菌分类和菌种鉴定的重要指标。根据实验结果分析,菌株TRM4064的(G+C)mol%是66.3%,当前 Actinopolyspora属已知有效命名种的(G+C)mol%范围在66.0% ~69.0%之间。因此,菌株 TRM4064的(G+C)mol%与Actinopolyspora属(G+C)mol%的范围一致。
3 讨论
多孢放线菌属Actinopolyspora最早被Gochnauer等[14]在1975年首次分离得到,即嗜盐多孢放线菌 Actinopolyspora halophila。当前 Actinopolyspora共含有5个有效命名的物种,即Actinopolyspora halophila、Actinopolyspora mortivallis、Actinopolyspora xinjiangensis、Actinopolyspora alba 和Actinopolyspora erythraea均分离自高盐环境。菌株TRM4064分离自高盐的盐湖环境,采用多相分类技术有效证明了该菌株为Actinopolyspora属的新成员,这一发现将进一步丰富Actinopolyspora属的物种多样性,从而为极端环境微生物资源的开发利用提供新的材料支撑。另外,云南大学微生物所对新疆、青海等地的高盐环境进行了放线菌资源的系统学研究,发现了大量嗜盐放线菌新物种[15]。这些极端环境中的嗜盐放线菌是依赖于一种或多种极端物化因子的极端生命形式,它们构成了地球生命形式的独特风景线,为更好地认知生命现象、发展生物技术提供了宝贵的材料源泉。
[1] 杨益芬.四川大英盐湖原核生物多样性初步研究[D].四川农业大学硕士学位论文,2006:19-20.
[2] Mata J A,Martínez-Cánovas M J,Quesada E.A detailed phenotypic characterization of the type strain of Halomonas species[J].Syst Appl Microbiol,2002,25(3):360-375.
[3] 徐丽华,李文均,刘志恒,等.放线菌系统学:原理、方法及实践[M].北京:科学出版社,2007.
[4] Collins M D,Pirouz T,Goodfellow M,et al.Distribution of menaquinones in actinomycetes and corynebacteria[J].J Gen Microbiol,1977,100(2):221-230.
[5] Groth I,Schumann P,Rainey F A,et al.Demetria terragena gen.nov.,sp.nov.,a new genus of actinomycetes isolated from compost soil[J].Int J Syst Bacteriol,1997,47(4):1129-1133.
[6] Sasser M.Identification of Bacteria by Gas Chromatography of Cellular Fatty Acids[M].DE:MIDI Inc,1990.
[7] Hasegawa T,Takizawa M,Tanida S.A rapid analysis for chemical grouping of aerobic actinomycetes[J].J Gen Appl Microbiol,1983,29(4):319-322.
[8] Mesbah M,Premachandran U,Whitman W B.Precise measurement of the G+C content of deoxyribonucleic acid by highperformance liquid chromatography [J].Int J Syst Bacteriol,1989,39(2):159-167.
[9] Ezaki T,Hashimoto Y,Yabuuchi E.Fluorometric DNA/DNA hybridization in microdilution wells as an alternative to membrane filter hybridization in which radioisotopes used to determine genetic relatedness among bacterial strain[J].Int J Syst Bateriol,1989,39(3):224-229.
[10]Hezayen F F,Tindall B J,Steinbüchel A,et al.Characterization of a novel halophilic archaeon,Halobiforma haloterrestris gen.nov.,sp.nov.,and transfer of Natronobacterium nitratireducens to Halobiforma nitratireducens comb.nov[J].Int J Syst Evol Microbiol,2002,52(Pt 6):2271-2280.
[11]Saitou N,Nei M.The neighbor-joining method:a new method for reconstructing phylogenetic tree[J].Mol Biol Evol,1987,4(4):406-425.
[12]Tang S K,Wang Y,Klenk H P,et al.Actinopolyspora alba sp.nov.and Actinopolyspora erythraea sp.nov.,isolated from a salt field,and reclassification of Actinopolyspora iraqiensis Ruan et al.1994 as a heterotypic synonym of Saccharomonospora halophile[J].Int J Syst Evol Microbiol,2011,61(Pt 7):1693-1698.
[13]Wayne L.G,Brenner D.J,Colwell R.R,et al.International Committee on Systematic Bacteriology.Report of the adhoc committee on reconciliation of approaches to bacterial systematics[J].Int J Syst Bacteriol,1987,37(4):463-464.
[14]Gochnauer M B,Leppard G,Komaratat P,et al.Isolation and characterization of actinopolyspora halophila,gen.sp.nov.,an extremely halophilic actinomycete[J].Can J Microbiol,1975,21(10):1500-1511.
[15]张永光,李文均,姜成林,等.嗜盐放线菌的研究进展[J].微生物学杂志,2002,22(4):45-48.