黄瓜转录因子CsCBF3的克隆及表达分析
2013-09-11苗永美娄群峰翁益群陈劲枫
宁 宇 苗永美,2 李 季 娄群峰 翁益群 陈劲枫*
(1南京农业大学园艺学院,作物遗传与种质创新国家重点实验室,江苏南京 210095;2安徽科技学院生命科学学院,安徽凤阳 233100;3美国威斯康星大学园艺系,威斯康星麦迪逊 53706)
黄瓜转录因子CsCBF3的克隆及表达分析
宁 宇1苗永美1,2李 季1娄群峰1翁益群3陈劲枫1*
(1南京农业大学园艺学院,作物遗传与种质创新国家重点实验室,江苏南京 210095;2安徽科技学院生命科学学院,安徽凤阳 233100;3美国威斯康星大学园艺系,威斯康星麦迪逊 53706)
利用RT-PCR技术从黄瓜中克隆了植物低温信号转导途径中的关键转录因子C-repeat-Binding Factor3,将其命名为CsCBF3,GenBank登录号为JQ900769。CsCBF3基因开放阅读框全长为615 bp,编码204个氨基酸。进化树分析表明,CsCBF3隶属于CBF基因家族,与葡萄CBF3蛋白亲缘关系最近,而与黑麦草、水稻CBF3蛋白亲缘关系较远。生物信息学分析结果表明,CsCBF3编码的蛋白包含保守的AP2dNA结合域,N端的核定位信号区和C端的酸性氨基酸富集区,且该蛋白中的一些磷酸化位点及二级结构在与DNA互作过程中发挥重要作用。荧光定量PCR检测结果显示低温可诱导CsCBF3的表达,且其表达量在迅速达到峰值后又降低,说明CsCBF3是一个快速响应基因,推测其在黄瓜耐冷过程中起着重要的作用。
黄瓜;低温;CBF3;基因克隆;表达分析
黄瓜(Cucumis sativus L.)属葫芦科甜瓜属,是一种深受大众喜爱的世界性蔬菜。黄瓜原产于喜马拉雅山南麓的印度北部地区(陈劲枫,2008),喜温暖湿润的气候,低温会使黄瓜的产量和品质下降,最终造成严重的经济损失,因此,提高黄瓜耐冷性具有非常重要的实践意义。目前的研究多利用一氧化氮、多胺、水杨酸等外源物质来提高黄瓜在低温环境下的适应能力(徐洪雷和于广建,2007;Zhang et al.,2009;王磊 等,2010),而有关黄瓜耐冷分子调控机理的报道则较少。
CBF3(C-repeat-Binding Factors)属于 A P2/EREBP(Ethylene-responsive element binding protein)基因家族,是一种受低温特异诱导的转录因子,可激活下游多个效应基因表达,从而提高植物对低温、干旱等多种逆境胁迫的抗性(Yang et al.,2005)。该转录因子首先在对拟南芥的研究中被发现(Yamaguchi-Shinozaki & Shinozaki ,1994),随后在油菜(Jaglo et al.,2001)、水稻(Dubouzet et al.,2003)等作物上也分离克隆出了CBF3基因,且证明其可被低温环境所诱导。由于CBF3可调控多个相关基因来提高植物的抗性,因此克隆CBF3基因和利用基因工程技术为改良植物耐冷品质提供了更好的选择,目前已有研究表明CBF3可显著提高转基因植株如高羊茅(Zhao et al.,2007)、黑麦草(Li et al.,2011)、烟草(Yang et al.,2011)等作物的耐冷性。而在黄瓜中,该基因的功能和分子作用机制还尚未明确。
本试验从黄瓜耐冷品种Chipper中克隆到了黄瓜CBF3基因,对其编码的氨基酸序列进行了生物信息学分析,为明确基因功能奠定了基础。同时利用荧光定量技术研究了低温胁迫与黄瓜CBF3表达量之间的关系,以期从一定程度上揭示黄瓜耐冷的 分子调控机理。
1 材料与方法
1.1 材料
以南京农业大学葫芦科作物遗传与种质创新实验室保存的美国生态型黄瓜Chipper为试材。试验于2011年秋季在南京农业大学进行,选取饱满的种子浸种催芽,将露白的种子播于育苗穴盘中,在昼温25℃,夜温18℃,光照12 h的人工气候箱中培养。当幼苗长至2~3片真叶时,将幼苗放置于4℃的人工气候箱中,分别于处理0、2、4、8、12、24 h后取幼嫩叶片,置于液氮中保存备用。
1.2 方法
1.2.1 DNA、RNA的提取及cDNA第一链的合成 黄瓜DNA的提取采用SDS-CTAB法(江彪,2011)。按Trizol法提取总RNA,并参照TAKARA公司的DNase I试剂盒说明消除微量DNA污染。取纯化后的总RNA用于cDNA第一链的合成(PrimeScriptTM1st Strand cDNA Synthesis Kit,TAKARA)。
1.2.2 CsCBF3基因的克隆 以拟南芥CBF3基因(AEE85065)的核酸序列为信息探针,在黄瓜基因组数据库(http://cucumber.genomics.org.cn/page/cucumber/index.jsp)中进行Blast同源搜索,找到黄瓜CBF3基因。根据开放阅读框的上下游末端设计引物,引物序列CBF3-F:5′-ATGTCTTCTTCGTCTTCAAGCT-3′;CBF3-R:5'-TCACTCATGACTCCATAACGAC-3′。 引 物委托上海英骏生物科技公司合成。
分别以基因组DNA和cDNA为模板进行扩增,PCR扩增程序为:94℃预变性5min;94℃变性30 s,55℃退火30 s,72℃延伸1min,35个循环;72℃7min。PCR产物在1%琼脂糖凝胶中电泳,按凝胶回收试剂盒说明书回收目的片段,并将其克隆在pMD19-T载体上。热激法转化大肠杆菌DH5α感受态细胞,挑经Amp抗性筛选和菌液PCR检测鉴定过的阳性克隆送交南京金斯瑞公司测序。
1.2.3 CsCBF3蛋白的生物信息学分析 利用DNAman6.0软件对CsCBF3编码的氨基酸序列进行比对和分析。系统进化树由MEGA5.0构建,采用邻接法(Neighbor Joiningmethod)作图,重复计算次数设为1000。分别使用Expasy蛋白质组分析工具中的ProtParam软件(http://web.expasy.org/protpara)和 COR软件(http://npsa-pbil.ibcp.fr/cgi-bin/npsa_automat.pl?page=npsa_gor4.html)对蛋白质进行理化性质分析和二级结构预测。蛋白质翻译后修饰的磷酸化位点分析采用在线软件Netphos2.0 Server完成(http://www.cbs.dtu.dk/services/NetPhos/)。利用在线工具Smart分析蛋白质结构域(http://smart.embl-heidelberg.de/)。
1.2.4 实时定量PCR检测CsCBF3基因的表达 提取低温胁迫下不同时间段内(0、2、4、8、12、24 h)的黄瓜叶片的总RNA,经DNase I消化后反转录为cDNA第一链,取适量作为模板。
根据已获得的CsCBF3的开放阅读框全长序列设计实时定量PCR引物,引物序列qCBF3-F:5'-CTCAACCAACGACCACCT-3';qCBF3-R:5'- CGCAAACCCATTTACCAT-3′, 扩 增 片 段长度为128 bp。选取黄瓜看家基因EF1a作为内参,引物序列EF1a-F:5'-ACTGTGCTGTCCTCATTATTG-3′;EF1a-R:5'- AGGGTGAAAGCAAGAAGAGC-3′。
试验在Bio-Rad iCycler iQ实时定量PCR 仪上进行,每个样品设3次重复。反应采用20 μL体系,包括1μL cDNA,上、下游引物各1μL,10μL iQTMSYBR®green Supermix(Bio-Rad),7μLddH2O。两步法扩增,具体程序为:95℃预变性2min,95℃变性5 s,60℃退火30 s,40个循环,然后进行融解曲线分析。
将正常条件下CsCBF3的表达量设为1,按照2-ΔΔCT法计算出基因的相对表达量,采用Excel软件绘图。
2 结果与分析
2.1 CsCBF3基因的克隆
以cDNA第一链为模板,用引物CBF3-F/CBF3-R扩增出了615 bp左右的目的片段(图1)。测序结果显示该片段与黄瓜基因组数据库中目的基因核酸序列的一致性为100%,表明已成功克隆到黄瓜CBF3基因,命名为CsCBF3,GenBank登录号为JQ900769。与黄瓜基因组数据库比对分析发现CsCBF3基因没有内含子;以黄瓜基因组DNA为模板也可扩增出同样大小的片段(图1),再次证明了该基因不含有内含子,这与已报道的拟南芥CBF3基因的结构特征相一致(Gilmour et al.,1998)。
2.2 CsCBF3氨基酸序列比对及进化树分析

图1 琼脂糖电泳检测PCR扩增产物
利用DNAman6.0对CsCBF3编码的氨基酸序列与其他物种的CBF3氨基酸序列进行同源比对分析。比对结果显示,黄瓜CBF3氨基酸序列与拟南芥(ABV27154)的相似性最高,达到43.70%,其次是葡萄(ACT45468)和辣椒(ADM73296),分别为41.84%、36.52%,而与茄子(AAS77819)、蒺藜苜蓿(ABG75914)、黑麦草(AAX57275)、水稻(AEW67332)、大麦(ABE02655)、玉米(NP001105651)和大豆(ACA63936)等物种的CBF3蛋白相似性较低(图2)。由MEGA5.0构建的系统进化树表明黄瓜CBF3与葡萄CBF3的亲缘关系最近,在进化上属同一分支,而与水稻、黑麦草的CBF3蛋白的亲缘关系则较远(图3)。

图2 CsCBF3与其他物种CBF3蛋白氨基酸序列的同源性比对

图3 黄瓜及其他物种CBF3蛋白进化树分析
2.3 CsCBF3蛋白结构与功能分析
CsCBF3基因编码的多肽链包含204个氨基酸残基,等电点(PI)为8.42,不稳定系数为64.86,较高,符合其转录因子的特性。大量研究报道表明,转录因子的功能活性受到磷酸化修饰的调控(Ciceri et al.,1997),因此本试验对CsCBF3蛋白的磷酸化位点进行了预测和分析。如图4所示,分值大于0.5的磷酸化位点有:丝氨酸磷酸化位点12个,苏氨酸磷酸化位点2 个,酪氨酸磷酸化位点1个,这些磷酸化位点大部分集中于氨基酸序列100~150范围内(图4)。

图4 CsCBF3蛋白序列磷酸化位点分析
在蛋白二级结构上,CsCBF3基因编码的肽链具有3个结构域,即N端信号区域(第1~35位)、AP2/EREBPdNA结合域(第36~98位)和C端酸性激活区域(第99~204位)。其中在第20~32位序列区段内富含多个碱性氨基酸(精氨酸R和赖氨酸K),为该转录因子的核定位信号区NLS(图2),转录因子CsCBF3进入细胞核的过程受到该区域的调控。在AP2dNA结合域内存在着3个β折叠和1个α螺旋(图5),这些结构在参与识别各类顺式作用元件及同转录因子或DNA的相互作用中起关键作用(Allen et al.,1998;Kanaya et al.,1999)。

图5 CsCBF3蛋白二级结构分析
2.4 CsCBF3基因在不同逆境胁迫下的表达分析
利用荧光定量PCR技术检测了CsCBF3在低温胁迫下表达量的变化情况,结果表明(图6),CsCBF3在低温处理后的表达量显著高于处理前,证明低温可诱导CsCBF3的表达。当黄瓜受到低温胁迫后,CsCBF3的表达量迅速增加并在2 h时达到峰值,约为对照的56倍,在随后4~8 h内表达量下降,而后又有所上升。这种变化规律说明CsCBF3可能在快速提高黄瓜对低温胁迫抵抗能力的过程中起着非常重要的作用。
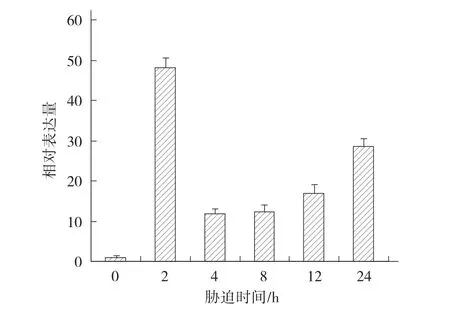
图6 荧光定量PCR检测CsCBF3在低温胁迫下的表达
3 讨论
本试验通过RT-PCR技术首次克隆到了黄瓜CBF3基因(JQ900769),并从生物信息学的角度对其编码的氨基酸序列进行了分析,为该基因的功能鉴定奠定了基础。CsCBF3蛋白不稳定系数高,不含跨膜结构域和信号肽,推测其可能在细胞质内发挥其转录因子的作用。CsCBF3蛋白具有12个潜在的磷酸化位点,说明其可能在翻译后通过磷酸化和去磷酸化修饰而完成该转录因子与DNA结合的过程(Medina et al.,1999),其中第37位的酪氨酸、第67位的丝氨酸位于AP2基因家族保守的AP2/EREBPdNA结合域内,因此可推断这两个氨基酸残基在蛋白与DNA结合过程中发挥着非常重要的作用。同源比对结果显示,CsCBF3与拟南芥CBF3相似度较高,表明其可能具有相同的分子生物学作用,但还需要进一步的试验验证。
转录因子CBF3在植物低温信号转导过程中发挥着重要的作用。当低温胁迫发生时,CBF3基因被诱导激活,与CRT/DRE顺式作用元件特异结合,激活下游多个启动子区域含有该元件的效应基因表达,从而提高植物对低温及其他胁迫的抵抗能力(Liu et al.,1998;Fowler & Thomashow,2002;Gilmour et al.,2004)。水稻上的研究表明,CBF3是一个快速响应基因,可在短时间内提高植株的耐冷性,当胁迫时间过长时,CBF3的表达量会因受到MYBS3基因的抑制而降低,同时MYBS3表达量逐渐升高,提高了水稻对长期低温环境的耐受性(Su et al.,2010)。本试验中荧光定量PCR检测结果表明,CsCBF3的表达量一般在胁迫2 h后就达到峰值,证明其是一个快速响应基因。随后其表达量先是降低,而后又有所升高,这与水稻CBF3基因的表达模式略有不同,推测在黄瓜中可能并不存在一个特异的基因专门负责调控长时间的低温胁迫,而是由CsCBF3在整个胁迫过程中发挥作用。
此外,笔者还成功构建了由CaMV35S启动子驱动的植物表达载体pCAMBIA1304-CsCBF3,对黄瓜的遗传转化工作正在进行中,以期能进一步对CsCBF3的功能进行验证,同时也希望能够获得对低温有较高耐受性的转基因黄瓜植株,为黄瓜耐冷育种提供新的种质资源材料。
陈劲枫.2008.基于种间渐渗的甜瓜属野生优异基因发掘研究.中国瓜菜,(6):1-3.
江彪.2011.甜瓜属Ty1-copia类逆转座子分析及其对异源四倍体遗传变异的影响研究〔博士论文〕.南京:南京农业大学.
王磊,刘兴旺,金宝燕,陈明远,张雷,任华中.2010.水杨酸提高黄瓜低温耐受性的生理及CATmRNA基因响应机制.华北农学报,25(3):92-96.
徐洪雷,于广建.2007.一氧化氮(NO)对黄瓜低温胁迫的缓解作用.东北农业大学学报,38(5):606-608.
Allenmd,Yamasskai K,Ohme-Takagim,Tatenom,Suzukim.1998.A novelmode ofdNA recognition by a beta-sheet revealed by the solution structure of thegCC-box bindingdomain in complex withdNA.EMBO J,17:5484-5496.
Ciceri P,Gianazza E,Lazzari B,Lippolig,Genga A,Hoscheckg,Schmidt R J,Viotti A.1997.Phosphoyrlation of opaque Ⅱ changesdiurnally and impacts itsdNA binding activity.Plant Cell,9:97-108.
Dubouzel Jg,Sakuma Y,Ito Y,Kasugam,Dubouzet Eg,Miura S,Sekim,Shinozaki K,Yamaguchi-Shinozaki K.2003.OsDREBgenes in rice(Oryza sativa L. ) encode transcription activators that function indrought,high salt and cold responsivegene expression.Plant Journal,33:751-763.
Fowler S,Thomashowm F.2002.Arabidopsis transcriptome profiling indicates thatmultiple regulatory pathways are activatedduring cold acclimation in addition to thECBF cold response pathway.Plant Cell,14:1675-1690.
Gilmour S J,Zarkadg,Stoekinger E J,Salazarm P,Houghton Jm,Thomashowm F.1998.Low temperature regulation of the Arabidopsis CBF family of AP2 transcriptional activators as an early step in cold induced CORgene expression.Plant J,16:433-442.
Gilmour S J,Fowler Sg,Thomashowm F.2004.Arabidopsis transcriptional activators CBF1,CBF2,and CBF3 havematching functional activities.Plantmol Biol,54:767-781.
Jaglo K R,Kleff S,Amundsen K L,Zhang X,Haake V,Zhang J Z,Deits T,Thomashowm F.2001.Componets of the Arabidopsis C-repeat/dehydration-responsive element binding factor cold-response pathway arEConserved in Brassica napus and other plant species.Plant Phyciology,127:910-917.
Kanaya E,Nakajima N,Morikawa K,Okada K,Shimura Y.1999.Characterization of the transcriptional activator CBF1 from Arabidopsis:evidence for colddenaturation in regions outside of thedNA bindingdomain.J Biol Chem,274:16064-16076.
Li X,Cheng X X,Liu J,Zeng Hm,Han L B,Tang W.2011.Heterologous expression of the ArabidopsisdREB1A/CBF3gene enhancesdrought and freezing tolerance in transgenic Lolium perenne plants.Plant Biotechnol Rep,5:61-69.
Liu Q,Kasugam,Sakuma Y,Abe H,Miura S,Yamaguchi-Shinozalti K,Shinozaki K.1998.Two transcription factors,DREB1 anddREB2,with an EREBP/AP2dNA bindingdomain separate two cellular signal transduction pathways indrought and lowtemperature responsivegene expression,respectively,in Arabidopsis.Plant Cell,10:1391-1406.
Medina J,Bgaruesm,Terol J,Pérez-Alonsom,Salinas J.1999.The Arabidopsis CBFgene family is composed of threegenes encoding AP2domain containing proteins whose expression is regulated by low temperature but not by ABA ordehydration.Plant Physiology,119:463-470.
Su C F,Wang Y C,Hsieh T H,Lu C A,Tseng T H,Yu Sm.2010.A novelmYBS3-dependent pathway confers cold tolerance in rice.Plant Physiology,153:145-158.
Yamaguchi-Shinozaki K,Shinozaki K.1994.A novel cis-acting element in an Arabidopsisgene is involved in responsiveness todrought,low-temperature or high-salt stress.Plant Cell,6:251-264.
Yang T W,Zhang L J,Zhang Tg,Zhang H,Xu S J,An L Z.2005.Transcriptional regulation network of cold-responsivegenes in higher plants.Plant Science,169:987-995.
Yang S,Tang X F,Ma N N,Wang L Y,Meng Q W.2011.Heterology expression of the sweet pepper CBF3genEConfers elevated tolerance to chilling stress in transgenic tobacco.Journal of Plant Physiology,168:1804-1812.
Zhang W P,Jiang B,Li Wg,Song H,Yu Y S,Chen J F.2009.Polyamines enhancEChilling tolerance of cucumber(Cucumis sativus L.)throughmodulating antioxidative system.Scientia Horticulturae,122:200-208.
Zhao J S,Ren W,Zhid Y,Wang L,Xiagm.2007.ArabidopsisdREB1A /CBF3 bestowed transgenic tall fescue increased tolerance todrought stress.Plant Cell Rep,26:1521-1528.
Cloning and Expression Analysis of Transcription Factor CsCBF3gene from Cucumber
NING Yu1,MIAO Yong-mei1,2,LI Ji1,LOU Qun-feng1,WENG Yi-qun3,CHEN Jin-feng1*
(1State Key Laboratory of Cropgenetics andgermplasm Enhancement,College of Horticulture,Nanjing Agricultural University,Nanjing210095,Jiangsu,China;2College of Life Science,Anhui Science and Technology University,Fengyang233100,Anhui,China;3Horticulturedepartment,University of Wisconsin,Madison53706,USA)
In order todetermine its role in cold response in cucumber (Cucumis sativus L.),CsCBF3 was cloned from Chipper using RT-PCRmethod and itsgenBank accession number was JQ900769.CsCBF3 had an open reading frame of615 bp,which was supposed to encode a protein of204 amino acids.Phylogenetic tree analyses showed that CsCBF3 belonged to CBFgene family and had close relationship with CBF3 from Vitis amurensis,while had far relationship with CBF3 fromLolium perenne and Oryza sativa.Bioinformatics analysis showed that CsCBF3 protein was consisted of AP2dNA bindingdomain,which included amino acid residues,nuclear localization signal region and acidic activationdomain,and some phosphorylation sites and protein secondary structuresmay play important roles in process of interactions between proteins anddNA.Quantitative real-time PCR results showed that expression of CsCBF3 could be induced by low temperature,and the level reached to peak very fast and thendecreased.These results suggested that CsCBF3 was a rapid responsegene and played an important role in resistance to low temperature in cucumber.
Cucumber;Low temperature;CBF3;GenECloning;Expressive analysis
S642.2
A章编号:1000-6346(2013)06-0030-07
2012-09-17;接受日期:2012-11-23
国家“973”计划项目(2009CB119001),国家自然科学基金重点项目(30830079),江苏省农业科技自主创新基金项目(CX(11)1002)
宁宇,男,硕士研究生,专业方向:蔬菜生物技术,E-mail:2010104050@njau.edu.cn
*通讯作者(Corresponding author):陈劲枫,男,教授,博士生导师,专业方向:蔬菜遗传育种与生物技术,E-mail:jfchen@njau.edu.cn
