团头鲂微卫星多重PCR体系的建立及应用
2013-08-07罗伟高泽霞曾聪邓伟易少奎钱雪桥王卫民
罗伟,高泽霞,曾聪,邓伟,易少奎,钱雪桥,王卫民
(1.华中农业大学水产学院农业动物遗传育种与繁育教育部重点实验室农业部淡水生物繁育重点实验室,湖北武汉430070; 2.广东海大集团股份有限公司,广东广州511400)
团头鲂微卫星多重PCR体系的建立及应用
罗伟1,高泽霞1,曾聪1,邓伟1,易少奎1,钱雪桥2,王卫民1
(1.华中农业大学水产学院农业动物遗传育种与繁育教育部重点实验室农业部淡水生物繁育重点实验室,湖北武汉430070; 2.广东海大集团股份有限公司,广东广州511400)
利用团头鲂Megalobrama amblycephala20个微卫星标记组合建立了6个微卫星多重PCR体系,包括2个四重PCR和4个三重PCR体系。利用构建的6个微卫星多重PCR体系评估了团头鲂淤泥湖群体的遗传多样性,结果表明,平均等位基因数为5.8,平均期望杂合度和平均观测杂合度分别为0.72和0.77,平均多态性信息含量为0.601,20个位点中有9个位点显著偏离Hardy-Weinberg平衡 (P<0.05)。本研究中筛选出的多重PCR组合为团头鲂的群体遗传结构分析和亲子鉴定等研究提供了技术基础。
团头鲂;微卫星;多重PCR;遗传多样性
微卫星DNA(microsatellite)由2~6个核苷酸为核心序列的串联重复而成,其数量巨大,随机分布于整个基因组。自 20世纪 80年代被发现以来[1],微卫星标记以其分布广泛、共显性遗传、多态性高等特点,被广泛应用于个体鉴定、群体遗传学,以及进化和基因组作图等领域[2-4]。目前,应用微卫星标记对动植物进行群体遗传和亲子鉴定等的研究,通常需要对多个群体大量个体的多个微卫星位点进行PCR扩增和检测,操作繁琐、工作量大、试验成本高。1988年,Chamberlian等[5]首次提出在同一PCR反应体系中加入多对微卫星引物,同时进行多个目标序列的扩增,即多重 PCR (multiplex PCR)。该技术可一次扩增多个靶位点,同时实现多个位点的基因型鉴别,使基因分型变得简捷,提高了检验效率,并且多重PCR技术同时扩增两条以上的靶基因片断,出现假阳性的概率较常规PCR小得多,有效地减少了假阳性现象。目前,多重PCR技术已经在多个物种的群体遗传多样性分析[6-7]、谱系分析[8-9]和 DNA检测[10]中被广泛应用。在水产领域,对棕鳟[11]、大菱鲆[12]和中国明对虾[13]等已建立了多重PCR体系。
团头鲂Megalobrama amblycephala隶属于鲤形目Cypriniformes、鲤科Cyprinidae、鲂属Megalobra-ma,俗称武昌鱼,是中国特有的经济名贵鱼类之一,原产于长江中下游的通江湖泊,由于其生长快、抗病力强、味道优美、营养丰富,自20世纪60年代人工繁殖取得成功以来,在中国各地得到广泛推广,目前团头鲂已成为了中国最重要的池塘混养鱼类之一。几十年来,由于原产地野生资源被过度捕捞以及各地广泛移植和人工近亲繁殖,团头鲂的种质资源不断衰退和混杂[14]。为了保护团头鲂野生种质资源并获得更好的良种,国家现代农业产业技术体系——大宗淡水鱼类产业技术研发中心启动了 “团头鲂种质资源与育种”项目。为实现团头鲂分子育种,目前已有不少有关其微卫星标记开发的报道[15-18],但尚未见其多重PCR体系的开发。本研究中,根据本课题组开发的多态性微卫星标记,作者优化筛选了团头鲂多重微卫星PCR扩增体系,并用于评估了团头鲂淤泥湖野生群体的遗传多样性。
1 材料与方法
1.1 材料
2009年6月,采集团头鲂湖北公安淤泥湖野生群体,随机选取40尾,剪取鳍条,用体积分数为95%的乙醇固定,于-20℃下保存备用。
1.2 方法
1.2.1 团头鲂基因组DNA的提取 团头鲂个体基因组DNA按照Tangs等[16]所用的蛋白酶K/SDS消化、苯酚-氯仿抽提法提取,将得到的DNA用双蒸水稀释成100 ng/μL,于-20℃下保存备用。
1.2.2 团头鲂多态性微卫星的准备 本试验中选用的团头鲂多态性微卫星标记来自华中农业大学农业动物遗传育种与繁育教育部重点实验室已开发的团头鲂EST-SSR标记[19],基本信息见表1。

表1 用于多重PCR体系构建的微卫星位点的基本信息Tab.1 Information of the SSR loci for establishment ofmultiplex PCR system
1.2.3 团头鲂微卫星多重PCR体系的筛选和优化
在单个微卫星座位PCR的优化条件基础上,通过8%聚丙烯酰胺凝胶电泳 (PAGE)结合硝酸银染色进行多重PCR组合的筛选和优化。步骤如下:
1)选取带型清晰、杂带少的微卫星位点;
2)利用 OLIGO 7软件 (http://www.oligo. net)检测不同位点的引物序列间是否存在较强的互补,并选择互补较低的引物同时进行扩增;
3)确认多个位点的产物大小两两之间有明显差异,一般大于20 bp;
4)挑选引物互不干扰、产物片段差异较大的引物筛选二重PCR;
5)在条带清晰、效果良好的二重PCR体系中添加另一对引物筛选三重PCR体系;
6)按类似方法筛选四重PCR体系。
多重PCR扩增体系为20μL,包括100 ng基因组 DNA,1×PCR缓冲液,1.5 mmol/L Mg2+,1 UTaq酶,0.2 mmol/L dNTPs,多重PCR扩增体系内各位点上、下游引物各为0.25μmol/L。PCR反应条件为:94℃下预变性5 min;94℃下变性45 s,退火45 s,72℃下延伸1 min,共进行30个循环;最后在72℃下延伸7 min,于4℃下保存备用。反应产物用于聚丙烯酰胺凝胶电泳分型。
1.3 数据处理
用PopGene 32软件计算等位基因数 (Na)、有效等位基因数 (Ne)、观测杂合度 (Ho)、期望杂合度 (He),多态性信息含量 (PIC)。用Genepop on the web(http://genepop.curtin.edu.au/genepop_op1.html)进行Hardy-Weinberg平衡检验,其P值用PHWE表示。
2 结果
2.1 多重PCR反应体系的构建
设计的引物组合经过筛选后共获得了4个三重PCR和2个四重PCR体系,体系的引物组合、产物大小、退火温度详见表2。聚丙烯酰胺凝胶电泳结果见图1。
2.2 群体遗传多样性
团头鲂淤泥湖野生群体的遗传多样性指数见表3。在野生群体中,平均等位基因为5.8,平均观测杂合度和平均期望杂合度分别为0.77和0.72,平均多态性信息含量为0.601,20个位点中有3个位点显著偏离Hardy-Weinberg平衡 (P<0.05),有6个位点极显著偏离Hardy-Weinberg平衡 (P<0.01)。
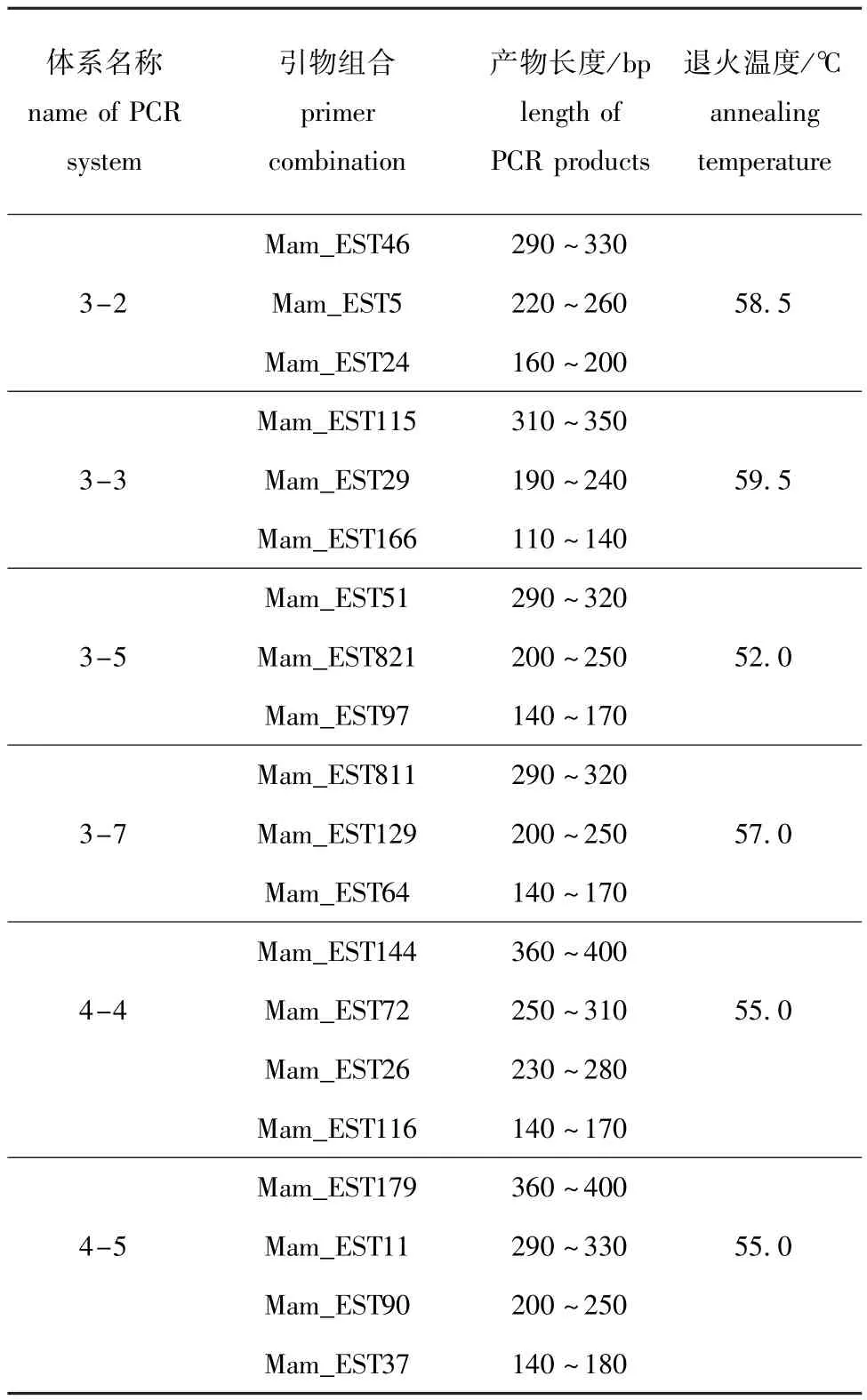
表2 团头鲂多重微卫星PCR体系Tab.2 The systems of m icrosatellite multiplex PCR for bluntsnout black bream Megalobrama amblycephala
3 讨论
微卫星多重PCR是在同一PCR体系中同时用多个引物对同一模板进行扩增,同时检测多个微卫星位点的基因型,可简化试验操作步骤和节省试验材料,从而提高检测效率[20]。在本试验中,仅用6个PCR反应体系就完成了20个微卫星位点的分型,能够在短时间内实现团头鲂群体的遗传多样性分析,充分体现了多重PCR操作简便、节省材料和高效性的特点。
为了确保多个目的片段在同一个PCR反应体系中同时有特异性扩增,微卫星引物组合的选择和PCR反应条件的优化是关键。引物间的兼容性(复性温度、引物二聚体)、引物浓度、PCR缓冲液的成分和浓度,以及PCR反应体积和反应程序等是影响多重PCR扩增效率的主要因素。有学者认为,引物间的兼容性和引物浓度比例是两个核心因素[20-21];也有一些学者认为,PCR缓冲液的成分影响多重PCR的扩增效果,而反应体积、循环次数对其扩增效果影响较小[22]。本试验中,主要针对引物组合、退火温度和反应体积的大小进行优化,结果发现,引物间的配对是最重要的影响因素,要选择的引物间不能发生二聚体,浓度也不能过大,并且他们的最佳退火温度要相近 (温差≤5℃);此外,在同一个多重PCR反应中存在多对引物间的竞争,多重PCR中各种化学成分 (Taq酶, dNTP、DNA模板)都应高于单重PCR,同时还要降低复性温度,并增加复性和延伸时间。
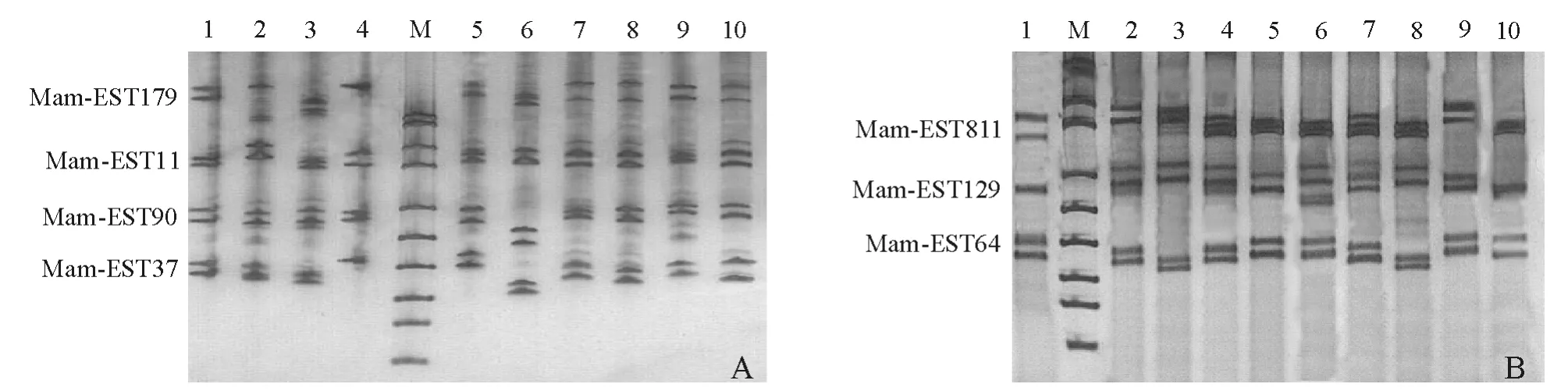
图1 体系4-5(A)和3-7(B)聚丙烯酰胺凝胶电泳结果Fig.1 The PAGE picture of 4-5(A)and 3-7(B)
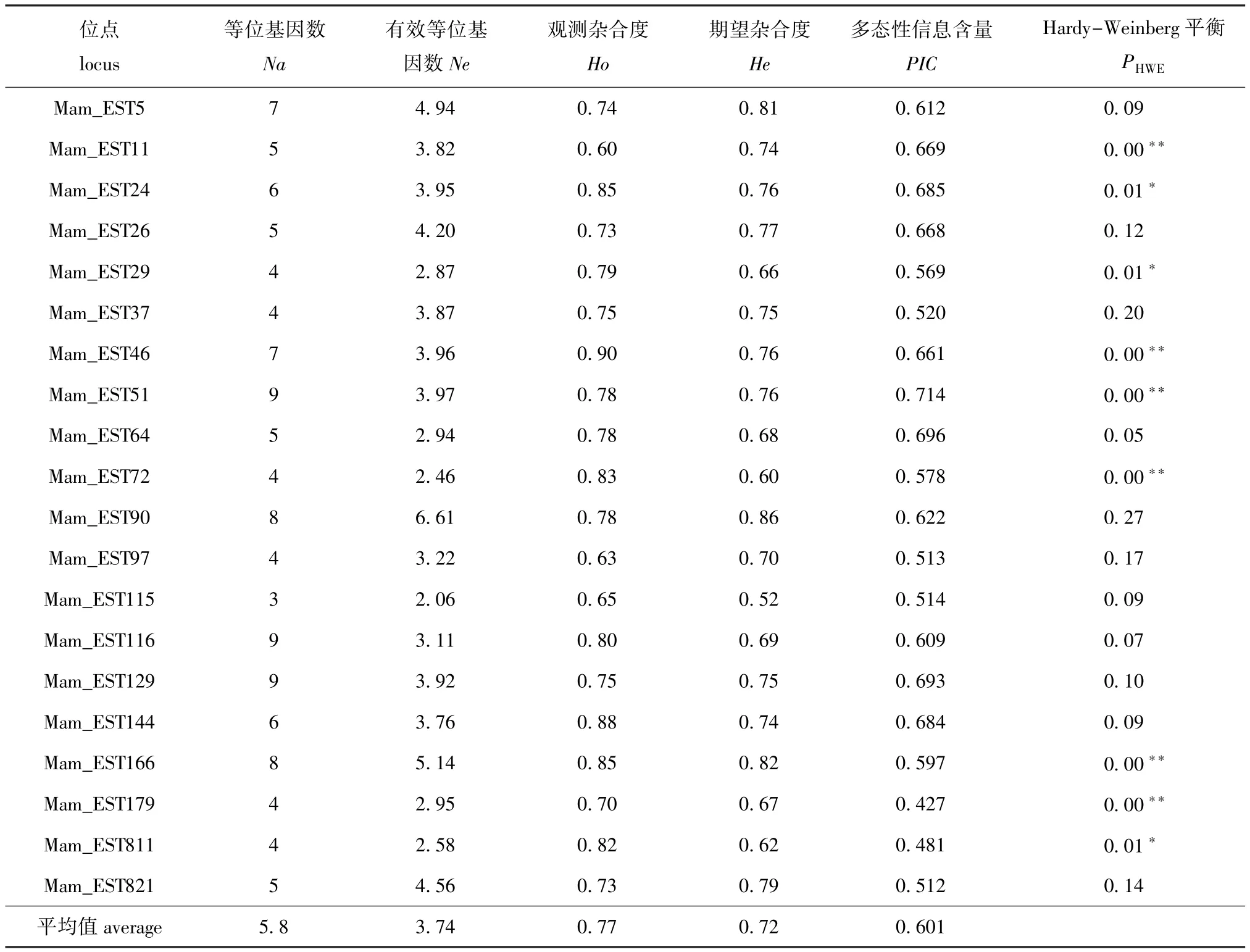
表3 淤泥湖团头鲂野生群体各SSR位点的信息Tab.3 Basic information of the SSR lociof bluntsnout black bream Megalobrama amblycephala population in Yuni Lake
生物群体的遗传多样性是生物多样性的核心和本质,也是评估生物资源状况的一个重要指标。一个种群遗传多样性越丰富,其环境适应能力就越强,生存和进化的潜力也就越大[23-24]。遗传多样性水平与生物的生长速度、抗病能力等重要经济性状密切相关[25]。一个种群的遗传多样性越丰富,从其中选出优良品种的可能性就越高,其优良性状保持下去的潜力也就越大。本研究中采用20对Mam-EST标记分析野生团头鲂遗传多样性水平,发现其遗传多样性处于较低水平,该研究结果与其他学者的结果相似,张德春[26]运用RAPD技术研究淤泥湖和梁子湖团头鲂,认为淤泥湖和梁子湖团头鲂的遗传多样性均比较低;边春媛等[27]用mtDNA控制区的RFLP研究了北方人工养殖的3个团头鲂群体,只发现2种单倍型,据此也认为其遗传多样性比较低,李弘华[28]使用mtDNA变异序列对梁子湖、鄱阳湖和淤泥湖3个野生群体团头鲂进行了遗传多样性和遗传结构分析,结果在3个原产地的团头鲂群体中仅发现3个变异位点和5个单倍型,也认为遗传多样性水平较低。与同属的鱼相比,李思发等[29]通过线粒体DNA控制区分析,团头鲂的多态性低于三角鲂和广东鲂。团头鲂的遗传多样性水平较低主要是由于其生长环境分布狭窄,仅限于长江中下游的通江湖泊,栖息环境相似,生境多态性不高,以致团头鲂进化中遗传分化不明显。本研究中,在野生群体的20个位点中,有9个位点显著偏离 Hardy-Weinberg平衡,这与李杨[30]的研究结果相符。该现象在多种水产动物中出现,如拟穴青蟹[31]、青虾[32]、日本蟳[33]等,说明群体中纯合子个体所占的比例较大,普遍存在杂合子缺失现象,这可能是造成群体偏离 Hardy-Weinberg平衡的主要原因。作者认为,造成多位点杂合子缺失的主要原因是由于近年来人为活动的加剧,造成团头鲂生境破碎,其繁殖受到干扰,致使团头鲂群体内部隔离;同时,人工繁殖 (特别是近亲繁殖)的子代放养或人工养殖的个体逃逸到湖泊中,打破了湖泊中原有的平衡。
本研究中筛选出的微卫星多重PCR体系不仅可以应用于团头鲂群体的遗传多样性分析和群体遗传关系研究,还可为亲子鉴定平台的建立以及分子标记辅助育种等方面的研究提供技术基础。
[1] Tautz D.Hypervariability of simple sequences as a general source for polymorphic DNA markers[J].Nucleic Acids Res,1989,17 (16):6463-6471.
[2] 阮晓红.大菱鲆Turbot微卫星标记的筛选与应用[D].青岛:中国海洋大学,2009.
[3] 徐浩,鲁翠云,孙效文.利用164个微卫星标记分析镜鲤家系的遗传多样性和经济性状[J].大连海洋大学学报,2013,28 (3):247-252.
[4] 董世瑞,孔杰,张天时,等.中国对虾微卫星家系鉴定的模拟分析与应用[J].水生生物学报,2008,32(1):96-101.
[5] Chamberlain JS,Gibbs R A,Rainer JE,et al.Deletion screening of the Duchennemuscular dystrophy locus viamultiplex DNA amplification[J].Nucleic Acids Res,1988,16(23):11141-11156.
[6] Sun W,Chen H,Lei C,et al.Study on population genetic characteristics of Qinchuan cows usingmicrosatellitemarkers[J].JGenet Genomics,2007,34(1):17-25.
[7] 周艳,朱庆,李亮.利用多重PCR分析丝羽乌骨鸡遗传多样性[J].湖北农业科学,2006,45(5):546-548.
[8] Porta J,Porta JM,Bejar J,et al.Development of a microsatellite multiplex genotyping tool for the fish gilthead seabream(Sparus aurata):applicability in population genetics and pedigree analysis [J].Aquacult Res,2010,41(10):1514-1522.
[9] Fishback A G,Danzmann R G,Sakamoto T,et al.Optimization of semi-automated microsatellitemultiplex polymerase chain reaction systems for rainbow trout(Oncorhynchusmykiss)[J].Aquaculture,1999,172(3/4):247-254.
[10] Mutiangura A,Greenberg F,Buler M G,et al.Multiplex PCR of three dinucleotide repeats in the Prader-Willi/Angelman critical region(15q11-q13):molecular diagnosis and mechanism of uniparental disomy[J].Hum Mol Genet,1993,2(2):143-151.
[11] Lerceteau-Kohler E,Weiss S.Development of a multiplex PCR microsatellite assay in brown troutSalmo trutta,and its potential application for the genus[J].Aquaculture,2006,258(1):641-645.
[12] 苗贵东,杜民,杨景峰,等.大菱鲆亲子鉴定的微卫星多重PCR技术建立及应用[J].中国海洋大学学报,2011,41(Z1): 97-106.
[13] 孔杰,高焕.中国明对虾微卫星三重PCR家系识别技术:中国,ZL 200510042402.9[P].2005-12-28.
[14] 李思发,蔡完其.团头鲂双向选育效应研究[J].水产学报, 2000,24(3):200-205.
[15] LiW T,Liao X L,Yu X M,et al.Isolation and characterization of polymorphicmicrosatellite loci in Wuchang bream(Megalobrama amblycephala)[J].Mol Ecol Notes,2007,7(5):771-773.
[16] Tangs J,Li S F,CaiW Q.Development ofmicrosatellitemarkers for blunt snout breamMegalobrama amblycephalausing 5'-anchored PCR[J].Mol Ecol Resour,2009,9(3):971-974.
[17] Gao Z,LuoW,Liu H,etal.Transcriptome analysis and SSR/SNP Markers information of the blunt snout bream(Megalobrama amblycephala)[J/OL].PLoSOne,2012,7(8):e42637.
[18] Luo W,Deng W,Yi S,et al.Characterization of 20 polymorphic microsatellites for blunt snout bream(Megalobrama amblycephala)from EST sequences[J].Conserv Genet Resour,2013,5(2): 499-501.
[19] Sambrook J,Russell DW.分子克隆试验指南[M].3版.黄培堂,王嘉玺,朱厚础,等译.北京:科学出版社,2001.
[20] 公维华,张宁波,程佳月,等.小型猪微卫星标记多重PCR体系的建立与应用[J].中国比较医学杂志,2009,19(2):21-25.
[21] 张毅,孙东晓,俞英,等.家养水牛30个微卫星标记的多重PCR体系建立及其多态性检测[J].遗传,2008,30(1):59-64.
[22] 黄银花,胡晓湘,徐慰倬,等.影响多重PCR扩增效果的因素[J].遗传,2003,25(1):65-68.
[23] 刘宏毅,黎明星,肖俊,等.叉尾斗鱼遗传多样性的RAPD分析[J].大连海洋大学学报,2012,27(2):158-161.
[24] 徐钢春,魏广莲,李建林,等.基于线粒体DNA D-loop序列分析养殖刀鲚与湖鲚的遗传多样性[J].大连海洋大学学报, 2012,27(5):448-452.
[25] 孙博.海湾扇贝分子标记的研究及应用[D].青岛:中国科学院海洋研究所,2003.
[26] 张德春.淤泥湖和梁子湖团头鲂遗传多样性的研究[J].三峡大学学报:自然科学版,2001,23:282-284.
[27] 边春媛,董仕,谭书贞.3个群体团头鲂mtDNA D-loop区段的限制性片断长度多态性分析[J].大连水产学院学报,2007, 22:175-179.
[28] 李弘华.淤泥湖、梁子湖、鄱阳湖团头鲂mtDNA序列变异及遗传结构分析[J].淡水渔业,2008(4):63-65.
[29] 李思发,朱泽闻,邹曙明,等.鲂属团头鲂、三角鲂及广东鲂种间遗传关系及种内遗传差异[J].动物学报,2002,48:339-345.
[30] 李杨.团头鲂三个野生群体的遗传结构分析及遗传图谱的构建[D].武汉:华中农业大学,2010.
[31] 舒妙安,周宇芳,朱晓宇,等.中国沿海拟穴青蟹群体遗传多样性的微卫星分析[J].水产学报,2011,35:977-984.
[32] 傅洪拓,乔慧,李法君,等.长江不同江段青虾的遗传多样性[J].水产学报,2010,34(2):204-211.
[33] 宋春妮,李健,刘萍,等.日本蟳4个野生群体遗传多样性的微卫星分析[J].水产学报,2011,35:985-991.
Establishment and application of microsatellite multilex PCR system for bluntsnout black bream Megalobrama ambl?ycephala
LUO Wei1,GAO Ze-xia1,ZENG Cong1,DENG Wei1, YIShao-kui1,QIAN Xue-qiao2,WANGWei-min1
(1.Key Lab of Agricultural Animal Genetics,Breeding and Reproduction,Ministry of Education,Key Lab of Freshwater Animal Breeding,Ministry of Agriculture,College of Fisheries,Huazhong Agricultural University,Wuhan 430070,China;2.Guangdong Haida Group Co.,Ltd.,Guangzhou 511400,China)
Six multiplex-PCR-sets including two tetraploid and four triplex combinations were developed from 20 microsatellitemarkers in bluntsnout black breamMegalobrama amblycephalabased on electrophoresis analysis of PCR products in denaturing polyacrylamide gels and visualization by silver-staining.Then genetic variability of these microsatellites was examined in wild population of bluntsnout black bream in Yuni Lake.The number of alleles at 20 loci was shown to vary from 3 to 9 with an average of5.8 alleles per locus,withmean polymorphic information content value of 0.601 and themean observed value of 0.77 and expected heterozygo site of 0.72,indicating that the bluntsnout black bream had middle level genetic diversity,and that there were 9 loci,significantly deviated from Hardy-Weinberg equilibrium(P<0.05)in 20 loci.These optimized multiplex-PCR-sets provide a technical basis for genetic diversity study and parentage test of bluntsnout black bream.
Megalobrama amblycephala;microsatellite;multiplex PCR;genetic diversity
S917.4
A
2095-1388(2013)05-0418-06
2013-02-18
国家自然科学基金资助项目 (31201988);现代农业产业技术体系建设专项 (CARS-46-05);中央高校基本科研业务费专项(2011PY023);广东海大集团团头鲂育种专项
罗伟 (1986-),男,博士研究生。E-mail:lwdjn@live.cn
王卫民 (1959-),男,教授。E-mail:wangwm@mail.hzau.edu.cn
