简并引物法克隆保加利亚乳杆菌β-N-乙酰氨基己糖苷酶基因
2013-07-06崔文明张书文逄晓阳李红娟吕加平
崔文明, 刘 鹭, 张书文, 逄晓阳, 李红娟, 吕加平
(中国农业科学院农产品加工研究所/农业部农产品加工综合性重点实验室,北京 100193)
β-N-乙酰氨基己糖苷酶(β-N-Acetylhexosaminidase,β-Hexcase)是在细菌、植物和动物中广泛存在的一种多糖水解酶,可以催化切除β-N-乙酰氨基己糖的非还原性氨基己糖残基,在细胞的正常分裂增殖中起着重要作用[1-3].正常细菌分裂增殖过程中,肽聚糖水解酶作用于细胞壁从而实现母代细胞与子代细胞分离.而当正常的调控失衡时,肽聚糖水解酶则会导致细胞的自溶[4].
保加利亚乳杆菌LJJ是本实验室从传统乳制品中分离得到的一株乳酸菌,该菌种在乳制品发酵过程中表现出较强的自溶特性,而在乳制品的发酵过程中,发酵剂的自溶并不总是有利的[5].因此,为了有效地调控乳酸菌的自溶性,拟对其自溶的发生机理进行研究.已有的研究结果表明肽聚糖水解酶在乳酸菌自溶过程中起着重要作用[6-7].
本研究选择iCODEHOP[8]在线软件设计简并引物用于β-N-乙酰氨基己糖苷酶基因片段的克隆.鉴于CODEHOP的诸多优点[8-10],迄今为止,利用此方法已成功扩增出了鹅[11]、多种细菌[12-13]、人[14]、植物[15]、动物[16]等生物体中的多种未知基因序列片段.选择iCODEHOP在线软件设计简并引物扩增克隆保加利亚乳杆菌LJJ的β-N-乙酰氨基己糖苷酶,可有效地保证目的基因片段的获取,为后续荧光定量RTPCR研究其在乳酸菌自溶过程中的作用奠定基础.
1 材料与方法
1.1 材料
1.1.1 主要数据库和生物软件
NCBI、BlockMaker(http:∥ blocks.fhcrc.org/blocks/makeblocks.html或 http:∥blocks.fhcrc.org/blockmkr/makeblocks.html)、CodeHop(http:∥blocks.fhcrc.org/blocks/codehop.html)、clustalW2(http:∥www.ebi.ac.uk/Tools/clustalw2/index.html)、iCodeHop(http:∥dbmi-icode-01.dbmi.pitt.edu/i-codehop-context/Welcome).
1.1.2 菌株与质粒
德式乳杆菌保加利亚亚种(Lactobacillus delbrueckiisubsp.bulgaricus)LJJ,本实验室-20℃冰箱保存;大肠杆菌 DH5α,北京天根生化公司;质粒pGET-T,美国Promega公司.
1.1.3 试剂
细菌基因组提取试剂盒、2×Bench TopTMTaq Master Mix、琼脂糖凝胶回收试剂盒,美国Biomiga公司;克隆试剂盒,美国Promega公司;引物合成,北京华大基因公司.
1.2 仪器设备
TP600型梯度PCR仪,日本Takara株式会社;FluorChem FC2型凝胶成像系统,美国Alpha Innotech公司;DYY-6C型电泳仪,北京六一仪器公司;DL-CJ-IN型高性能无菌试验台,哈尔滨市东联公司;DHP-9082型电热恒温培养箱,上海一恒科学仪器有限公司;LDZX-50KB型立式压力蒸汽灭菌锅,上海申安医疗器械厂.
1.3 方法
1.3.1 简并引物的设计及筛选
从NCBI蛋白质数据库中,查找已知相应菌种N-乙酰氨基己糖苷酶的氨基酸序列.通过16S rDNA查找比对选取亲缘关系较近、同源性较高菌种的N-乙酰氨基己糖苷酶的氨基酸序列,分别为:Lactobacillusdelbrueckiisubsp.lactisDSM 20072 GB_EGD27790.1,Lactobacillus amylovorusGRL 1112,YP_004031587.1,Lactobacillus suebicusKCTC 3549,ZP_09451258.1,Lactobacillus salivariusUCC118,YP_536455.1,LactobacilluskefiranofaciensZW3,YP_004563601.1,选上述氨基酸序列以FASTA格式保存.
利用iCODEHOP程序设计简并引物的步骤如下:1)进入在线主页选择开始一个non-named或者named session;2)选择引物设计(Design Primers)选项,选择上传提交(SUBMIT)本地以FASTA保存的上述序列;3)选择“进行分析”(Proceed with analysis)选项.程序显示通过Clustal W2对这些氨基酸进行多序列比对,选择SUBMIT生成比对结果.并可以进化树的形式表示这些氨基酸的关系远近,命名比对结果后,选择 Proceed,获得保守区(block),本实验共得到6个保守区.4)简并引物的设计.设置引物主要参数为:遗传密码(Genetic code)standard,其余参数均采用默认设置;简并度(Degeneracy)128(默认);退火温度 (Temperature)为60.0℃(默认);精密度(strictness)为0.0(默认),选择“找寻引物”(Look for primers).5)简并引物的筛选.页面显示所有符合条件的引物以箭头表示,并按照保守区的顺序以字母表顺序排列.点击相应箭头即显示其对应氨基酸序列,依据得到的引物得分(Clamp score)及简并度(Core degeneracy)情况选择引物 .引物得分应越高越好,简并度应越小越好.
1.3.2 简并引物扩增N-乙酰氨基己糖苷酶基因
依照Biomiga细菌组DNA提取试剂盒中操作提取细菌总DNA,用设计筛选的简并引物进行扩增.
PCR反应体系为 20 μL:2×Bench TopTMTaq Master Mix,10 μL,上游引物为 20 μmol,1 μL,下游引物为 20 μmol,1 μL,DNA 模板,5 ng,加 ddH2O 补至20 μL.PCR 反应条件:94℃预变性,5 min,然后94℃变性30 s,60℃至50℃间退火,72℃延伸1 min,共进行30个循环;最后72℃延伸10 min.
1.3.3 PCR产物的检测、鉴定、回收、克隆及测序
采用1.5%的琼脂糖凝胶电泳检测PCR扩增产物.采用北京天根生化公司的凝胶回收试剂盒回收目的片段.克隆步骤参见Promega公司的pGEM-T试剂盒说明书所述方法.胶回收DNA片段与pGEM-T载体在4℃连接过夜,转化大肠杆菌DH5α,37℃培养14~16 h,挑选6个白色菌落溶于10 μL ddH2O中,以此为 DNA模板,以RV-M 和M13-47通用引物进行菌落PCR鉴定,将阳性克隆送至华大基因进行测序.
2 结果与分析
2.1 筛选得到的简并引物
综合考虑不同引物对的引物简并度,引物得分及引物退火温度情况,选取引物对Hex1-f,Hex1-r和Hex2-f,Hex2-r作为扩增β-Hecase基因的简并引物.引物具体序列如表1.预计两引物对所扩增产物大小约为620 bp.

表1 N-乙酰氨基己糖苷酶的简并引物Tab.1 Degenerate primer sequences of β-Hexcase
2.2 简并引物PCR扩增结果
分别以 Hex1-f,Hex1-r和 Hex2-f,Hex2-r作为上下游引物,以LJJ基因组DNA为模板,经梯度PCR扩增.由图1可知,引物对Hex1-f,Hex1-r在约600 bp处获得清晰明亮的产物条带,与目的产物片段大小一致.此时在大于1 500 bp处亦存在较模糊的条带,而随着退火温度提高到60℃,条带开始变得模糊,这主要是由于简并引物具有一定的简并性,存在扩增出其他非特异性片段的可能性.退火温度较低时,由于引物特异性有所降低,这一现象表现得较为严重,该非特异性产物经目的条带的凝胶回收分离后将不会影响后续的实验结果[17-19].
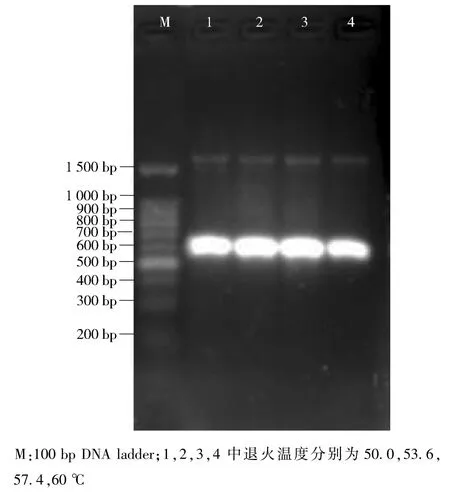
图1 简并引物梯度PCR扩增产物结果Fig.1 Result of CODEHOP by gradient PCR
2.3 简并引物PCR产物的克隆与鉴定
菌液PCR扩增结果如图2.以所挑选的6个白色菌落菌液为模板,经载体通用引物扩增6个阳性克隆的菌液PCR均在略大于600 bp处得到清晰明亮的特异性产物条带,产物大小与插入目的基因片段大小一致.

图2 TA克隆阳性克隆的菌液PCR琼脂糖凝胶电泳图Fig.2 Agarose gel electrophoresis of PCR amplification of positive cloning
2.4 PCR产物的测序及序列比对
选取两个阳性克隆进行测序,测序结果显示,简并引物PCR产物长度为614 bp,详细序列如下:AACTGGCCAGCCTTTTCCTAATCGACAGCACTGGCGA TTTTACTGAAAACCTCCGACTTTTGGAGCAGTACCAG CCAGCCGGCCTCCTACTCTTTGCCAAAGACCTGGCGG GCCTAAGCGAAAAGGCTGTTAAAGAGCGGCTGGCGA TTTACCGGCAGGCCCGGCCAGGTTTGCTGCTCGCCAT TGACCAGGAAGGCGGCTTAGTCAGCCGCTTGTCCACC CTTTACCCAAACCGGTCTTACCCCAGCCAAGCTGAAC TTTTGAAAAAGGGGGCTGACTTTTTCTTAGCAGAAAA CCAAAAGACGGCCCTGAAATTAAAAGATTTAGGCAT CAACTTGAACTTTGCTCCGGTGGCTGATCTTGCGCTT GATCCGGCCAGTTTCATCTACAGCCGGACCCTGCAAG CAGGAGCTGAGGAAACCGGGCCGGCAATCGCGGCCT TTATCAAGCTCTACCGGCAGCTGGGAGTCGCCTCCTG CGCCAAGCATTTTCCTGGCTACGGGGATGCCGGCGAC ACCCACCAGGCTCCGGCTAAAGACCTTAGAAGCCTTA AAGAAGCCCAGCTGGATCTTTTACCTTTTAAAGCTGC GATCGCGGCGAGCGTGCCGACTATC
简并引物预测的产物长度约为620 bp,而实际测序得到的产物长度为614 bp.可能是在去除载体序列时多去掉了几个碱基造成的,但测序结果与预测产物长度基本一致.如表2所示,扩增物序列的blastx比对结果显示,LJJ与L.delbrueckiisubsp.lactisDSM 20072中β-N-乙酰氨基己糖苷酶的一致性最高,而与Lactobacillus suebicusKCTC 3549中β-N-乙酰氨基己糖苷酶的一致性最低,其一致性仍达到40%.因此,可以证明该扩增片段确为LJJ中β-N-Acetylhexosaminidase的部分基因片段.

表2 简并引物Hex1-f,Hex1-r扩增序列的blastx比对结果Tab.2 Blastx results of amplified sequence with degenerate primer Hex1-f and Hex1-r
3 结 论
利用iCODEHOP在线简并引物设计软件,设计得到的简并引物对Hex1-f和Hex1-r,可以从保加利亚乳杆菌LJJ中扩增得到大小为614 bp的扩增产物,经TA克隆测序,blastx比对验证其为β-N-乙酰氨基己糖苷酶部分基因片段.β-N-乙酰氨基己糖苷酶基因的获得为下一步采用荧光定量RT-PCR分析β-N-乙酰氨基己糖苷酶基因在LJJ自溶的变化调控创造了条件.
LJJ β-N-乙酰氨基己糖苷酶保守基因片段的成功克隆证明由已知亲缘关系较近的同源物种的已知基因序列,利用iCODEHOP简并引物设计软件设计的简并引物可有效地扩增克隆得到目的物种的基因序列片段,从而为未知基因片段的获取提供高效便捷的途径.
[1] Slámová K, Bojarová P, Gerstorferová D, et al.Sequencing, cloning and high-yield expression of a fungal β-N-acetylhexosaminidase inPichia pastoris[J].Protein Expression and Purification,2012,82:212217.
[2] Slámová K, Bojarová P, Petrásková L, et al. β-NAcetylhexosaminidase:What's in a name...?[J].Biotechnology Advances, 2010, 28:682693.
[3] Masahiro Ogawa, Mai Kitagawa, Hideharu Tanaka, et al.A β-N-acetylhexosaminidase from Symbiobacterium thermophilum gene cloning, overexpression, purification and characterization[J].Enzyme and Microbial Technology,2006,38:457464.
[4] Uehara T, Parzych K R, Dinh T, et al.Daughter cell separation is controlled by cytokinetic ring-activated cell wall hydrolysis[J].EMBO Journal, 2010, 29:1412-1422.
[5] Chopin A,Bolotin A,Sorokin A,et al.Analysis of six prophages inLactococcus lactisIL1403 different genetic structure of temperate and virulent phage populations[J].Nucleic Acids Research,2001, 29:644 -651.
[6] Bernard E, Rolain T, Courtin P, et al.Characterization of O-Acetylation of N-Acetylglucosamine a novel structural bariation of bacterial peptidoglycan[J].Journal of Biological Chemistry, 2011, 286:23950 -23958.
[7] Atsushi Furukawa, Kumiko Nakadi-Tsukui, Tomoyoshi Nozaki.Novel transmembrane receptor involved in phagosome transport of lysozymes and b-hexosaminidase in the enteric protozoanEntamoeba histolytica[J].PLoS Pathogens, 2012, 8:115.
[8] Boyce1 R,Chilana P,Rose T M.iCODEHOP a new interactive program for designing consensus-degenerate hybrid oligonucleotide primers from multiply aligned protein sequences[J].Nucleic Acids Research,2009,37:222 -228.
[9] Rose T M,Schultz E R,Henikoff J G,et al.Consensusdegenerate hybrid oligonucleotide primers for amplification of distantly related sequences[J].Nucleic Acids Research,1998,26:1628 -1635.
[10] 高安崇,史旭升,孙永峰,等.鹅CAT4基因的简并引物设计及克隆[J].中国家禽,2010,32(12):15-19.
[11] 逄晓阳,刑鑫,刘国文,等.用CODEHOP设计简并引物克隆反刍月形单胞菌K6乙酸激酶基因片段[J].中国兽医学报,2010,30(1):102106.
[12] 任南琪,林海龙,李建政,等.用CODEHOP设计简并引物克隆B49乙酸激酶基因片段[J].哈尔滨工业大学学报,2007,39(8):1226 -1230.
[13] 李夏,袁建军,卢英华,等.简并PCR法克隆L.starkeyi苹果酸酶基因[J].泉州师范学院学报:自然科学版,2010,28(2):59-62.
[14] 姜晶,孙颖,刘红艳,等.人免疫球蛋白重链可变区基因引物设计方法的改良[J].微生物学杂志,2012,32(2):6063.
[15] 智强,李淑慧,高利宏,等.利用简并PCR克隆烟草节杆菌02181肌酸酶基因[J].第三军医大学学报,2007(12):6568.
[16] 侯林,李楠,伊男,等.中国卤虫早期胚胎发育相关基因的研究—Orthodenticle基因克隆中简并引物的设计和优化[J].辽宁师范大学学报:自然科学版,2009,32(1):99101.
[17] Chen M,Liu H L,Bai Y F,et al.Homologous-restraint polymerase Chain reaction:an efficient and rapid protocol to clone multiple homologous genes[J].Current Microbiology, 2008, 57:51-54.
[18] Zlateva K T,Crusio K M,Leontobich A M,et al.Design and validation of consensus-degenerate hybrid oligonucleotide primers for broad and sensitive detection of corona-and toroviruses[J].Journal of Virological Methods, 2011, 177:174183.
[19] Acevedo J P, Reyes F, Parra L P, et al.Cloning of complete genes for novel hydrolytic enzymes from Antarctic sea water bacteria by use of an improved genome walking technique[J].Journal of Biotechnology, 2008,13:277286.
