鸡Lpin1基因5′侧翼区微卫星变异的鉴定
2013-07-04张建宏张淑平康相涛黄艳群
张建宏,张淑平,陈 文,康相涛,黄艳群
(河南农业大学牧医工程学院,河南 郑州 450002)
微卫星标记由于为多等位基因标记,在基因定位、遗传多样性研究上具有重要的应用价值,开发新的鸡微卫星标记可进一步丰富鸡微卫星数据库.位于启动子区域的微卫星具有潜在重要的功能[1,2].鸡Lpin1基因是鸡重要的脂肪沉积相关的候选基因,鸡Lpin1基因微卫星的分布特性及其5′侧翼区微卫星变异的遗传变异研究,可为进一步开展鸡Lpin1基因的表达调控研究打下基础.微卫星DNA 又称短串联重复序列(Short tandem repeat,STR),是基因组中广泛存在的一种以1~6个核苷酸为重复单位串联组成的核苷酸序列[3].STR 标记能揭示比RFLP 高得多的多态性,曾被作为第2代分子标记广泛应用于遗传变异分析、物种起源进化、基因定位、指纹鉴定、遗传图谱构建、法医科学和动植物遗传育种等领域.因为STR 标记必须依赖每类微卫星两端序列设计引物,这造成微卫星标记的应用与开发的数量受到了一定的限制[4].微卫星的功能目前尚不甚明了,曾一度被认为呈选择中性、缺乏功能性,但最近的报道显示,微卫星重复序列长度的变异能诱使启动子构象的变异,影响其结合转录调控子的能力[2].对基因非翻译区的微卫星变异研究显示其参与基因的表达与调控[5];重复序列的动态突变可引起mRNA和蛋白质水平的变化[6];还有研究显示,微卫星参与细胞分化过程,有自身特异性结合蛋白,还能直接编码蛋白质,是一种非常活跃的碱基序列[5].Lpin1基因是2001年发现的与机体脂肪沉积有密切相关性的基因,为Lpin 蛋白家族成员[7],是一种Mg2+依赖性磷脂酸磷酸化酶基因,具有磷脂酸磷酸化酶的活性[8,9],与动物脂肪形成密切相关[10].在小鼠、人类Lpin1基因上的研究表明,该基因存在多种可变剪接产物,Lpin1-α 与 Lpin1-β 剪 接 体 在 人(NM _145693)[11]和鼠(NM_172950)[12]具有不同但互补的功能[13].Lpin1的表达调控受多种因素的影响,磷酸化可改变基因的亚细胞定位,进而影响Lpin1的活性并最终导致其表达变化[14].鸡Lpin1基因的编码序列(HM473175)包含19个外显子,王伯君等[15]发现鸡Lpin1基因编码区的变异与皮下脂肪厚、腹脂重呈现显著的关联.李素雅等[16]进行了鸡Lpin1基因3′ UTR的变异研究.目前尚未见对鸡Lpin1基因微卫星序列分布及变异的报道.本研究进行了鸡Lpin1基因组内微卫星分布特性、5′侧翼区的微卫星遗传变异鉴定及其潜在效应研究,该研究可丰富鸡的微卫星数据库,为进一步进行该微卫星变异的功能鉴定奠定基础,同时也为Lpin1基因的表达调控机制及功能研究提供一定的依据.
1 材料与方法
1.1 血样及其DNA 提取
河南家禽种质资源创新工程中心的固始鸡(GS)、丝羽乌骨鸡(SK)、河南斗鸡(HG)、卢氏绿壳蛋鸡(LS)、白来航(WL)、白洛克(WP),每品种10 只,共计60份具有特色鸡种的血样.取80μL全血于1.5 mL Eppendorf 管中,加入500μL TE 缓冲液,8μL 25% SDS和9μL 蛋白酶K (10 g·L-1),混匀置55℃水浴过夜.采用常规的酚氯仿抽提方法提取基因组DNA.抽提产物经1%琼脂糖凝胶电泳检测后-20℃保存备用.
1.2 微卫星搜索和PCR 引物设计
采用SSR hunter 在鸡Lpin1基因组(NC_006090)序列搜索潜在的微卫星.根据鸡Lpin1基因组(NC_006090)序列,使用Oligo 软件设计1 对引物(MS)扩增位于鸡 Lpin1 编码序列(HM473175)的5′ 侧翼区(Flanking region,FLR)预测的启动子区域微卫星序列,引物序列为:F:5′-AGCGGTCTCTCTTTGGTGAT-3′,R:5′-AGTAGTTGCTGCCTGAGACA-3′.预期PCR 扩增产物长度为180 bp 左右.引物由上海生工生物工程技术服务有限公司合成.
1.3 PCR 扩增及基因分型
PCR 反应体系:DNA 模板0.5μL,上游引物0.25μL,下游引物0.25μL,Taq 酶0.1μL,2×Reaction Mix 6.25μL,双蒸水5.15μL,总反应体积12.5μL.PCR 反应程序:94℃预变性5 min,94℃变性30 s,56℃退火35 s ,72℃延伸35 s,72℃延伸10 min,30个循环,4℃终止反应.
PCR 扩增产物首先经2.5%琼脂糖凝胶电泳进行初步检测.然后取2μL 非变性PCR 产物在12%非变性聚丙烯酰胺凝胶上电泳2 h(130 V 电压),采用改良的银染技术显色、分型,并用UVP 自动凝胶成像系统扫描保存图像.
1.4 克隆测序
分别选取包含不同等位基因型的个体进行50μL 体系PCR 扩增,采用PCR 回收试剂盒进行产物纯化(Tiangen).PCR 产物连接到pGM-T 载体(Promega)并转化大肠杆菌,转化产物涂布于含有氨苄和X-gal的LB 固体培养基表面,挑取白斑进行摇菌,12~16 h后进行菌液PCR 扩增做进一步鉴定.经PCR 鉴定为阳性克隆的菌液送上海生物工程有限公司测序.对于包含不同等位基因的杂合型个体通过从同一个体选择多个克隆进行测序,以保证获得2 条链的序列信息.
1.5 基因分析预测
采用FirstEF 软件进行Lpin1基因启动子和第1 外显子预测,采用SSR hunter 软件进行微卫星搜索,采用TFSEARCH 软件进行转录因子结合位点预测.
1.6 统计分析
采用BandScan 软件进行非变性聚丙烯酰胺凝胶的基因型判别,采用Cervus2.0 进行数据统计分析,具体指标包括等位基因数、等位基因频率、杂合度及多态信息含量(PIC)等参数.
2 结果与分析
2.1 鸡Lpin1基因的启动子预测
将鸡Lpin1基因的基因组序列(NC_006090)与其编码序列(HM473175)进行比对分析表明,鸡Lpin1基因的第1 编码外显子位于基因组序列(NC_006090)的20629~20822 区域.采用FirstEF 软件(http://rulai.cshl.org/cgi-bin/tools/FirstEF/)[17]从鸡基因组序列(NC_006090)的正义链预测到鸡Lpin1基因的启动子区域(12875~13444),1个CpG window 位于13113~13314.CpGPlot 程序(http://www.ebi.ac.uk/Tools/emboss/cpgplot/index.html)也预测到1个723 bp的CpG 岛(12631~13353).
2.2 鸡Lpin1基因微卫星位点的搜索与分析
鸡Lpin1基因组(NC_006090)序列约为50 kb,SSR hunter 软件从中搜索到9个STR(表1),其频率为每5.5kb 有1个STR.这些序列多为二核苷酸型串联重复序列,仅包含1个三核苷酸型和1个复合型(四核苷酸与二核苷酸复合型)STR,其重复次数在5~18 间.其中3个STR 位于鸡Lpin1基因(HM473175)的5′ FLR,6个位于内含子区域.复合微卫星(命名为CLP532)的核心序列正好位于预测的启动子区域附近,CLP532 微卫星的核心序列在鸡基因组(NC_006090)参考序列为(TATT)6/(TG)16(图1).

图1 位于预测的鸡Lpin1基因启动子区域附近的复合微卫星序列Fig.1 The composite microsatellite located at the predicted promoter region of chicken Lpin1 genomic sequence

表1 从鸡Lpin1基因组序列(NC_006090)搜索到的STRsTable 1 STRs searched from the chicken Lpin1 genome sequence (NC_006090)
2.3 CLP532 复合微卫星变异的鉴定
采用MS 引物从6个不同品种鸡的基因组中扩增CLP532 复合微卫星核心序列,其扩增产物经2.5%琼脂糖凝胶电泳检测(图2),表明MS 引物对该微卫星的PCR 扩增特异性好、扩增效率高,获得了预期的180 bp 左右的条带.从琼脂糖凝胶电泳图上可以清楚地看出,PCR 扩增产物在所检测个体间存在多种长度差异,显示该微卫星在群体中存在丰富的变异.
由于受检测灵敏度限制,对于一些长度差异在20个碱基内的等位基因,采用琼脂糖凝胶电泳常常无法有效分离,从而影响基因型判别的准确性.因此本试验进一步采用非变性聚丙烯酰胺凝胶电泳检测CLP532 微卫星的变异.采用12%非变性聚丙烯酰胺凝胶电泳,PCR 扩增产物均得到了有效分离,在固始鸡(GS)、丝羽乌骨鸡(SK)、河南斗鸡(HG)、卢氏绿壳蛋鸡(LS)、白来航(WL)、白洛克(WP)6个品种中检测共得到A,B,C,D,E,F 6种等位基因型(图3).式为:(TATT )5/(TG )7,(TATT )5/(TG )8,(TATT)4/(TG)13,(TATT)4/(TG)15,(TATT)5/(TG)18和 (TATT)5/(TG)19.MS 引 物 扩 增的CLP532 微卫星序列长度在158~182 bp 间,其等位基因的序列长度变异在25 bp 左右.该复合微卫星的核心序列TTAT和TG的重复数同时都发生了变异,且发现TG 核心序列的变异类型相对比TATT 多.
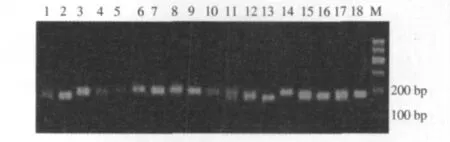
图2 MS 引物部分扩增产物琼脂糖凝胶电泳检测Fig.2 The agarose gel electrophoresis photo of the part of PCR amplification products by MS primer set
选择包含不同等位基因的代表性个体进行克隆测序,成功获得了A,B,C,D,E,F 6种等位基因的序列信息(图4,表2).其核心序列的具体变异形

图3 非变性聚丙烯酰胺凝胶电泳检测Fig.3 The map of MS primer amplification products by non-denaturing polyacrylamide gel electrophoresis

图4 CLP532 微卫星不同等位基因的克隆测序峰Fig.4 The clone sequencing peak map of different alleles of CLP532 microsatellite

表2 CLP532 微卫星等位基因核心序列及其PCR 扩增长度Table 2 The core sequence of different alleles for CLP532 microsatellite and the PCR amplification length
2.4 CLP532 微卫星在品种间的遗传多态性
CLP532 微卫星在品种间的变异统计结果表明(表3),从6个鸡品种中共检测到6种等位基因.B,C,D 等位基因广泛存在于所检测的6个品种中,A 等位基因存在于4个品种中,E 等位基因存在于3个品种中,而F 等位基因仅存在于固始鸡和卢氏绿壳蛋鸡中,未检测到品种特异性的等位基因.从固始鸡、河南斗鸡和卢氏绿壳蛋鸡检测到了5种等位基因,从白来航、白洛克和丝羽乌骨鸡检测到4种等位基因.6个品种鸡的平均等位基因数为4.5,平均多态信息含量为0.665,平均杂合度为0.763,其中丝羽乌骨鸡平均杂合度最高,而白洛克表现了最低的平均杂合度.
2.5 CLP532 微卫星变异对转录因子结合位点的影响预测
采用在线分析软件TFSEARCH 进行CLP532微卫星G 等位基因核心序列(来自于基因组序列NC_006090)的转录因子结合位点预测(图5),在CLP532 微卫星核心序列区域共预测到23个转录因子结合位点,包括13个CdxA,3个HNF-3b,3个HNF-2,3个Pbx-1和1个XFD-1 转录因子结合位点.分别进行CLP532 微卫星不同等位基因核心序列的转录因子预测表明,该微卫星核心序列长度的变异引起了转录因子结合位点的改变.E,F 等位基因与G 等位基因相比少了6个转录因子结合位点,分别为3个CdxA,1个HNF-3b,1个HNF-2和1个Pbx-1.C,D 等位基因与G 等位基因相比则少了12个转录因子结合位点,分别为6个CdxA,2个HNF-3b,2个HNF-2和2个Pbx-1.

表3 CLP532 微卫星在品种间的多态性Table 3 CLP532 microsatellite Polymorphisms among breeds

图5 CLP532 微卫星G 等位基因核心序列的转录因子结合位点预测Fig.5 Transcription factor binding site prediction of CLP532 microsatellite core sequence for G allele
3 小结与讨论
3.1 新的微卫星序列的开发
微卫星广泛分布于真核生物的基因组中,有报道显示,除着丝粒及端粒外染色体的其它区域广泛分布有微卫星位点[18].在哺乳动物中似乎每个基因都至少存在1个微卫星标记[19],PRIMMER等[20]报道在禽类基因组中微卫星分布的密度相对较低,大约为20~39 kb 1个STR,并认为这可能是家禽基因组中所含的非编码DNA相对少于哺乳动物所致.本研究显示,鸡Lpin1基因组上存在高频率的微卫星序列(平均每5.5 kb 则分布1个STR).目前主要应用的6种微卫星开发技术包括直接文库筛选法、基于锚定PCR 技术法、单引物延伸富集法、选择杂交富集法、生物信息学法、转移扩增法.但是利用生物信息学方法开发微卫星只能应用于序列信息已知的物种[21].然而,随着基因组序列、mRNA 序列等核酸序列的不断增加,生物信息学法已会发展成为一种简便、快速、实用的微卫星开发技术.本研究采用生物信息学方法从鸡Lpin1基因组(NC_006090)序列发现了丰富的串联重复序列,采用琼脂糖电泳、聚丙烯酰胺凝胶电泳和克隆测序的方法从6个鸡品种中鉴定了1个位于鸡Lpin1基因5′侧翼区(预测启动子区域)的微卫星序列多态性,丰富了鸡微卫星标记数据库.
3.2 CLP532 微卫星在鸡品种间的遗传多样性
本研究采用新鉴定的CLP532 微卫星标记初步分析了该微卫星在6个鸡种的遗传多样性.从有限样本数的6个鸡品种中鉴定了6种等位基因,从固始鸡、河南斗鸡和卢氏绿壳蛋鸡检测到了5种等位基因,白来航、白洛克和丝羽乌骨鸡检测到4种等位基因.李素雅等[16]采用直接PCR 测序的方法从470 bp的3′-UTR 检测到8个变异,采用与本试验相同的样本,从固始鸡和卢氏绿壳蛋鸡检测到7个变异位点,而从10 只河南斗鸡中未检测到任何变异,显示了河南斗鸡在该区域的高度保守性.河南斗 鸡 在 鸡Lpin1基 因3′-UTR和5′ 侧 翼 区CLP532 微卫星标记变异的高度差异,显示了河南斗鸡在鸡Lpin1基因不同区域选择强度上的差异,这些差异是否与基因的表达调控乃至河南斗鸡表现型有关联尚需要进一步研究.
3.3 Lpin1基因5′ 侧翼区微卫星潜在功能分析
微卫星快速进化而且存在于真核生物基因组的各个地方,但优势存在于非编码DNA 区域[22].本研究从鸡Lpin1基因的基因组中搜索到9个微卫星序列,3个位于基因的5′ 侧翼区,6个位于内含子区域,微卫星在真核生物的丰度和位置及其高的突变率提示其在基因调控上的潜在和广泛的作用.启动子存在于结构基因上游,是与基因转录启动有关的一段特殊DNA 序列,控制基因表达(转录)的起始时间和表达的程度.启动子区域的变异常会影响转录因子的结合从而影响基因的表达调控.很多的报道证实了位于启动子区域的长度变异的STR 被克隆入载体能表现不同的表达能力[1,2,23],SHIMAJIRI 等[1]研究表明matrix metalloproteinase 9基因的表达被短的STR 序列下调,TAE等[23]报道1个双核苷酸重复序列在acetyl-CoA carboxylase基因的负调控效应是CAAT box 依赖性的,可被CAAT 增强子结合蛋白而逆转.非编码区的微卫星序列对于基因表达调控具有一定的作用,主要是由于形成了特异DNA,如Z-DNA[24],HDNA[25].Z-DNA 在特定的染色体区域上执行着重要的功能,如结构基因的启动子和重组热点均富含Z-DNA 结构[26].本研究预测鸡Lpin1基因5′侧翼区的STR 不同等位基因型可引起转录因子结合位点的变化,显示该基因可能会通过该微卫星序列长度变异程度进行基因的表达调控.
鸡Lpin1基因组内存在丰富的串联重复序列,从位于鸡Lpin1基因5′侧翼区(预测的启动子区域)的复合微卫星CLP532中鉴定了7个等位基因,其长度差异在25 bp,显示该微卫星可作为新的微卫星标记进行遗传多样性等研究.软件预测该微卫星长度的变异可引起CdxA,HNF-3b,HNF-2和Pbx-1 转录因子结合位点及其结合位点数目的改变,显示了该微卫星长度的变异对Lpin1基因表达调控的潜在效应.
[1]SHIMAJIRI S,ARIMA N,TANIMOTO A,et al.Shortened microsatellite d (CA)21 sequence down-regulates promoter activity of matrix metalloproteinase 9 gene[J].FEBS Letters,1999,455(1):70-74.
[2]STREELMAN J T,KOCHER T D.Microsatellite variation associated with prolactin expression and growth of salt-challenged tilapia[J].Physiological Genomics,2002,9(1):1-4.
[3]TAUTZ D.Hypervariability of simple sequences as a general source for polymorphic DNA markers[J].Nucleic Acids Research,1989,17(16):6463-6471.
[4]刘学军,童继平,李素敏,等.DNA 标记的种类、特点及其研究进展[J].生物技术通报,2010 (7):35-40.
[5]SAVELIEV A,EVERETT C,SHARPE T,et al.DNA triplet repeats mediate heterochromatin-protein-1-sensitive variegated gene silencing[J].Nature,2003,422:909-913.
[6]黄建军.非编码区三核苷酸重复序列动态突变及相关疾病机制的研究进展[J].国外医学生理:病理科学与临床分册,2003,23(2):119-122.
[7]DONKOR J,SARIAHMETOGLU M,DEWALD J,et al.Three mammalian lipins act as phosphatidate phosphatases with distinct tissue expression patterns[J].Journal of Biological Chemistry,2007,282(6):3450-3457.
[8]SANTOS-ROSA H,LEUNG J,GRIMSEY N,et al.The yeast lipin Smp2 couples phospholipid biosynthesis to nuclear membrane growth[J].EMBO Journal,2005,24(11):1931-1941.
[9]HAN G S,WU W I,CARMAN G M.The Saccharomyces cerevisiae Lipin homolog is a Mg2+-dependent phosphatidate phosphatase enzyme[J].Journal of Biological Chemistry,2006,281(14):9210-9218.
[10]GALE S E,FROLOV A,HAN X,et al.A regulatory role for 1-acylglycerol-3-phosphate-O-acyltransferase 2 in adipocyte differentiation[J].Journal of Biological Chemistry,2006,281(16):11082-11089.
[11]SUVIOLAHTI E,REUE K,CANTOR R M,et al.Cross-species analyses implicate Lipin1 involvement in human glucose metabolism[J].Human Molecular Genetics,2006,15(3):377-386.
[12]PETERFY M,PHAN J,REUE K.Alternatively spliced lipin isoforms exhibit distinct expression pattern,subcellular localization,and role in adipogenesis[J].Journal of Biological Chemistry,2005,280(38):32883-32889.
[13]CHEN Z,GROPLER M C,NORRIS J,et al.Alterations in hepatic metabolism in fld mice reveal a role for Lipin1 in regulating VLDL-triacylglyceride secretion[J].Arteriosclerosis,Thrombosis,and Vascular Biology,2008,28(10):1738-1744.
[14]HARRIS T E,HUFFMAN T A,CHI A,et al.Insulin controls subcellular localization and multisite phosphorylation of the phosphatidic acid phosphatase,Lipin1[J].Journal of Biological Chemistry ,2007,282(1):277-286.
[15]王伯君,孙 玲,黎绍波,等.鸡Lpin1基因c.1727C_T 位点遗传变异及其与脂肪沉积性状的关联分析[J].畜牧兽医学报,2010,41(9):1076-1081.
[16]李素雅,陈 文,黄艳群,等.鸡Lpin1基因3′-UTR遗传变异及其对miRNA 结合位点的潜在效应[J].中国农业科学,2012,45(8):1613-1620.
[17]DAVULURI R V,GROSSE I,ZHANG M Q.Computational identification of promoters and first exons in the human genome[J].Nature Genetics ,2001,29(4):412-417.
[18]WINTERO A K,FREDHOLM M,THOMSEN P D.Variable (dG-dT)n.(dC-dA)n sequences in the porcine genome[J].Genomics,1992,12(2):281-288.
[19]NICOLAS A L,MUNZ P L,YOUNG C S.A modified single-strand annealing model best explains the joining of DNA double-strand breaks mammalian cells and cell extracts[J].Nucleic Acids Research,1995,23(6):1036-1043.
[20]PRIMMER C R,RAUDSEPP T,CHOWDHARY B P,et al.Low frequency of microsatellites in the avian genome[J].Genome Research,1997,7(5):471-482.
[21]HAN Z G,GUO W Z,SONG X L,et al.Genetic mapping of EST-derived microsatellites from the diploid Gossypium arboreum in allotetraploid cotton[J].Molecular Genetics and Genomics,2004,272(3):308-327.
[22]TÓTH G,GÁSPÁRI Z,JURKA J.Microsatellites in different eukaryotic genomes:survey and analysis[J].Genome Research ,2000,10(7):967-972.
[23]TAE H J,LUO X,KIM K H.Roles of CCAAT/enhancer-binding protein and its binding site on repression and derepression of acetyl-coA carboxylase gene[J].Journal of Biological Chemistry,1994,269 (14):10475-10479.
[24]ROTHENBURG S,KOCH-NOLTE F,RICH A,et al.A polymorphic dinucleotide repeat in the rat nucleolin gene forms Z-DNA and inhibits promoter activity[J].Proceedings of the National Academy of Sciences,USA,2001,98(16):8985-8990.
[25]FABREGAT I,KOCH K S,AOKI T,et al.Functional pleiotropy of an intramolecular triplex-forming fragment from the 3′-UTR of the rat pigr gene[J].Physiol Genomics,2001,5(2):53-65.
[26]WAHLS W P.Meiotic recombination hotspots:shaping the genome and insights into hypervariable minisatellite DNA change[J].Current Topics in Developmental Biology,1998,37:37-75.
