一种新的DyP-type过氧化物酶在大肠杆菌中的重组表达、纯化及鉴定
2013-06-30汪立群AlanChang袁文杰白凤武
汪立群,Alan K.Chang,袁文杰,白凤武
大连理工大学生命科学与技术学院,辽宁大连 116024
过氧化物酶是氧化还原酶的一种,它的特点是可以利用过氧化氢氧化各种有机和无机化合物,在生物合成、降解和防御功能[1-4]等生命过程中发挥重要的作用。大多数过氧化物酶都是含有亚铁血红素的蛋白,在活性位点有一个铁卟啉Ⅸ。最近,亚铁血红素蛋白中的DyP-type 过氧化物酶类在疾病诊断和木质素等顽固废弃物的降解方面都表现出很大的应用潜能[5-9],越来越受研究者所关注。
在1995年发现了一株具有染料脱色功能的担子菌纲真菌水稻纹枯病菌 Thanatephorus cucmeris Dec 1,它能使18种不同类型的外源染料脱色[10],随后对该菌株中有染料脱色能力的酶进行了纯化,发现这种酶的底物特异性和传统过氧化物酶不同,鉴定为含亚铁血红素的新过氧化物酶,称为DyP[11]。研究者已从不同的真菌中纯化出DyP-type 过氧化物酶[12-15],晶体结构研究表明其与以前描述的具有代表性的过氧化物酶是不同的。例如,传统过氧化物酶主要是由α螺旋蛋白构成,而缺乏β折叠,但DyP-type 过氧化物酶中β折叠如三明治一样的结构夹在α螺旋之间;另外,在亚铁血红素依赖的植物类型过氧化物酶中,为了亚铁血红素的定位,会有Fe-His-His 三个联体保守氨基酸,而DyP-type 中是Fe-His-Asp 这样的特定结构[16-18]。在细菌中也发现了DyP-type 过氧化物酶,YcdB 是从E.coli中分离出来,研究最早也是最深入的DyP-type过氧化物酶之一[19]。目前,DyP-type 过氧化物酶基因已被克隆表达,并进行了酶的分离纯化及性质研究[20-24],已经有超过80种推测的DyP-type过氧化物酶在 PeroxiBase database(http:peroxibase.isbsib.ch/)中注册,超过400种同源蛋白可以通过PSI-Blast 检测到。
运动发酵单胞菌Zymomonas mobilis ZM4是一株重要的乙醇发酵细菌,以它高乙醇得率而受到研究者关注。那么除了它独特的 Entner-Doudoroff 途径外,是否自身可以产生某种酶保护细胞,防御及抵抗有毒物质的损害?通过RHA1 DypB 的系统进化分析,揭示Z.mobilis 中推断的过氧化物酶与DypB 有高同源性[23]。利用生物信息学方法进行分析,将Z.mobilis 中推断的过氧化物酶与其他DyP-type 过氧化物酶进行蛋白一级序列对比,它同样具有DyP-type 过氧化物酶的一些典型特征。为了证明Z.mobilis 中确实存在DyP-type 过氧化物酶基因,将Z.mobilis中的推断过氧化物酶基因首次通过序列分析并克隆到E.coli 中,继而将重组蛋白进行表达和纯化,并研究其性质,为这种新的DyP-type 过氧化物酶结构功能和反应机制研究奠定基础。
1 材料与方法
1.1 材料
Z.mobilis ZM4(ATCC 31821)菌株、E.coli DH5α和BL21(DE3)/pLysS 为本实验室保存。引物合成及蛋白MALDI-TOF-MS 测定委托深圳华大基因(BGI)完成。DNA 序列测定由上海生工生物工程技术服务有限公司完成。Pfu DNA 聚合酶、T4 DNA 连接酶和其他所有的限制性核酸内切酶购自Fermentas 公司。DNA 基因组提取试剂盒、DNA 纯化试剂盒、质粒提取试剂盒与凝胶回收试剂盒均购自OMEGA 公司。DNA 和蛋白marker 购自上海生工生物工程技术服务有限公司。pET21b(+)质粒购自Invitrogen 公司。琼脂糖金属螯合Ni 柱和PD-10脱盐柱购自GE 公司。其他试剂及药品均为进口或国产分析纯。
YMG 培养基(g/L):无水葡萄糖4,麦芽提取物10,酵母浸粉4,121℃灭菌20 min,用于Z.mobilis 培养。
LB 培养基(g/L):蛋白胨10,酵母浸粉5,氯化钠10,121℃灭菌20 min,用于E.coli 培养。
LB 选择培养基(g/L):蛋白胨10,酵母浸5,氯化钠10,121℃灭菌20 min,冷却后加入Amp,使其终浓度为100μg/mL,用于E.coli 转化子培养。
对于固体培养基,在液体培养基中加入1.5%琼脂粉。
2YT 培养基(g/L):蛋白胨16,酵母浸粉10,氯化钠5,121℃灭菌20 min。
1.2 方法
1.2.1 pET21b-ZmDyP 载体的构建
Z.mobilis中Putative DyP-type过氧化物酶基因(GenBank Accession No.Q5NM63) DNA 序列从NCBI 细菌基因组数据库中得到。以序列信息为依据,设计引物扩增编码区基因片段(表1)。引物1中,为了遵守引物设计原则,保证GC 含量在合适的范围内,将引物中个别碱基进行替换(单个碱基下有下划线代表原始碱基被替换),但翻译后不改变氨基酸残基。
以Z.mobilis 基因组DNA 作为模板,F1和R1为引物(表1),使用Pfu DNA 聚合酶进行扩增。扩增时各组分含量为:0.4 mmol/L F1,0.4 mmol/L R1,100 ng DNA 模板,0.2 mmol/L dNTPs,4% DMSO,1×Buffer,1 U Pfu DNA 聚合酶。PCR 条件:95℃5 min;94℃1 min,55℃30 s,72℃2 min,30个循环;72℃10 min。将扩增的DNA 进行纯化,作为模板,进行第二次扩增,使用F2和R2为引物(表1)。PCR 参数及条件如第一次扩增。将扩增的DNA 片段进行纯化,用NdeⅠ和XhoⅠ进行消化,再次纯化,然后与NdeⅠ-XhoⅠ粘性末端的pET-21b(+)载体进行连接,成为一个整合有目的片段的质粒pET21b(+)-ZmDyP (图1)。将pET21b(+)-ZmDyP转化入E.coli DH5α感受态细胞,通过氨苄青霉素抗性和蓝白斑筛选单克隆进行扩增培养,提取质粒进行PCR 和XhoⅠ和NdeⅠ双酶切验证后进行DNA 测序。
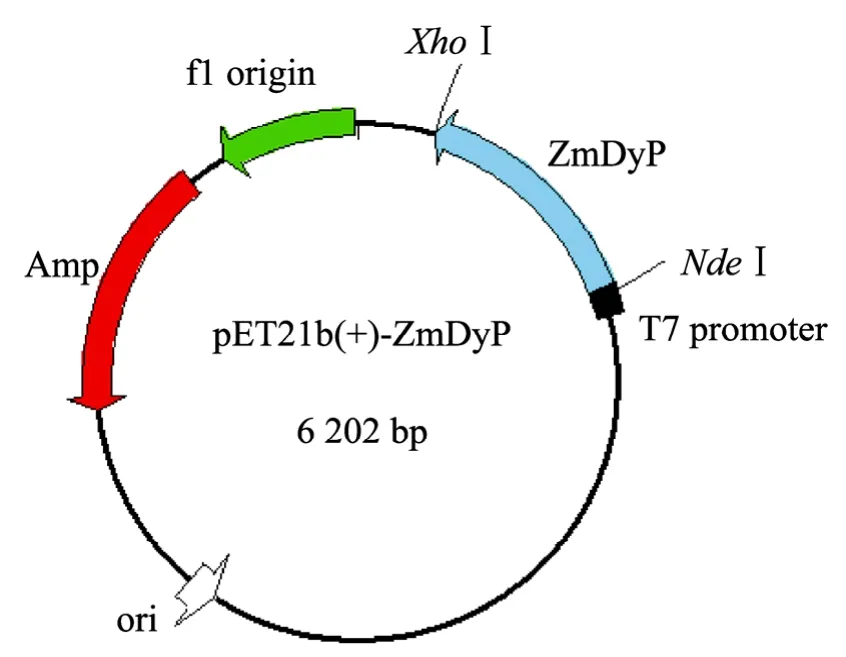
图1 重组质粒pET21b(+)-ZmDyP 载体结构示意图Fig.1 Map of the recombinant pET21b(+)-ZmDyP.
1.2.2 序列分析
多序列对比网址 www.ebi.ac.uk/Tools/msa/clustalw2/。
1.2.3 ZmDyP 在E.coli BL21(DE3)/pLysS 中的重组表达
将测序正确的质粒pET-21b-ZmDyP 转化入E.coli BL21(DE3)/pLysS 中,然后挑取单克隆,接种在含有100μg/mL 氨苄青霉素的5 mL 2YT培养基中。培养条件是200 r/min、37℃过夜。然后将活化的细菌以1∶100(V/V)接种于15 mL 新鲜的2YT 培养基中,培养条件与上述一致,直到细菌OD600为0.8~1.0之间,加入诱导剂IPTG。诱导温度为30℃,诱导表达时间分别为1、2、3和4 h,从而确定最佳诱导时间。诱导时间定为4 h,IPTG 浓度分别为0.1、0.2、0.5、1.0 mmol/L,从而确定最佳IPTG 诱导浓度。

表1 引物DNA 序列Table 1 DNA sequences of primers
在确定的最佳表达条件下,将菌株接种于500 mL 新鲜的2YT 培养基中进行扩大培养。IPTG 最终浓度0.2 mmol/L,菌液在30℃、200 r/min 条件下培养4 h。然后通过离心的方法收获菌体,用来进行下一步酶的纯化实验。
1.2.4 ZmDyP 的纯化
收获的菌体用缓冲液(Binding Buffer,25 mmol/L Tris-HCl,pH 7.5)溶解,缓冲液中含有0.5 mol/L NaCl 及1 mmol/L 苯甲基磺酰氟(PMSF)。用超声仪来破碎细胞,将细胞裂解液于12000× g、4℃条件下离心30 min 获得上清。在用金属螯合层析之前,先用0.4μm 过滤膜进行过滤,以除去杂质。将Ni-Chelating 琼脂糖柱用结合缓冲液进行平衡后上样,最后用含有50~200 mmol/L 咪唑的结合缓冲液进行洗脱。小部分收集的蛋白溶液可以通过颜色和SDS-PAGE 来判断纯度,然后合并在一起,用PD-10柱子进行除盐。最后通过超滤装置将蛋白进行浓缩。
1.2.5 ZmDyP 的PAGE 及MALDI-TOF MS 分析
蛋白浓度定量使用Bradford Assay 方法进行测定,SDS-PAGE 胶的蛋白显色使用Coomassie G-250。利用PAGE 分析的方法进行蛋白活性的测定,将没有加热的和加热的蛋白样品进行SDS-PAGE,电泳结束后,用25 mmol/L Tris-HCl将胶冲洗1次,然后每隔20 min 用50 mmol/L醋酸钠缓冲液冲洗,共冲洗2次,再将胶放在含有2.5 mmol/L 底物的50 mmol/L 醋酸钠缓冲液中,并加入最终浓度为0.1 mmol/L 的H2O2共同孵育10 min。
蛋白样品经除盐后浓缩到大约10 mg/mL。然后将样品与等体积的芥子酸混合,用Bruker UltrafleXtrem MALDI-TOF 质谱仪测定,通过Flexanalysis 3.3软件进行数据分析。
1.2.6 ZmDyP 的光谱分析
使用 Thermo Scientific Varioskan Flash 96-well Plate Reader 进行300 nm 到700 nm 的波长扫描。使用96孔板时,每个孔为上样200μL,包含40μmol/L ZmDyP 和25 mmol/L Tris-HCl。进行第1次读数扫描,扫描后加2μL 10 mmol/L H2O2到样品中,混合后立即进行第2次读数扫描。10 min 后进行第3次扫描。
1.2.7 酶活力测定
一个酶活力单位定义为:在25℃、pH 4.5条件下,氧化1μmol/L ABTS 所需要的酶量。以ABTS 为底物在96孔板中进行酶活力测定。在标准200μL 体系中,反应混合液包括50 mmol/L醋酸钠缓冲液,2.5 mmol/L ABTS,0.1 mmol/L H2O2和0.1μmol/L ZmDyP。ZmDyP、ABTS 和缓冲液在25℃预温育5 min,然后通过加入H2O2启动反应。反应过程的动力学曲线在414 nm 及25℃恒温条件下进行检测。
终点酶活力测定方法:反应混合体系包含同样浓度的底物和酶,加入H2O2启动反应,在25℃或其他特定温度下反应5 min,然后通过加入200μL 2% SDS 终止反应。将样品在414 nm 进行吸光度测定。Km和催化常数(kcat)通过非线性Michaelis-Menten 模型方程:v=kcat[E]t[S]/(Km+[S])拟合得到,相应图表用软件 Prism Gradpad Version 6绘制。为了测定pH 对酶活力的影响,如上述实验操作,但在pH 2.5~9.5条件下进行测定。酶的热稳定性的测定:酶在25℃~75℃条件下孵育20 min,然后在标准条件下测定酶的剩余酶活力。
2 结果与分析
2.1 pET21b(+)-ZmDyP 载体的验证
以Z.mobilis 基因组DNA 为模板进行PCR,2%琼脂糖凝胶电泳检测。可见ZmDyP 的PCR产物在1000 bp 附近有特异性条带,片段大小与GenBank 中登录的相符。将重组质粒pET21b(+)-ZmDyP 进行酶切验证,所切下的片段与预期结果相符(图2),证明整合载体构建成功。
2.2 序列分析
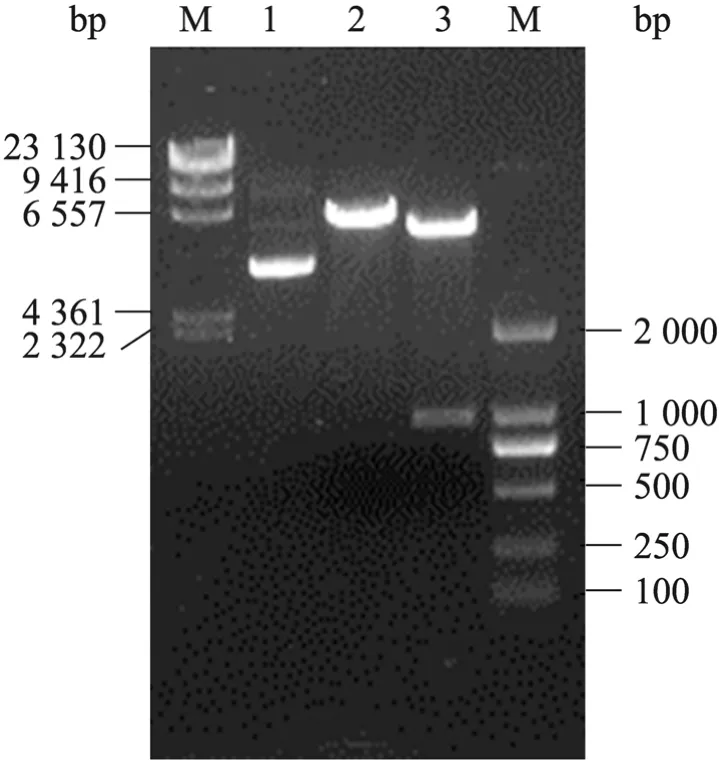
图2 重组质粒pET21b(+)-ZmDyP 的酶切验证Fig.2 Verification of the recombinant plasmid pET21b(+)-ZmDyP by enzyme digestion.M:DNA marker;1:pET21b(+)-ZmDyP;2:pET21b(+)-ZmDyP digested with Nde I;3:pET21b(+)-ZmDyP digested with Nde I and Xho I.
通过 Clustal W 对比扩增 DNA 序列和GenBank 中得到的序列,结果完全一致,证明了扩增基因的正确性。将ZmDyP 的一级序列与其他4种DyP-type 过氧化物酶(DyPPa,RAH1 DypB,BtDyP 和TyrA)序列进行多重序列比对(图3)。可见,ZmDyP 与其他DyP-type 过氧化物酶一样都存在GXXDG 这种保守结构序列。除了GXXDG 结构外,还有一些保守氨基酸残基(D149,R239,T254和F256)*,形成一个活性口袋来结合H2O2。对于传统意义上的过氧化物酶来说,His 在催化过程中起到重要作用,而DyP-type 过氧化物酶中Asp 代替His 起到这个作用。从真菌T.cucmeris Dec 1中得到的晶体结构显示DyP-type 过氧化物酶的催化机制可能涉及到保守氨基酸Asp (D171)残基的摇摆运动[15]。Roberts 等表征了RHA1 DypB 的晶体结构[25],在酶的亚铁血红素口袋中发现了(D153,H226,R244,N246和D288)这5个重要氨基酸残基,而它们在ZmDyP 中同样也是保守的。ZmDyP 与其他一些DyP-type 过氧化物酶家族成员的这些共同结构特征显示它也是一种DyP-type 过氧化物酶。由此,称它为ZmDyP (Z.mobilis DyP)。
2.3 ZmDyP 在大肠杆菌BL21(DE3)/pLysS 中表达条件的优化
诱导温度定为30℃,IPTG 浓度为0.2 mmol/L,诱导表达时间分别为1、2、3和4 h,结果如图4A 所示。由于诱导表达时间3 h 和4 h时表达水平差不多,但4 h 培养菌体生物量较高,可以满足后续研究工作需要,从而确定本研究的最佳诱导时间为4 h。IPTG 浓度分别为0.1、0.2、0.5和1.0 mmol/L 的实验结果如图4B 所示,可见IPTG 的浓度对于表达的影响并不明显,所以采用IPTG 诱导浓度为0.2 mmol/L。
2.4 C 端带有6×His tag ZmDyP 的纯化
ZmDyP 是从质粒pET-21b-ZmDyP 中表达的,在质粒的C 端有6×His tag 融合蛋白。该酶是一种可溶的胞内蛋白,超声后溶解在细胞破碎产物中,可利用Ni-chelating 树脂进行纯化。通过扩大培养,1.5 L 2YT 培养基可产大约7.5 g 细菌(湿重),经纯化后计算各步骤总蛋白、总酶活、比酶活及回收率(表2)。
2.5 ZmDyP 的PAGE 及MALDI-TOF MS分析
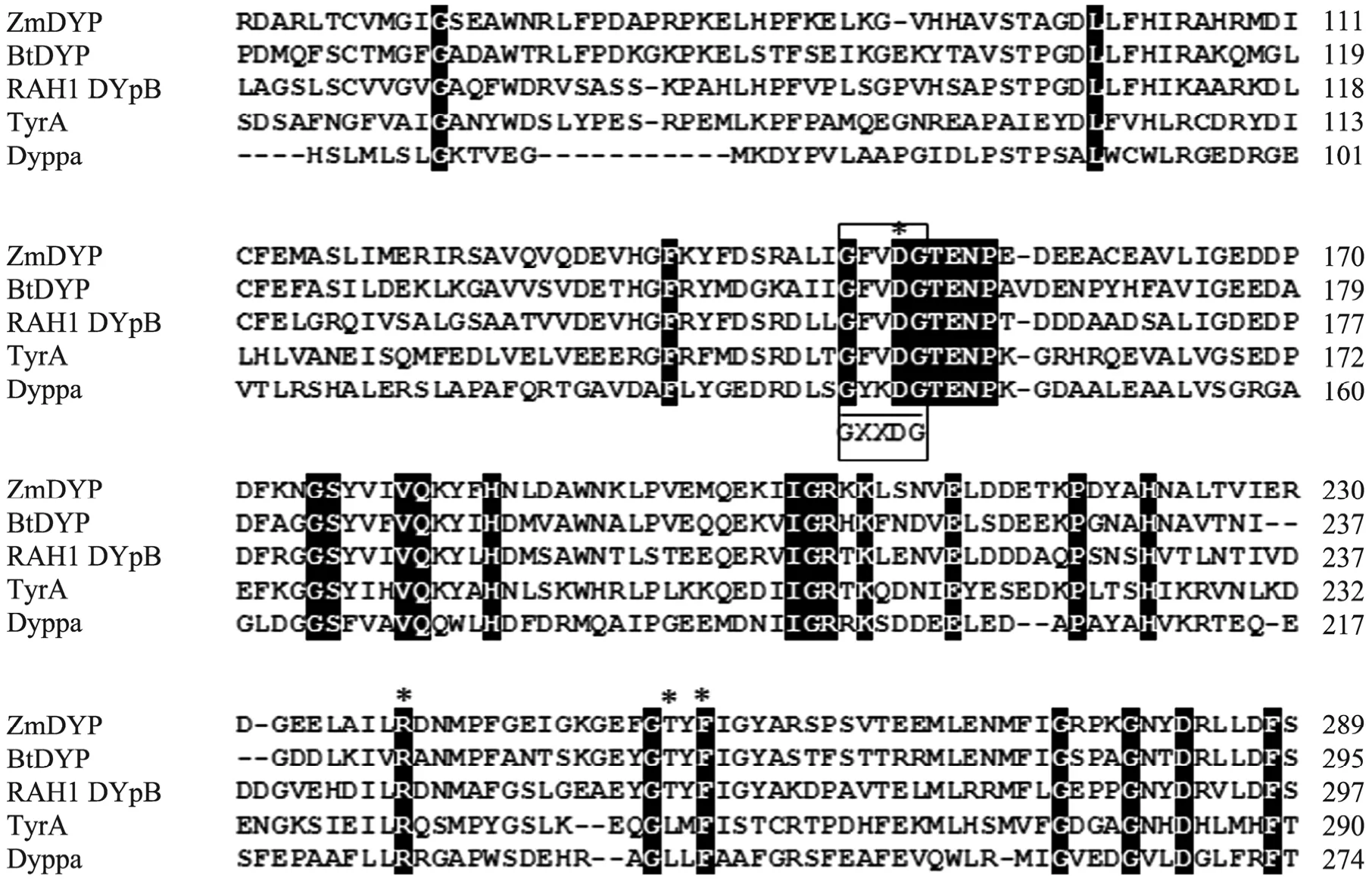
图3 ZmDyP 与其他DyP-type 过氧化物酶的多重序列比对Fig.3 Multiple sequence alignment of ZmDyP with other members of DyP-type peroxidases family.BtDyP:Bacteroides thetaiotaomicron (Accession No.AA076326);RHA1 DypB:Rhodococus jostii RHA1(Accession No.Q0SE24);TyrA:Shewanella oneidensis MR-1(Accession No.AAN53816).DyPPa:Pseudomonas aeruginosa PKE117(Accession No.ADF43017).Identical residues are shown as white in black shade.The unique GXXDG motif characteristic of DyP-type peroxidases and conserved residues of the active site are indicated by box and asterisks,respectively.

图4 ZmDyP 在大肠杆菌BL21(DE3)/pLysS 中的表达及SDS-PAGE 分析Fig.4 SDS-PAGE analysis of ZmDyp expression in E.coli BL21(DE3) pLyS.Effect of induction time (A) and IPTG concentration (B) on ZmDyP expression.
将超声破碎的菌体全蛋白和纯蛋白进行还原型12% SDS-PAGE,结果如图5A 所示,可以看到ZmDyP 展示了一条清晰的条带,分子量大约为36 kDa。而在没有还原剂的条件下,10% SDS-PAGE 也出现了图5B 所示的同样分子量的一条带,由此可以判断该酶没有分子间二硫键。ZmDyP 通过MALDI-TOF MS 显示分子量是35812.21 Da,经氨基酸预测分子量是35745.49 Da。将SDS-PAGE 胶进行酶活力染色,可以看到在116 kDa区域的条带(无加热,图5C,泳道1),但却没有检测到单体的形式(加热,图5C,泳道2)。结果表明ZmDyP 分子结构是很紧密的,甚至暴露在有SDS 存在的SDS-PAGE系统中,也是以具有酶活力的三聚体的形式存在。根据SDS-PAGE 测定的ZmDyP 相对分子质量,可以得到线性回归曲线为y=2.22-0.0188x,R2=0.9978。经以上研究确定ZmDyP 单体蛋白分子量约为36 kDa,三聚体蛋白约为108 kDa。
细菌 DyP-type 过氧化物酶单体分子量从32 kDa 到54 kDa 不等,当然也会以多聚体的形式存在,例如,绿脓杆菌Pseudomonas aeruginosa中的酶是64 kDa 的二聚体[24],蓝细菌鱼腥藻cyanobacterium Anabaena 中的酶是206 kDa 的四聚体[21]。因此,这些细菌的亚铁血红素过氧化物酶是以不同的大小和不同多聚体的形式而存在的。

表2 纯化得率及比活力测定Table 2 Purification yield and enzyme activity analysis
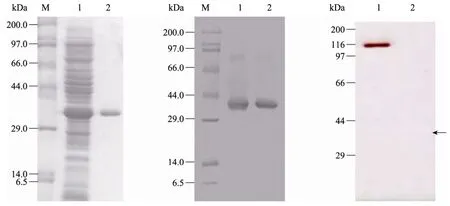
图5 SDS-PAGE 分析纯化的重组酶ZmDyPFig.5 SDS-PAGE analysis of purified recombinatant ZmDyP.(A) Commassie blue-stained gel showing the purification of ZmDyP.M:molecular weight markers;1:soluble fraction of cell lysate prepared from BL21(DE3)/pLysS expressing ZmDyP;2:ZmDyP purified by Ni-chelating Sepharose chromatography.(B)Identificaton of intramolecular disulfide bond gel.M:molecular weight markers;1:ZmDyP without DTT;2:ZmDyP with DTT.(C) Activity-stained gel showing the size of ZmDyP.M:molecular weight markers;1:ZmDyP without heating;2:ZmDyP with heating.
2.6 ZmDyP 的光谱分析
纯化后的ZmDyP 溶液呈棕红色,暗示着在酶的活性位点存在着亚铁血红素基团。酶在400 nm 处有一个明显的亚铁血红素酶的特征吸收峰Soret band (图6),这在其他DyP-type 过氧化物酶中也是普遍存在的[21-22,25]。在酶中加入H2O2会导致400 nm 和510 nm 吸收峰降低。10 min 后,酶又逐渐回到静息状态的趋势。这种现象在其他的含亚铁血红素过氧化物酶中也有报道[11,25]。因此,更加确定了ZmDyP 确实是一种含有亚铁血红素的过氧化物酶。
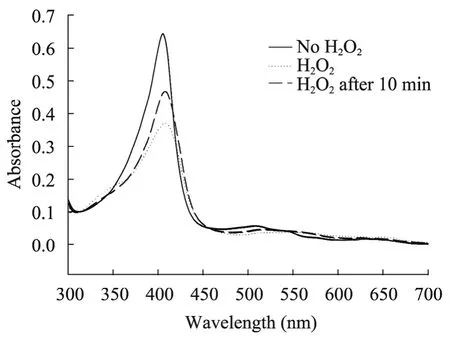
图6 ZmDyP 氧化H2O2的光谱吸收峰Fig.6 Spectrophotometric analysis of ZmDyP oxidation of H2O2.
2.7 酶学性质评价
pH 对ZmDyP 酶活力的影响,是以ABTS 为底物,在不同pH (2.5~9.5)缓冲液中进行的,结果如图7A 所示。最大酶活力是在pH 4.0,pH 4.5以上酶活力会急剧下降。因为酶在pH 4.0反应速度太快,为了实验数据的准确性,将ZmDyP 标准状况下酶活力的测定设为pH 4.5。对于大多数亚铁血红素依赖的过氧化物酶而言,无论来源是细菌[22,24]还是真菌[26-27],最佳活力都在pH 4.0左右,因为酸性pH 能增加亚铁血红素的氧化能力[28]。温度对ZmDyP 酶活力的影响,在标准测定条件下,通过25℃~75℃的温度范围进行测定,结果如图7B 所示,可见最大酶活力是在37℃,比25℃高出大约10%的活力。在37℃以上,酶活力开始下降,75℃没有检测到酶活力。热稳定性实验结果如图7C 所示,可见ZmDyP表现中等热稳定性,在50℃加热20 min 后,大约有85%的剩余酶活力。在55℃以上时,酶表现得很不稳定,在75℃以上酶活力几乎完全丧失。ZmDyP 的热稳定性与其他细菌如Anabaena[21]和P.aeruginosa[24]的DyP-type 过氧化物酶是相近的。
ZmDyP 是基于E.coli 的重组酶,它的底物特异性用表3所示的几种化合物来检测。在这些底物中,该酶对2,2-二氨-双(3-乙基苯并噻唑-6-磺酸)(ABTS)棓表现出最高酶活力,焦酸(Pyrogallol)表现出最低酶活力,两者相差13倍。细菌R.jositii RHA1中的DypB 分别以ABTS 和Pyrogallol 为底物时,对ABTS 表现出的酶活力比Pyrogallol 高出16倍[25],但真菌Anabaena sp.strain PCC 7120中的 AnaPX 以 ABTS 和Pyrogallol 为底物时,表现出的酶活力却是相当水平[25]。藜芦醇(Veratryl alcohol)是一种木质素过氧化物酶的模式底物,在DyP-type 过氧化物酶中目前只有真菌来源的 AjP 和细菌来源的DypB 和TfuDyP 对它表现出活性[22,25,27]。对于底物蒽醌染料(Reactive blue 4)和三芳基甲烷类染料(Crystal violet),ZmDyP 表现出弱活性。对偶氮类染料(Azure B)、丁香酸(Syringic acid)、香草酸(Vanillic acid)、愈创木酚(Guaiacol)都无活性。与其他一些已报道的DyP-type 过氧化物酶来比,ZmDyP 呈现出较窄的底物特异性。

图7 ZmDyP 的最佳pH 和温度及热稳定性Fig.7 Determination of optimal pH (A) and temperature dependence of ZmDyP assayed with ABTS as substrate using the endpoint assay (B),and the enzyme’s thermal stability evaluated by incubating at different temperatures for 20 min and its residual activity was determined under standard conditions (C).
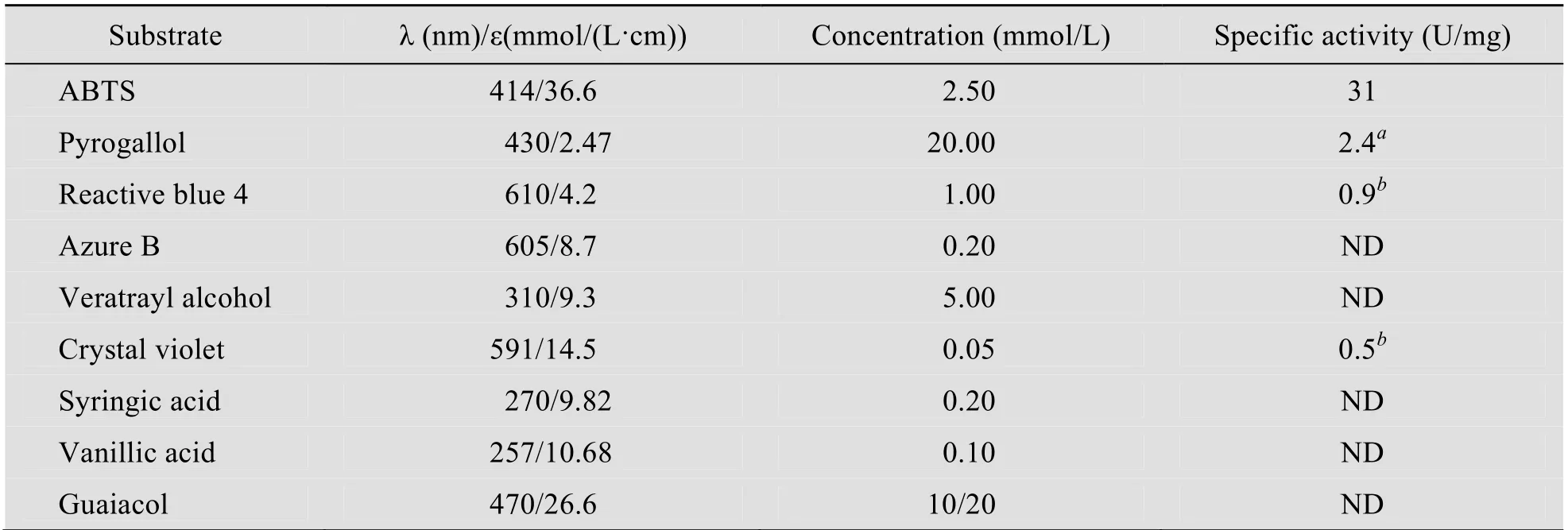
表3 重组酶ZmDyP 的底物特异性Table 3 Substrate specificity of the recombinant ZmDyP
3 讨论
Z.mobilis 是一株重要的乙醇生产菌株,它可以通过Entner-Doudoroff 途径生产乙醇,与酵母相比消耗同等质量的葡萄糖产生更少的ATP,因此生物量合成较少,相应可以提高乙醇对糖的表观收率,可能成为发酵生产乙醇的一个优势菌株[29]。那么Z.mobilis 的高乙醇得率是否和自身的氧化应激反应有关?H2O2是氧化胁迫的一个来源,它能够形成活性氧自由基,比如羟基自由基和单氧自由基。这些自由基能够损害细胞一些重要的组成部分,比如DNA、蛋白和脂类等[30-31]。通过光谱分析证明ZmDyP 可以将H2O2作为底物清除掉,这样就会减少一些氧化自由基对菌体的损害。
本研究首次通过序列分析将Z.mobilis 中的推断 DyP-type 过氧化物酶基因插入到pET-21b(+)表达载体中,通过转化E.coli 得到表达并纯化出可溶性目的蛋白。通过Clustal W与其他DyP-type 过氧化物酶进行序列分析,证明ZmDyP 中存在GXXDG 保守序列结构和一些保守氨基酸残基(D149,R239,T254和F256)。通过PAGE 及MALDI-TOF MS 分析,ZmDyP 单体蛋白分子量约为36 kDa,在有活性状态下是以同源三聚体形式存在,分子量约为108 kDa。光谱分析表明,酶在400 nm 处有一个明显的亚铁血红素酶的特征吸收峰,并且能够以H2O2作为底物。
以上结果证实 Z.mobilis 中确实存在DyP-type 过氧化物酶基因,并且这种酶可以清除氧化自由基。酶活力测定表明,ZmDyP 最适pH为pH 4.0,最适反应温度为37℃,表现出中等热稳定性。与RHA1 DypB 进行催化效率比较,表明ZmDyP 是一种有更高催化效率的酶。通过对 ZmDyP 表达纯化及性质的研究,丰富了DyP-type 过氧化物酶家族的信息,为进一步研究ZmDyP 生理功能及后续的结构和反应机制奠定基础。
[1]Passardi F,Cosio C,Penel C,et al.Peroxidases have more functions than a Swiss army knife.Plant Cell Rep,2005,24(5):255−265.
[2]Davies MJ,Hawkins CL,Pattison DI,et al.Mammalian heme peroxidases:from molecular mechanisms to health implications.Antioxid Redox Signal,2008,10(7):1199−1234.
[3]Almagro L,Gómez Ros LV,Belchi-Navarro S,et al.Class III peroxidases in plant defence reactions.J Exp Bot,2009,60(2):377−390.
[4]Park JG,Oh GT.The role of peroxidases in the pathogenesis of atherosclerosis.BMB Rep,2011,44(8):497−505.
[5]Robinson T,McMullan G,Marchant R,et al.Remediation of dyes in textile effluent:a critical review on current treatment technologies with a proposed alternative.Bioresour Technol,2001,77(3):247−255.
[6]Shah V,Nerud F.Lignin degrading system of white-rot fungi and its exploitation for dye decolorization.Can J Microbiol,2002,48(10):857−870.
[7]Matsunaga I,Shiro Y.Peroxide-utilizing biocatalysts:structural and functional diversity of heme-containing enzymes.Curr Opin Chem Biol,2004,8(2):127−132.
[8]Husain Q.Potential applications of the oxidoreductive enzymes in the decolorization and detoxification of textile and other synthetic dyes from polluted water:a review.Crit Rev Biotechnol,2006,26(4):201−221.
[9]Qayyum H,Maroof H,Yasha K.Remediation and treatment of organopollutants mediated by peroxidases:a review.Crit Rev Biotechnol,2009,29(2):94−119.
[10]Kim SJ,Ishikawa K,Hirai M,et al.Characteristics of a newly isolated fungus,Geotrichum candidum Dec 1,which decolorizes various dyes.J Ferment Bioeng,1995,79(6):601−607.
[11]Kim SJ,Shoda M.Purification and characterization of a novel peroxidase from Geotrichum candidum Dec 1 involved in decolorization of dyes.Appl Environ Microbiol,1999,65(3):1029−1035.
[12]Johjima T,Ohkuma M,Kudo T.Isolation and cDNA cloning of novel hydrogen peroxide-dependent phenol oxidase from the basidiomycete Termitomyces albuminosus.Appl Microbiol Biotechnol,2003,61(3):220−225.
[13]Faraco V,Piscitelli A,Sannia G,et al.Identification of a new member of the dye-decolorizing peroxidase family from Pleurotus ostreatus.World J Microbiol Biotechnol,2007,23(6):889−893.
[14]Scheibner M,Hülsdau B,Zelena K,et al.Novel peroxidases of Marasmius scorodonius degrade beta-carotene.Appl Microbiol Biotechnol,2008,77(6):1241−1250.
[15]Yoshida T,Tsuge H,Konno H,et al.The catalytic mechanism of dye-decolorizing peroxidase DyP may require the swinging movement of an aspartic acid residue.FEBS J,2011,278(13):2387−2394.
[16]Sato T,Hara S,Matsui T,et al.A unique dye-decolorizing peroxidase,DyP,from Thanatephorus cucumeris Dec 1:heterologous expression,crystallization and preliminary X-ray analysis.Acta Crystallogr D Biol Crystallogr,2004,60(pt1):149−152.
[17]Zubieta C,Krishna SS,Kapoor M,et al.Crystal structures of two novel dye-decolorizing peroxidases reveal a beta-barrel fold with a conserved heme-binding motif.Proteins,2007,69(2):223−233.
[18]Zubieta C,Joseph R,Krishna SS,et al.Identification and structural characterization of heme binding in a novel dye-decolorizing peroxidase,TyrA.Proteins,2007,69(2):234−243.
[19]Sturm A,Schierhorn A,Lindenstrauss U,et al.YcdB from Escherichia coli reveals a novel class of tat-dependently translocated hemoproteins.J Biol Chem,2006,281(20):13972−13978.
[20]Ebihara A,Okamoto A,Kousumi Y,et al.Structure-based functional identification of a novel heme-binding protein from Thermus thermophilus HB8.J Struct Funct Genomics,2005,6(1):21−32.
[21]Ogola HJ,Kamiike T,Hashimoto N,et al.Molecular characterization of a novel peroxidase from the cyanobacterium Anabaena sp.strain PCC 7120.Appl Environ Microbiol,2009,75(23):7509−7518.
[22]Van Bloois E,Torres Pazmiño DE,Winter RT,et al.A robust and extracellular heme-containing peroxidase from Thermobifida fusca as prototype of a bacterial peroxidase superfamily.Appl Microbiol Biotechnol,2010,86(5):1419−1430.
[23]Ahmad M,Roberts JN,Hardiman EM,et al.Identification of DypB from Rhodococcus jostii RHA1 as a lignin peroxidase.Biochemistry,2011,50(23):5096−5107.
[24]Li J,Liu C,Li B,et al.Identification and molecular characterization of a novel DyP-type peroxidase from Pseudomonas aeruginosa PKE117.Appl Biochem Biotechnol,2012,166(3):774−785.
[25]Roberts JN,Singh R,Grigg JC,et al.Characterization of dye-decolorizing peroxidases from Rhodococcus jostii RHA1.Biochemistry,2011,50(23):5108−5119.
[26]Shin K,Oh I,Kim C,et al.Production and purification of remazol brilliant blue R decolorizing peroxidase from the culture filtrate of Pleurotus ostreatus.Appl Environ Microbiol,1997,63(5):1744−1748.
[27]Liers C,Bobeth C,Pecyna M,et al.DyP-like peroxidases of the jelly fungus Auricularia auricula-judae oxidize nonphenolic lignin model compounds and high-redox potential dyes.Appl Microbiol Biotechnol,2010,85(6):1869−1879.
[28]Oyadomari M,Shinohara H,Johjima T,et al.Electrochemical characterization of lignin peroxidase from the white-rot basidiomycete Phanerochaete chrysosporium.J Mol Catal B:Enzym,2003,21(4-6):291−297.
[29]Bai FW,Anderson WA,Moo-Young M.Ethanol fermentation technologies from sugar and starch feedstocks.Biotechnol Adv,2008,26(1):89−105.
[30]Miller RA,Britigan BE.Role of oxidantsin microbial pathophysiology.Clin Microbiol Rev,1997,10(1):1−18.
[31]Hofer T,Badouard C,Bajak E,et al.Hydrogen peroxide causes greater oxidation in cellular RNA than in DNA.Biol Chem,2005,386(1):333−337.
