FOX基因捕获技术在植物中的研究进展与应用
2013-06-09刘亚运吴金霞路铁刚
刘亚运, 吴金霞, 路铁刚
中国农业科学院生物技术研究所,北京100081
FOX基因捕获技术在植物中的研究进展与应用
刘亚运, 吴金霞, 路铁刚∗
中国农业科学院生物技术研究所,北京100081
利用功能获得突变技术已在模式植物拟南芥与水稻中获得了大量的突变群体,通过对这些突变群体进行研究分析,极大促进了拟南芥与水稻基因功能的研究。其中FOX基因捕获技术是利用单一或者有限数量的全长cDNA使植物基因异位表达,从而系统地获得功能获得型突变体。FOX技术不但在拟南芥、水稻等模式植物中的应用日趋成熟,同时在其他植物物种内的研究也已取得极大的进展。
功能获得突变;FOX基因捕获技术;研究进展
在全基因组水平上诱发突变体的产生以及突变体材料的收集为功能基因组学的研究搭建了技术平台[1]。目前在全基因组水平上利用物理化学诱变[2]、插入突变包括T⁃DNA插入突变[3]和转座子插入突变[4]等方法创制功能缺失型突变体以及利用激活标签技术(activation tagging technol⁃ogy)和FOX捕获系统(full⁃length cDNA over⁃ex⁃pression gene hunting system)创制功能获得型突变体[5~8]已经成为人工创制突变体的主要途径。本文着重介绍了FOX捕获系统的基本原理以及在研究植物基因功能方面的应用。FOX系统利用单一或者有限数量的全长cDNA使植物基因异位表达,从而系统地获得功能获得型突变体。
1 FOX技术的原理与创建
激活标签系统是一种有效建立大规模功能获得突变体库的手段,但是这种系统具有一些缺点,因为在基因组上插入转录增强子的效果受临近增强子的多个基因的变化影响[9]。最近几年来,多个种类的植物fl⁃cDNA(全长cDNA)克隆已经获得。拟南芥fl⁃cDNA群体,也被称为“RIKEN拟南芥全长(RAFL)cDNA克隆”,已经被构建并测序完成[10]。Ichikawa等[5]在此基础上开发了一种新型的功能获得性研究方法,并命名为“the FOX gene hunting system”,利用单一或者有限数量的全长cDNA使植物基因异位表达,从而系统地获得功能获得型突变体。该技术可以产生大量的显性突变体。拟南芥FOX库的构建流程可见图1。
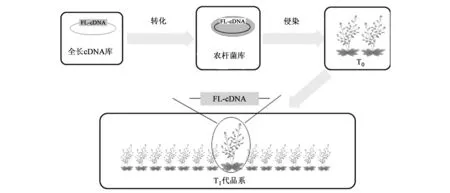
图1 FOX gene hunting system的基本技术路线Fig.1 The basic technical line of FOX gene hunting system.
2 FOX技术的发展与应用
2.1 FOX技术在模式植物拟南芥与水稻中的发展与应用
Ichikawa等[5]将大约10 000个独立的拟南芥cDNA与CaMV 35S控制下的转化载体相连结,通过遗传转化将这个cDNA表达库导入拟南芥体内,从而得到大概 15 000个表达拟南芥全长cDNA的转基因植株(拟南芥FOX⁃line)。这些全长cDNA被整合到一个植物表达载体中构建了一个FOX农杆菌库。通过浸花法把每个拟南芥全长cDNA转化到拟南芥植株中。Sothern杂交检测分析表明每个转基因系平均含有2.6个cDNA克隆,大小在0.3~4.2 kb之间,平均为1.4 kb。cDNA的大小平均值和分布范围与在拟南芥fl⁃cDNA中随机挑选的 277个 fl⁃cDNA非常接近[5]。FOX技术可以使每个插入基因通过表型得到系统的分析研究。这些明显的表型包括:形态、生长速率、叶片颜色、花期和生育力的明显改变。在拟南芥FOX⁃line中,形态上的突变体(总转化植株的9%)的出现高于激活标签体系(总转化植株的2%)。
阐明胁迫相关基因的功能是该技术的主要应用之一。通过构建一个包括各种基于胁迫表达的转录因子文库(小规模FOX系统)并且进行分析[6],拟南芥中 43个胁迫诱导的转录因子 fl⁃cDNA被混合并转化224个植株,经过高盐胁迫培养观察,所有的耐盐系都存活并保持叶片绿色。由于拟南芥拥有高转化效率和短生长周期的优点,可以利用拟南芥作为FOX捕获系统的寄主用以阐明其他植物的基因功能。
多达28 000条独立的水稻fl⁃cDNA已经被农业生物资源研究所(National Institute of Agrobio⁃logical Sciences,NIAS,日本)收集[11]。利用约13 000条独立的水稻fl⁃cDNA,水稻⁃拟南芥FOX转基因系统已经构建成功。这些转基因系是在CaMV35S启动子控制下,水稻fl⁃cDNA转化拟南芥而构成的[7]。Koudou等[7]已经建立了多达23 000个独立的水稻FOX拟南芥转基因系。PCR检测分析,平均每个植物包括 1.1个水稻fl⁃cDNA,其大小在0.5~4.5 kb之间。这些水稻FOX拟南芥转基因系被用于大规模的表型检测,如:可见的表型、光合作用活性、矿质元素积累、色素积累、激素功能、抗生物胁迫、耐盐胁迫与耐高温胁迫、抗紫外胁迫与高光耐受等。在可见表型的突变体,1 297个突变系(约5%)被从23 715个T1代转基因系中分离出来。从这些突变体扩增出fl⁃cDNA重新转入野生型拟南芥中,大约有一半表型重现(48%)。其中的一些水稻fl⁃cDNA被转入水稻中,22%的转基因后代产生了在拟南芥FOX系中检测到的同样表型。
Nakamura等人[8]建立了将近12 000个表达水稻fl⁃cDNA的水稻FOX⁃lines,每个水稻FOX⁃line平均含有1.04个水稻fl⁃cDNA,平均大小为1.66 kb。通过对238个水稻FOX⁃lines的检测表明,其平均FOX⁃lines大小接近原本水稻fl⁃cDNA库的平均大小。在 T0代就看见明显的突变体。在9 012个FOX⁃lines中共有1 496个转基因系表型发生了明显的改变。在水稻FOX⁃lines出现的表型主要有:矮化、致死、类病变、叶色浅、愈伤组织绿化、叶窄和高倍分蘖等。
2.2 利用FOX技术对其他植物物种中的研究应用
尽管对于基因功能的分析基本都聚焦在拟南芥与水稻这些模式植物中,但许多其他物种在特定酶的作用下产生特异的化学物或者综合代谢产物。随着测序技术的发展,许多植物物种的基因组测序已完成。然而,有些特别植物的基因功能研究仍进展缓慢,并且这些物种的转化很困难。这些作物基因功能的分析研究通常让研究者们花费大量的时间和精力在植株的转化和组织培养上。FOX技术为此带来了更方便的研究方法。对于具有较大基因组的物种如玉米和小麦来说,这种方法具有更大的优势。因此,FOX技术不仅是一种好的基础研究方法,而且在植物相关有益表型的研究方面具有极大的潜力。FOX技术得到的后代所引起的表型变化是由基因插入引起的,因为fl⁃cDNA是在CaMV 35S启动子以及NOS终止子的之间正向表达[12]。最近,从黄豆、木薯、白杨和日本柳杉等多种植物中提取的fl⁃cDNA已经克隆得到[13~15]。FOX捕获系统可能会是获取这些植物的功能获得型突变体的一种替代手段。例如在拟南芥中,利用饱和诱变的功能缺失研究和FOX捕获系统的功能获得研究可以用来进行基于基因的表型分析。在水稻中的相关研究发展也已经迅速的接近拟南芥。随着实验技术与生物信息学的发展,在许多其他作物或者蔬菜的研究也已经开展,这对改善粮食的供应与质量的提高有着重要意义。
2.3 FOX技术的相关资源与数据库
根据FOX技术已获得的材料,科学家们构建了相应的数据库资源,这些数据库中不但包含了转基因植株的全长cDNA插入位点,还囊括了转基因后代的一共8个大类24个子类的显著表型。这些数据库的更新信息都能从水稻功能基因数据库中查明(RiceGE)(http://signal.salk.edu/cgi⁃bin/RiceGE)。FOX技术相关的公共数据库信息见表1。

表1 FOX技术相关的公共数据库Table 1 The databases of FOX gene hunting system.
3 展望
各种突变体资源的利用都促进了大规模的表型分析的发展。基本上,功能缺失突变可因稳定的遗传而且可以达到饱和诱变。基于突变资源的基因型-表型分析是可行的,这点已经在拟南芥和水稻中得到了充分的验证。一旦在缺失突变中找到明显的表型,就能够分离出直接导致这个表型的基因。然而,只有极少数的突变有明显的表型,特别是单基因突变,这是因为重复基因或者基因家族的功能冗余所造成的结果。在拟南芥中基因组中,超过65%的预测蛋白属于基因家族的成员,这些基因家族中每个至少含有两个成员。与此相不同的是,功能获得突变就可以克服功能冗余或致死表型所带来的难题[16]。一旦诱导基因的效应得到了证明,所获得的结果很快就能应用到作物中进行研究。然而如果得到一个表型清晰的功能获得突变体,与功能缺失突变相比,对于生物基因功能的研究并不容易,这是因为功能获得突变体的突变表型可能是由过表达基因的异位表达所造成。因此,功能获得突变的表型需要经过仔细的分析鉴定。在分析功能获得突变表型时也要考虑到由于多代繁殖中对应基因的共抑制所导致的基因沉默的可能性[17]。
FOX技术作为功能获得突变技术新兴的一种,不但对于模式植物的功能基因研究有着明显的优势,并且对于较难转化的物种的功能基因研究另辟蹊径。本实验室就曾利用FOX技术通过农杆菌介导在水稻中过表达小麦转录因子基因全长cDNA,获得了大量的具有明显表型的转基因FOX后代[],这对于小麦转录因子功能的研究以及水稻的品种改良都提供了广阔的研究空间。
[1] Kuromori T,Takahashi S, Kondou Y, et al.Phenome analysis in plant species using loss⁃of⁃function and gain⁃of⁃function mutants[J].Plant Cell Physiol.,2009,50(7):1215 -1231.
[2] Wu JL,Wu C,Lei C,et al..Chemical⁃and irradiation⁃in⁃duced mutants of indica rice IR64 for forward and reverse ge⁃netics[J].Plant Mol.Biol.,2005,59(1):85-97.
[3] Krysan P J,Young J C,Sussman M R.T⁃DNA as an insertionalmutagen in Arabidopsis[J].Plant Cell,1999,11(12):2283-2290.
[4] Fedoroff N V,Smith D L.A versatile system for detecting transposition in Arabidopsis[J].The Plant J.,1993,3(2):273-289.
[5] Ichikawa T,Nakazawa M,Kawashima M,et al.The FOX hunting system:an alternative gain⁃of⁃function gene hunting technique[J].The Plant J.,2006,48:974-985.
[6] Fujita M,Mizukado S,Fujita Y,et al..Identifi cation of stress⁃tolerance⁃related transcriptionfactor genes via mini⁃scale full⁃length cDNA over⁃expressor(FOX)gene hunting system[J].Biochem.Biophys.Res.Commun.,2007,364:250-257.
[7] Kondou Y,Higuchi M,Takahashi S,et al..Systematic ap⁃proaches to using the FOX hunting system to identify useful rice genes[J].The Plant J.,2009,57:883-894.
[8] Nakamura H,Hakata M,Amano K,et al..A genome⁃wide gain⁃of function analysis of rice genes using the FOX⁃hunting system[J].Plant Mol.Biol.,2007,65:357-371.
[9] Ichikawa T,Nakazawa M,Kawashima M,et al..Sequence da⁃tabase of 1172 T⁃DNA insertion sites in Arabidopsis activation⁃tagging lines that showed phenotypes in T1generation[J].The Plant J.,2003,36:421-429.
[10] Seki M,Narusaka M,Kamiya A,et al..Functional annotation of a full⁃length Arabidopsis cDNA collection[J].Science,2002,296:141-145.
[11] Kikuchi S,Satoh K,Nagata T,et al.Collection,mapping,and annotation of over 28 000 cDNA clones from japonica rice[J].Science,2003,301:376-379.
[12] Hayashi H,Czaja I,Lubenow H,et al..Activation of a plant gene by T⁃DNA tagging:auxin⁃independentgrowth in vitro[J].Science,1992,258:1350-1353
[13] Nanjo T,Sakurai T,Totoki Y,et al..Functional annotation of 19 841 Populus nigra fulllength enriched cDNA clones[J].BMC Genomics,2007,8:448.
[14] Futamura N,Totoki Y,Toyoda A,et al.Characterization of expressed sequence tags from a fulllength enriched cDNA library of Cryptomeria japonica male strobili[J].BMC Genomics,2008,9:383
[15] Taji T,Sakurai T,Mochida K,et al.Large⁃scale collection and annotation of full⁃length enriched cDNAs from amodel hal⁃ophyte,Thellungiella halophila[J].BMC Plant Biol.,2008,8:115.
[16] Tani H,Chen X,Nurmberg P,et al.Activation tagging in plants:a tool for gene discovery[J].Funct.Integr.Genomics,2004,4:258-266.
[17] Weigel D,Ahn JH,Blazquez M A,et al..Activation tagging in Arabidopsis[J].Plant Physiol.,2000,122:1003-1013
Advance and Application of FOX Gene Hunting System in Plant
LIU Ya⁃yun,WU Jin⁃xia,LU Tie⁃gang∗
Biotechnology Research Institute,Chinese Academy of Agricultural Sciences,Beijing 100081,China
Large numbers ofmutant plant populations have been generated by‘gain⁃of⁃function’techniques.Great progress have been made in Arabidopsis and rice through the use of these tools.Using a single or a limited number of full length cDNAs,FOX system makes gene ectopic expression in plant,accessing functionalmutants,especially the gain⁃of⁃function mutant.The FOX gene hunting system have beenmaturely used notonly inmodel plantspecies,such as Arabidopsis and rice,butalso in other plant species.
gain⁃of⁃function mutants;FOX gene hunting system;progress
10.3969/j.issn.2095⁃2341.2013.05.03
2013⁃08⁃14;接受日期:2013⁃09⁃10
国家自然科学基金项目(C1306)资助。
刘亚运,硕士研究生,主要从事植物分子生物学与基因工程研究。E⁃mail:liuyayunxx@163.com。∗通信作者:路铁刚,研究员,主要从事植物分子生物学与基因工程研究。E⁃mail:lutiegang@caas.cn
