长江屏山江段犁头鳅种群的遗传结构
2013-04-19飞柳明吴清江谢松光
程 飞柳 明吴清江谢松光
长江屏山江段犁头鳅种群的遗传结构
程 飞1,2柳 明1吴清江1谢松光1,3
(1. 中国科学院水生生物研究所, 中国科学院水生生物多样性与保护重点实验室, 武汉 430072; 2. 中国科学院大学, 北京 100049; 3. 中国科学院水生生物研究所淮安研究中心, 淮安223002)
长江上游是我国淡水鱼类资源最为丰富的地区之一,已记录鱼类261种; 同时, 该江段也是我国特有鱼类最为集中的江段, 分布特有鱼类112种[1]。这一江段水流湍急,滩潭交错, 流态复杂多样。很多鱼类在生理、形态和生活史特征等方面表现出与该江段特殊的栖息地环境相适应的特化[2]。目前长江上游金沙江江段在建的水电站至少有两座(向家坝、洛溪渡), 规划建设的还有十余座[3]。这些水电站的建设和运行将极大地改变上游水文环境, 对上游鱼类(特别是特有鱼类)产生显著影响。
犁头鳅Lepturichthys fimbriata (Günther) 隶属鲤形目(Cypriniformes)爬鳅科(Balitoridae)平鳍鳅亚科(Homalopterinae) 犁头鳅属 (Lepturichthys), 是长江特有鱼类[4]。犁头鳅是生活于急流石滩的底栖小型鱼类, 利用宽大平展的偶鳍和平坦裸露的胸腹部适应急流生境[5]。金沙江犁头鳅的生殖季节为4月中旬至6月中旬, 在峡谷江段产漂流性卵[6]。犁头鳅是一种非渔业对象的小型特有鱼类, 是监测和研究环境变化对鱼类影响的理想材料之一。但迄今为止, 犁头鳅的相关遗传结构研究还是空白。本文利用线粒体DNA(mtDNA)的细胞色素c 氧化酶亚基Ⅰ(COⅠ)基因的部分序列, 研究了金沙江下游屏山江段犁头鳅的遗传结构, 为评估和监测金沙江水电梯级开发对犁头鳅种群遗传结构的影响提供本底资料。
1 材料与方法
1.1 样品采集
2008年5月至7月于长江屏山江段(28o60´N, 104o10´ E)采集漂浮性仔鱼样品(图 1)。采样工具为自制的定置网,类似于长江中下游传统捕苗工具弶网; 网目40目, 网口呈长方形, 宽2.0 m, 深1.5 m, 全长2.5 m。网具24h固定于江边缓流区, 浮于水面, 每天上午(10:00)、下午(5:00)各取样一次。同时, 在屏山江段渔获物中收集犁头鳅成鱼样品。所有样品均在95%的酒精中保存, 置于4℃冰箱中。
1.2 犁头鳅仔鱼的鉴定
测定个体mtDNA COⅠ基因5′端的部分序列, 计算Kimura双参数遗传距离(K2P Distance), 分析序列间的分化[7]; 基于鱼类barcoding研究结果[8], 将与犁头鳅成鱼K2P距离小于2%的仔鱼个体确定为犁头鳅仔鱼。
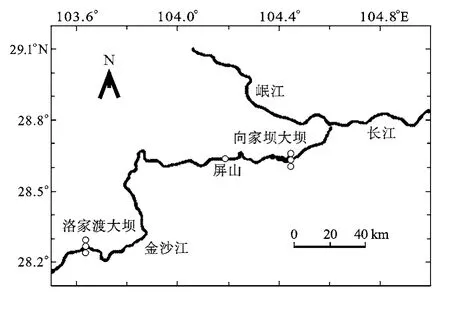
图 1 长江上游屏江江段犁头鳅种群样品采样位置()和两座在建的大坝 ()Fig. 1 Sampling location () for Lepturichthys fimbriata population in the Pingshan section and the locations of the two dams under construction () in the upper Yangtze River
1.3 DNA提取与扩增
随机选取1000尾仔鱼 (3.3—18.8 mm TL) 和12尾犁头鳅成鱼 (67—108 mm TL)。利用高盐法提取仔鱼个体和成鱼鳍条的总DNA, 琼脂糖电泳检测DNA质量和浓度。PCR扩增各样品的mtDNA COⅠ基因; 扩增引物采用FishF1 & FishR1; 引物序列、反应体系和程序参见Ward, et al.[8]。扩增使用Bio-Rad PCR仪(Bio-Rad, PTC-100)。PCR产物经琼脂糖电泳检测后, 纯化目标片段, 再利用扩增引物测序。测序由北京华大基因研究中心完成。
1.4 数据分析
利用软件Clustal X v 1.83[9]对序列数据进行全序列比对, 剪除两端不可靠碱基, 并加以人工校正。将碱基序列翻译成氨基酸, 以检验测序错误和假基因(Pseudogenes)。利用软件MEGA 4.0[10]计算Kimura K2P 距离, 分析序列间的分化, 并统计序列的碱基组成。
利用软件DnaSP v 5.1[11]检测犁头鳅仔鱼与成鱼的群体分化水平, 如果无显著性分化, 则作为一个群体分析;若存在显著分化, 则分为两个群体分析。同时利用该软件计算遗传多样性指数, 包括单倍型数目(Haplotype number, k)、单倍型多样性(Haplotypic diversity, h)、平均核苷酸差异数(Average pairwise sequence differences, П)和核苷酸多样性(Nucleotide diversity, π)。
利用软件STRUCTURE v 2.3[12]检验犁头鳅样品遗传分化群。从1到10分别运算遗传分化群(K)的可能性, 根据K的后概率值判定样品的遗传分化[12,13]。检验基于Bayesian判别的混合模型, 马尔科夫链的设置为5 × 105迭代, 5次重复。
如果犁头鳅样品存在遗传分化, 则将每一个遗传分化群视作一个群体。利用软件DnaSP v 5.1计算各群体的遗传多样性指数k、h、П和π; 并采用Tajima以及Fu & Li的检验方法[11], 检测群体DNA水平的自然选择作用。利用软件ARLEQUIN v 3.1[14]统计各种群间的遗传分化水平, 并采用分子变异梯度分析(Hierarchical analysis of molecular variance, AMOVA)检测遗传变异来源; 分析设置1 × 104次置换检验。
利用软件TCS 1.21[15]分析犁头鳅各单倍型的进化网络, 置信区间为95%。
2 结果
共成功扩增824尾仔鱼和12尾犁头鳅成鱼序列, 扩增长度在750 bp左右。对序列进行比对、分析和人工校正后, 长584 bp的碱基用于后续分析。584 bp序列中没有插入和缺失, 也不包含移框突变和终止密码子。根据2%K2P距离的标准, 共有57尾仔鱼被鉴定为犁头鳅, 仔鱼平均K2P距离是0.8%。57尾犁头鳅仔鱼最早出现时间是5月9号, 最晚时间是7月4号; 在6月1号出现一个明显的苗汛高峰。
在仔鱼和成鱼共69尾犁头鳅序列中, 检测到一尾仔鱼存在一个氨基酸替换(val → lys), 即84号密码子第一位置的碱基G转换成A, 其余个体均没有氨基酸变化。碱基组成T、C、A、G平均含量分别是27.1%、28.5%、26.2%、18.2%, A + T含量明显高于C + G含量。
卡方检验表明, 仔鱼与成鱼没有显著的群体分化(P = 0.776, df = 21), 因此, 合并为一个群体分析。584 bp序列含有33个(占全系列5.7%)多态位点, 其中21个(占3.6%)是简约性信息位点。遗传多样性指数k、h、П和π分别是22 (Hap 1—22, GenBank No:JN830334 – JN830355)、0.882、4.076和0.007。
Bayesian判别检验表明, ln P(D)在K = 3出现最大值(图 2A), 69尾犁头鳅可以分为3个(K = 3)遗传分化群(图2B), 记为sub1、sub2和sub3, 分别包含27、33和9尾个体。3个群体间遗传分化显著, 尤其是sub3与另两个群体的分化极为显著, 其配对分化分别是0.842、0.891(P < 0.001,表 1)。AMOVA分析表明, 群体间的变异占整个遗传变异的73.3%, 而来源于各群体内的变异占26.7% (表 2)。
22种单倍型在三个群体内分布极不平均, 只有一种单倍型(Hap 2)为sub1和sub2共享, 其余单倍型分别为不同群体独有。在3个群体中sub2的各多样性指数均表现为较低或最低(表3)。
Tajima 检验和Fu & Li 检验的P值均大于0.1, 表明犁头鳅各群体内mtDNA进化遵循中性模型。
TCS分析显示22种单倍型分为两个进化枝, 一个进化枝是群体sub3, 包括Hap15—19, 共5种单倍型, sub3的单倍型沿单一进化方向经历了多次突变, 明显与其他群体分离; 另一个进化枝包括其余17种单倍型, 分布在sub1和sub2之中(图3)。单倍型Hap2是犁头鳅最常见和原始单倍型, 也是唯一共享单倍型。
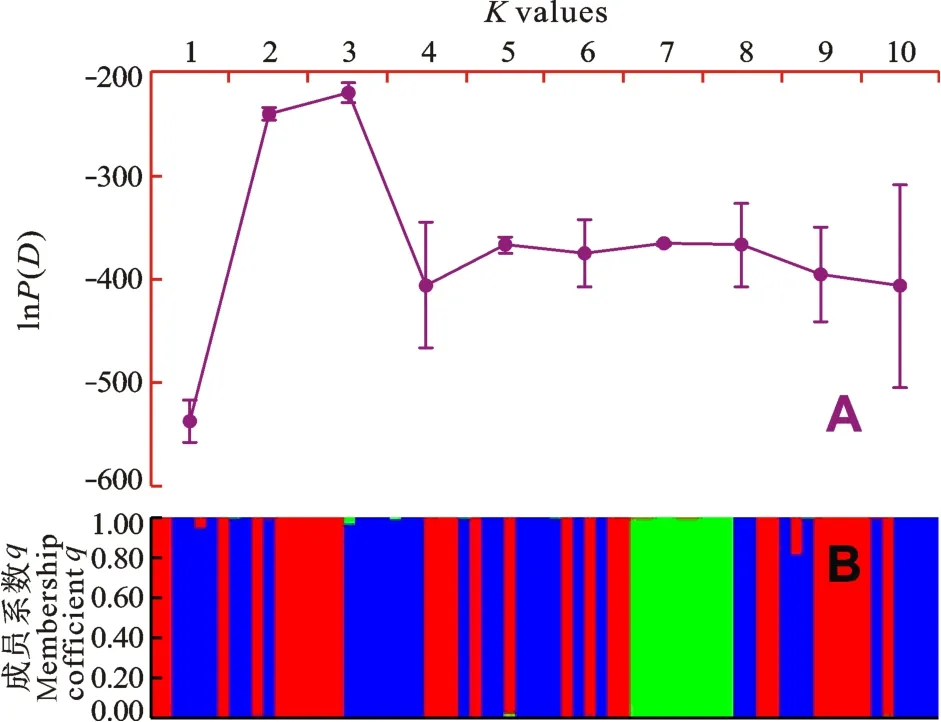
图 2 利用软件Structure v 2.3的混合模型分析长江上游屏江江段犁头鳅种群的遗传分化群Fig. 2 Varied curve of [lnP(D)] and inferred genetic clusters of the Bayesian clustering analysis of Lepturichthys fimbriata population in the Pingshan section of the upper Yangtze River performed with STRUCTURE 2.3
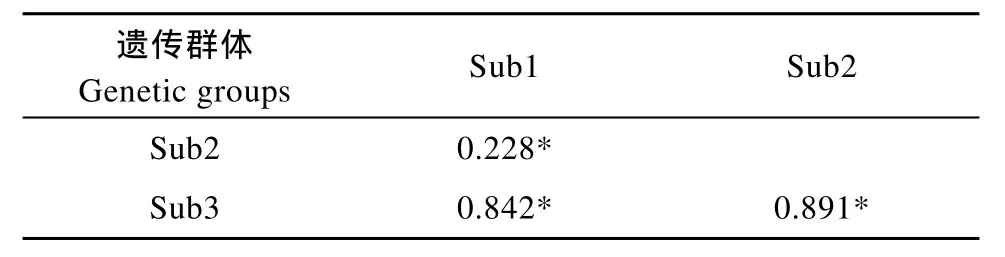
表1 长江上游屏山江段犁头鳅种群3个遗传分化群体间的配对分化系数(Fst)Tab. 1 Pairwise Fstvalues among the three genetic groups (Sub 1-3) of Lepturichthys fimbriata in the Pingshan section of the upper Yangtze River
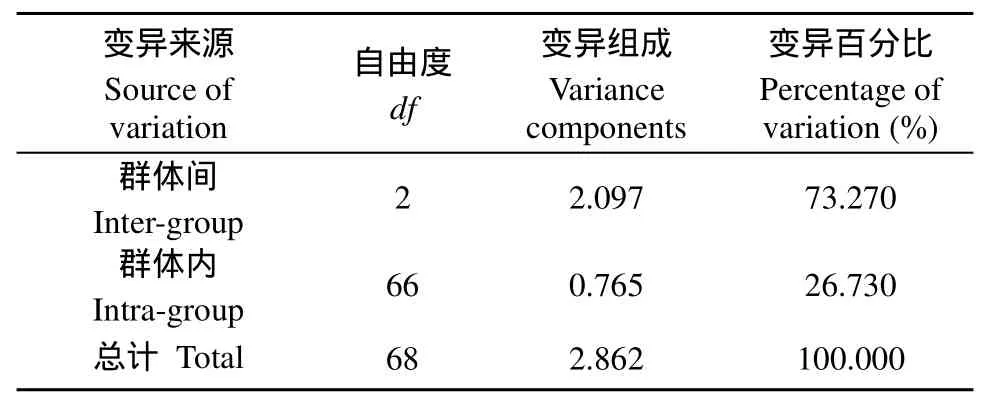
表2 长江上游屏江江段犁头鳅种群3个遗传分化群体分子变异梯度分析Tab. 2 Hierarchical analysis of molecular variance for the three genetic groups of Lepturichthys fimbriata in the Pingshan section of the upper Yangtze River
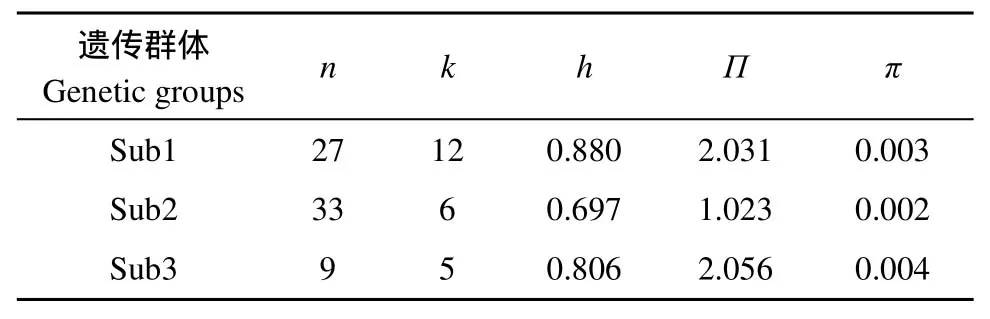
表3 长江上游屏江江段犁头鳅种群3个不同遗传群体的遗传多样性指数Tab. 3 Genetic diversity indices of the three genetic groups of Lepturichthys fimbriata in the Pingshan section of the upper Yangtze River
3 讨论
对犁头鳅种群分化的研究结果表明, 在采集的样品中存在遗传分化显著的3个群体(表1)。共享单倍型数目及Tajima 检验和Fu & Li 检验的结果均显示群体间缺乏基因流。这些结果说明研究江段犁头鳅种群存在具有一定遗传隔离的不同群体。鱼类的这种多遗传群体结构现象可能较为普遍[如鲱鱼 (Clupea harengus)[16]、红大马哈鱼(Oncorhynchus nerka)[17]]。在对具有多个遗传隔离群体的鱼类进行管理和保护时, 如果将其视为单一的管理单位 (Management unit, MU), 极有可能导致特定群体的消失[17,18]。加拿大Newfoundland & Labrador海域的鳕鱼(Gadus morhua)由若干遗传分化的群体组成, 由于对其多群体结构认识不足, 之前一直作为单一管理单位进行管理, 过度捕捞导致多个补充能力弱的亚群体消失或严重受损, 导致加拿大鳕鱼渔业在1992年几近崩溃, 其后虽经十多年的努力, 资源恢复缓慢[19]。考虑到长江上游丰富的特有鱼类和生境多样性, 与犁头鳅相似包含具有一定遗传隔离的多群体鱼类种类可能较为普遍。
遗传多样性指数π是度量mtDNA变异程度的主要指标之一, 其值的大小表示群体多态程度的高低[20]。尽管全部犁头鳅个体的π值(0.007)显示该物种的遗传多样性较高, 但3个分化群体的π值介于0.002—0.004之间(表 3),各群体内的遗传多样性并不高, 明显小于一级保护鱼类中华鲟Acipenser sinensis 的遗传多样性(π值: 0.008—0.016)[21]和二级保护鱼类胭脂鱼Myxocyprinus asiaticus的遗传多样性(π值: 0.008—0.052)[22]。由于群体间的显著分化, 基因流缺乏, 对犁头鳅种群的遗传多样性及种群对环境变化的潜在适应能力评价应该以各群体内(即MU)遗传多样性的大小为主。
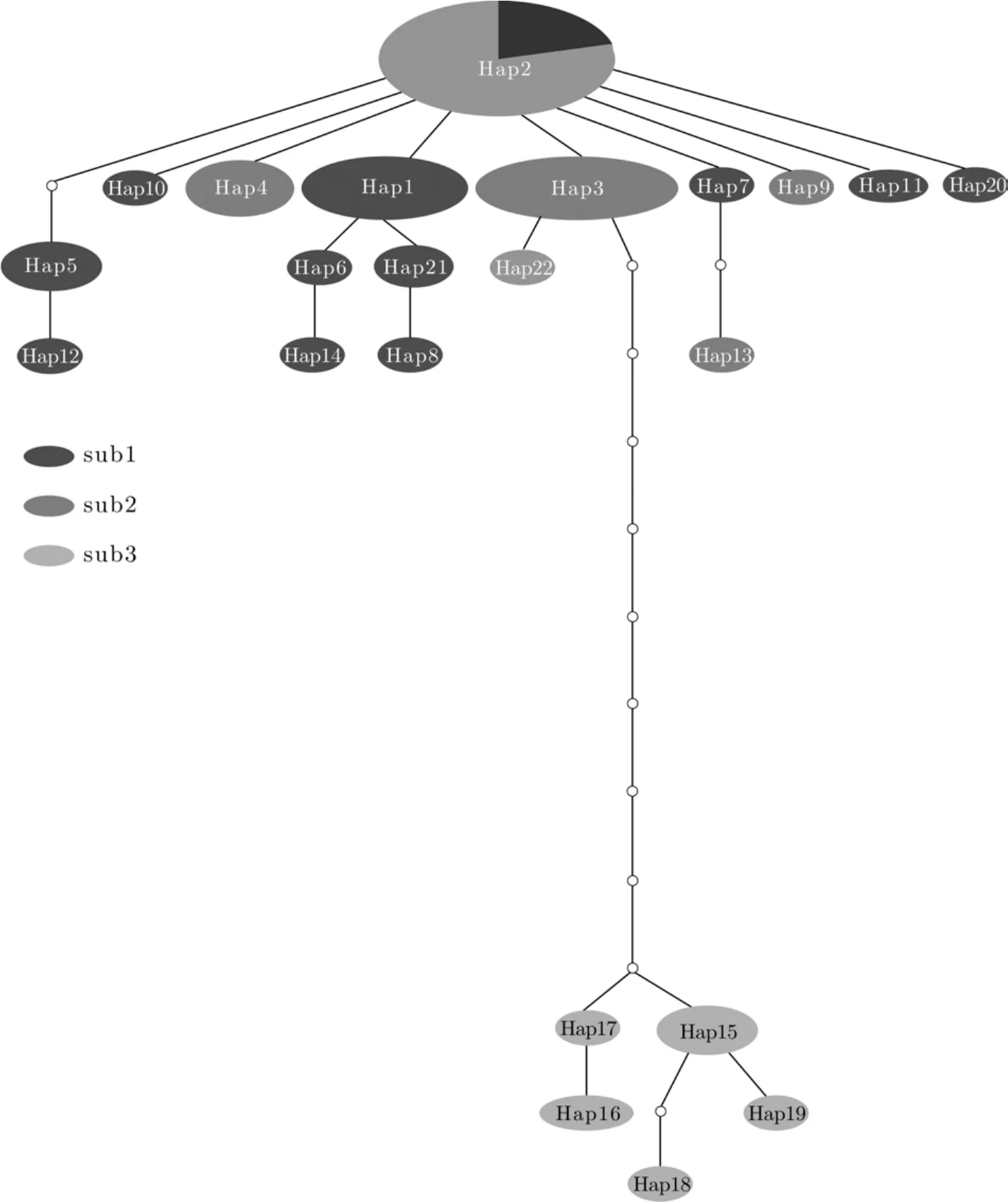
图 3 长江上游屏江江段犁头鳅种群单倍型的简约进化网络图Fig. 3 Statistical parsimony network based on haplotype frequencies of Lepturichthys fimbriata population in the Pingshan section of the upper Yangtze River
大坝的建设和运行是淡水鱼类生物多样性主要威胁之一[23]。梯级水电站的建设和运行将极大地改变长江上游鱼类的栖息环境, 是长江上游鱼类资源(特别是特有鱼类)的重要威胁。本研究表明, 长江上游犁头鳅种群包含具有一定遗传隔离, 遗传分化明显的多个群体, 且各群体内的遗传多样性较低。对于这种由多个群体构成的鱼类种群, 梯级大坝建设有可能导致各群体遗传多样性的下降, 甚至个别群体灭绝, 并进一步加快群体分化过程。有关这类鱼类种群的遗传结构及各群体的生物学特征研究,对评价大坝建设对这类种群的影响, 进而制定科学的保护和管理措施具有十分重要的意义。
[1] Cao W X, Yu Z T, Xu Y X, et al. The Preliminary Evaluation of Impact of the Three Gorges Project and the Proliferation Methods of Fish Resource [M]. Beijing: Science Press. 1987, 2—20 [曹文宣, 余志堂, 许蕴玕, 等. 三峡工程对长江鱼类资源影响的初步评价及资源增殖途径的研究. 北京: 科学出版社. 1987, 2—20]
[2] Wang Y M. The protection measures of fishes in the upper section of Yangtze River [D]. Thesis for Doctor of Science. Institute of Hydrobiology, Chinese Academy of Sciences, Wuhan. 2004 [王亚民. 长江上游鱼类资源保护对策. 博士学位论文, 中国科学院水生生物研究所, 武汉. 2004]
[3] Yang G S, Weng L D, Li L F. Yangtze Conservation and Development Report 2007 [M]. Beijing: Science Press. 2008,205—213
[4] Chen Y Y, Tang W Q. Homalopterinae [A]. In: Fauna Sinica Osteichthyes Scypriniformes 2 [C]. Beijing: Science Press. 2000, 520—563 [陈宜瑜, 唐文乔. 平鳍鳅亚科. 见: 中国动物志硬骨鱼纲鲤形目(下卷). 北京: 科学出版社. 2000, 520—563]
[5] Xiong Y Y, Qiao Y, Liu H Z, et al. Early development of Lepturichthys fimbriata (Günther) (Cypriniformes: Balitoridae) [J]. Acta Hydrobiologica Sinica, 2008, 32(3): 424—433 [熊玉宇, 乔晔, 刘焕章, 等. 犁头鳅早期发育. 水生生物学, 2008, 32(3): 424—433]
[6] Ding R H. The Fishes of Sichuan [M]. Chengdu: Sichuan Publishing House of Science and Technology. 1994, 429—432 [丁瑞华. 四川鱼类志. 成都: 四川科学技术出版社. 1994, 429—432]
[7] Kimura M. A simple method for estimating evolutionary rates of base substitutions through comparative studies of nucleotide sequences [J]. Journal of Molecular Evolution, 1980, 16(2): 111—120
[8] Ward R D, Zemlak T S, Bronwyn H, et al. DNA barcoding Australia’s fish species [J]. Philosophical Transactions of the Royal Society B-Biological Sciences, 2005, 360(1462): 1847—1857
[9] Thompson J D, Gibson T J, Plewniak F, et al. The Clustal X windows interface: Flexible strategies for multiple sequences alignment aided by analysis tools [J]. Nucleic Acids Research, 1997, 25(24): 4876—4882
[10] Tamura K, Dudley J, Nei M, et al. MEGA4: Molecular Evolutionary Genetics Analysis (MEGA) software version 4.0 [J]. Molecular Biology and Evolution, 2007, 24(8): 1596—1599
[11] Librado P, Rozas J. DnaSP v.5: a software for comprehensive analysis of DNA polymorphism data [J]. Bioinformatics, 2009, 25(11): 1451—1452
[12] Pritchard J K, Stephens M, Donnelly P. Inference of population structure using multilocus genotype data [J]. Genetics, 2000, 155(2): 945—959
[13] Cheng F, Wu Q J, Liu M, et al. Impacts of hatchery release on genetic structure of rock carp Procypris rabaudi in the upper Yangtze River, China [J]. Fisheries Science, 2011, 77(5): 765—771
[14] Excoffier L, Laval G., Schneider S. Arlequin ver. 3.0: An integrated software package for population genetics data analysis [J]. Evolutionary Bioinformatics Online, 2005, 1(1): 47—50
[15] Clement M, Posada D, Crandall K A. TCS: a computer program to estimate gene genealogies [J]. Molecular Ecology, 2000, 9(10): 1657—1659
[16] Jørgensen H B H, Hansen M M, Bekkevold D, et al. Marine landscapes and population genetic structure of herring (Clupea harengus L.) in the Baltic Sea [J]. Molecular Ecology, 2005, 14(10): 3219—3234
[17] Palsbøll P J, Be´rube´ M, Allendorf F W. Identification of management units using population genetic data [J]. Trends in Ecology and Evolution, 2006, 22(1): 10—16
[18] Reiss H, Hoarau G, Dickey-Collas M, et al. Genetic population structure of marine fish: mismatch between biological and fisheries management units [J]. Fish and Fisheries, 2009, 10(4): 361—395
[19] Sterner, T. Unobserved diversity, depletion and irreversibility– the importance of subpopulations for management of cod stocks [J]. Ecological Economics, 2007, 61(2—3): 566—574 [20] Aboim M A, Menezes G M, Schlitt T, et al. Genetic structure and history of populations of the deep-sea fish Helicolenus dactylopterus (Delaroche, 1809) inferred from mtDNA sequence analysis [J]. Molecular Ecology, 2005, 14(5): 1343—1354
[21] Zhang S M, Wang D Q, Zhang Y P. Mitochondrial DNA variation, effective female population size and population history of the endangered Chinese sturgeon, Acipenser sinensis [J]. Conservation Genetics, 2003, 4(6): 673—683
[22] Sun Y H. Genetic diversity of Chinese sucker (Myxocyprinus asiaticus) and molecular systematics of Catostomidae fishes [D]. Thesis for Doctor of Science. Wuhan University, Wuhan. 2004 [孙玉华. 中国胭脂鱼遗传多样性及亚口鱼科分子系统学研究. 博士学位论文, 武汉大学, 武汉. 2004]
[23] Coulon A, Fitzpatrick J W, Bowman R, et al. Effects of habitat fragmentation on effective dispersal of Florida Scrub-Jays [J]. Conservation Biology, 2010, 24(4): 1080—1088
GENETIC STRUCTURES OF LEPTURICHTHYS FIMBRIATA POPULATION IN THE PINGSHAN SECTION OF THE YANGTZE RIVER
CHENG Fei1,2, LIU Ming1, WU Qing-Jiang1and XIE Song-Guang1,3
(1.State Key Laboratory of Aquatic Biodiversity and Conservation of Chinese Academy of Sciences, Institute of Hydrobiology, Chinese Academy of Sciences, Wuhan 430072, China; 2. University of Chinese Academy of Sciences, Beijing 100049, China; 3. Huai’an Research Center, Institute of Hydrobiology, Chinese Academy of Sciences, Huai’an 223002, China)
犁头鳅; COⅠ; 遗传分化; 遗传多样性; 长江上游
Lepturichthys fimbriata; COⅠ; Genetic differentiation; Genetic diversity; The upper Yangtze River
Q346
A
1000-3207(2013)01-0145-05
10.7541/2013.145
2011-10-18;
2012-10-16
国家重点基础研究发展计划(973计划)项目(2010CB429000); 大自然保护协会(TNC)项目; 淡水生态与生物技术国家重点实验室开放课题(Y15C01); 中国科学院“百人计划”择优支持项目; 湖北省博士后科技活动择优资助; 国家自然科学基金(No. 51209202); 中国博士后科学基金(20100480924)资助
程飞(1977—), 男, 湖北黄陂人; 博士研究生; 主要从事鱼类生态学研究。E-mail: chengfei@ihb.ac.cn
谢松光, 研究员, 博士生导师; E-mail: xiesg@ihb.ac.cn
