获得构建miRNA高表达载体的pre-miRNA及其侧翼DNA序列的生物信息学方法
2013-03-16李嘉熙宋刘梅王美晨李冬民
李嘉熙,宋刘梅,王美晨,蓝 茜,李 玥,刘 莉,王 璇,韩 燕,李冬民
西安交通大学医学部:1生物化学与分子生物学系;2临床医学七年制,西安 710061
MicroRNA(miRNA)是一类长约20-24个核苷酸、具有重要调控功能的内源性非编码小RNA。miRNA基因通常在RNA聚合酶II作用下从基因组转录出长约300~1 000个碱基的初级miRNA(即primary mi-RNA、简写为pri-miRNA),经核内Drosha酶复合体剪接加工生成长约70~90个碱基、含有茎环和发夹结构的miRNA前体(即precursor miRNA、简写为premiRNA),然后pre-miRNA被运送到胞质再经Dicer酶酶切后生成长约20~24个核苷酸的miRNA:mi-RNA*双链;这种双链很快被组装进入RNA诱导沉默复合体(RNA induced silence complex,RISC)中,其中一条成熟的单链miRNA通过碱基互补配对的方式识别靶mRNA,并根据互补程度的不同指导RISC降解靶mRNA或阻遏靶mRNA的翻译[1-3]。最近研究表明,miRNA在细胞增殖和凋亡、发育、造血、脂肪代谢、器官形成、病毒防御等众多生命活动过程中具有非常重要的调节作用[4-7],因此,对于其功能的阐明成为各领域研究的热点。
目前,miRNA表达载体是miRNA功能研究使用最多、最广泛的手段之一;实际研究中,pre-miRNA应用最早,也最广泛,目前很多商业化的MicroRNA库都是pre-miRNA形式的。近几年来,研究发现premiRNA发夹结构两侧的序列(即microRNA的双臂)对成熟miRNA的形成有着十分重要的作用[8],premiRNA+侧翼序列的表达载体形式越来越多地被研究者采用,而获得miRNA前体及其侧翼的DNA序列是miRNA表达载体构建技术中至关重要的一环。尽管目前已有很多miRNA的数据库[9],但尚未有文献详细报道如何获得pre-miRNA及其侧翼的DNA序列。因此,该文将详细介绍如何联合利用miRBase和Ensembl数据库获得某个特定miRNA的前体(premiRNA)及其侧翼的DNA序列。
1 miRBase和Ensembl序列数据库简介
1.1 miRBase序列数据库介绍
miRBase序列数据库是曼彻斯特大学小RNA数据库,是一个提供包括miRNA序列数据、注释、预测基因靶标等信息的全方位数据库,是存储miRNA信息最主要的公共数据库之一,其网址是:http://www.mirbase.org[10]。它主要由三部分组成:miRNADatabase、miRBase Registry和miRBase Targets database。miRBase database也就是miRBase Sequence Database,主要提供的是已公布的miRNA序列(包括预测的pre-miRNA,成熟miRNA序列(称为miR)的位置和序列信息)和注释的搜索服务,发夹和成熟序列都可以通过mi-RBase里的searching和browsing找到,也可通过名称、关键词、参考文献和注释进行检索。miRBase Registry主要为miRNA提供注册服务,即为研究者们在研究结果发表之前给每个新发现的miRNA基因一个惟一的名称。而miRBaseTargets data-base(即miRNA靶标预测)目前已更名为microCosm并托管于EBI(european bioinformatics institute),主要针对动物miRNA靶基因的查询[10-11]。
1.2 Ensembl数据库简介
Ensembl数据库是三大人类基因组数据库(NCBI、USUC和Ensembl)之一,是欧洲生物信息学研究所(the European Bioinformatics Institute,EBI)与英国 Wellcome基金会的Sanger研究所的合作项目,其网址是:http://www.ensembl.org[12,13]。Ensembl数据库内含有不断更新的序列数据及真核细胞基因组自动产生的注释数据,现已有包括人类、小鼠、大鼠、河豚、蚊子与蜥蜴在内的40多个物种,其中心理念是将基因和其他丰富的注释数据在参考基因组上以自动可视化的方法展现。Ensembl提供的丰富功能包括:①方便取得序列数据;②已知基因的预测结构以及其在基因组上的定位;③预测的基因和有关的支持证据;④基因组生物特性的注释数据;⑤与国际上其他基因组资源的连接[12-13]。
2 利用miRBase获得pre-miRNA及其侧翼序列的详细步骤
首先打开miRBase序列数据库的主页,在主页右上方的搜索框内输入欲查询miRNA的注册号、名称或关键词,也可输入参考文献和注释,然后点击“Search”进行检索;也可通过在“search by miRNA name or keywords”搜索框内输入miRNA名称和关键词,然后点击“Go”来查询。如果输入的查询信息模糊、不精确,搜索结果则包括的范围广,需要浏览这些结果找到欲查询的miRNA;若输入的查询信息精确(如输入规范的miRNA名称),则可直接搜索到欲查询的miRNA(如图1所示)。下面我们以人miRNA 21(hsamir-21)为例来详细介绍如何利用miRBase获得hsamir-21的pre-miRNA及其侧翼的DNA序列。

图1 miRBase数据库中检索miRNA
2.1 利用miRBase数据库获得pre-miRNA在Ensemble数据库中的位置链接

图2 hsa-mir-21在miRBase数据库中的检索结果
在“search by miRNA name or keywords”搜索框内输入hsa-mir-21,点击“Go”或按回车键(如图1所示),则进入查询结果页面(图2),主要包括了4部分结果:“Stem-loop sequence hsa-mir-21”(即hsa-mir-21茎环/发卡序列)、“Mature sequence hsa-miR-21-5p”(即hsa-miR-21-5p成熟序列)、“Mature sequence hsa-miR-21-3p”(即 hsa-miR-21-3p 成熟序列)以及“References”(参考文献)。在“Stem-loop sequence hsa-mir-21”部分除了包括注册号、符号、描述、注释、基因家族、茎环及其序列等信息外,还包括有hsa-mir-21茎环/发卡(即hsa-mir-21的pre-miRNA)链接到Ensemble数据库的位置信息,即位于人17号染色体:57,916,627-57,920,698,如图2 椭圆内所示。
2.2 在Ensemble数据库获得pre-miRNA在基因组上的位置及碱基序列
点击hsa-mir-21在基因组上的位置链接:chr17:57918627-57918698[+],即可进入 Ensembl数据库检索结果页面,最上端显示的是17号染色体的整体结构情况。在中间位置的“Region in detail”部分则更详细地显示 Chromosome 17:57,916,627-57,920,698区域内的染色体带及基因叠连群,其中矩形框标示的是包含hsa-mir-21茎环结构所在的位置(如图3所示)。用鼠标左键单击“MIR21”,则可弹出一个注释MIR21特征的小窗口“HGNC Symbol:MIR21”;该窗口记录有基因号(“gene”)、位置(“location”)、基因类型(gene type)、所在链(strand)、分析(analysis)和预测方法(prediction method)(如图3所示)。用鼠标左键单击“location”后的超链接 Chromosome 17:57,918,627-57,918,698,则可详细显示其位置、碱基序列以及序列导出窗口按钮等(如图4所示)。
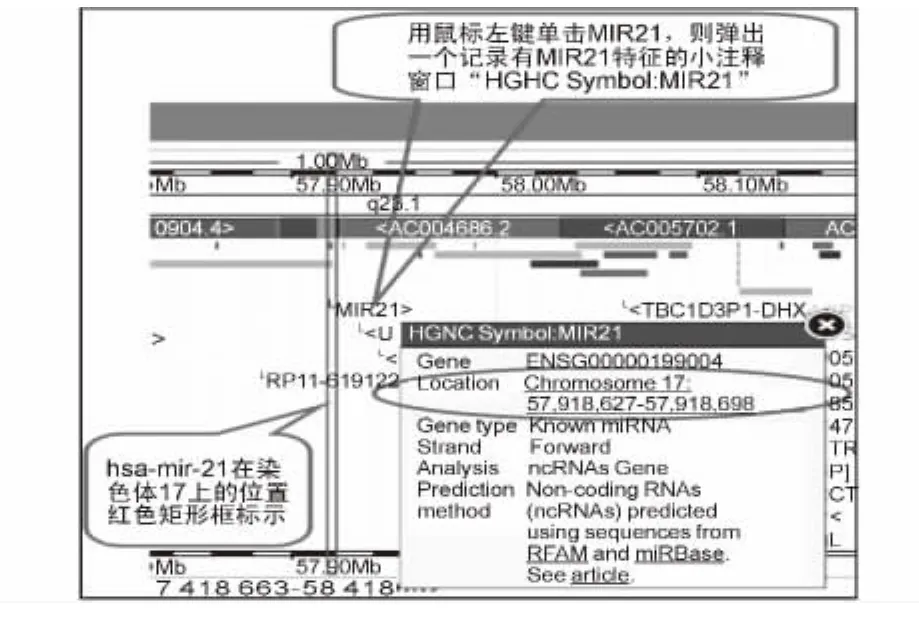
图3 利用Ensemble数据库获得hsa-mir-21的注释数据和其pre-miRNA在基因组上的位置链接
2.3 利用Ensemble数据库获得pre-miRNA及其侧翼的DNA序列
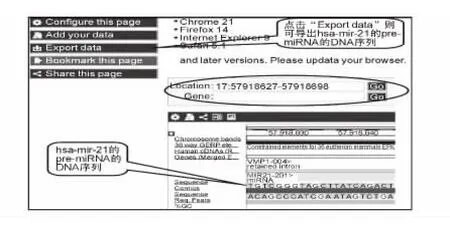
图4 利用Ensemble数据库获得hsa-mir-21的pre-miRNA的DNA序列及序列导出窗口按钮

图5 Ensemble数据库中hsa-mir-21 pre-miRNA及其侧翼序列导出窗口
点击图4左上方的“Export data”则可弹出“Export data”-设置窗口(如图5所示):包括位置、序列格式、选择的位置,最重要的是我们可以根据所需5'和3'侧翼序列的长度、在5'和3'侧翼序列输入框内填写相应的数字,设置的长度范围为:0-1000000。如果在5'和3'侧翼序列的长度输入框内填写“0”,则导出的序列只是hsa-mir-21的pre-miRNA基因序列;如果在5'和3'侧翼序列的长度输入框内填写大于0小于1000000的数字,则导出的序列不但包含hsa-mir-21的pre-miRNA基因序列,还包括其相应长度的5'和3'侧翼的DNA序列。目前在构建miRNA高表达载体时,会在其上下游各多扩增100-200bp的序列,以保证pre-miRNA剪切后能生成成熟miRNA[8]。例如:在hsa-mir-21的pre-miRNA“Export data”窗口的5'及3'侧翼序列输入框内各填写数字150,然后点击“next”,弹出“Export data-Export Con-figuration-Output format”窗口,在此窗口内列出了三种序列格式(HTML、Text和Compressed text),根据自己的需要,单击相应序列格式的超链接,则可导出hsa-mir-21相应序列格式的pre-miRNA及其5'和3'侧翼长各为150碱基的DNA序列。
综上所述,我们介绍了利用miRBase和Ensembl数据库获得pre-miRNA及其侧翼DNA序列的详细步骤,这对miRNA高表达载体的成功构建及其功能研究具有非常重要的意义。
[1]Winter J,Jung S,Keller S,et al.Many roads to maturity:microRNA biogenesis pathways and their regulation[J].Nat Cell Biol,2009,11(3):228-34
[2]Chen K,Rajewsky N.The evolution of gene regulation by transcription factors and microRNAs[J].Nat Rev Genet,2007,8(2):93-103
[3]Finnegan E F,Pasquinelli A E.MicroRNA biogenesis:regulating the regulators[J].Crit Rev Biochem Mol Biol,2013,48(1):51-68
[4]Raza A,Galili N.The genetic basis of phenotypic heterogeneity in myelodysplastic syndromes[J].Nat Rev Cancer,2012,12(12):849-859
[5]Carroll AP,Tooney PA,Cairns MJ.Context-specific micro RNA function in developmental complexity[J].J Mol Cell Biol,2013,5(2):73-84
[6]Vickers KC,Sethupathy P,Baran-Gale J,et al.Complexity of microRNA function and the role of isomiRs in lipid homeostasis[J].J Lipid Res,54(5):1182-1191
[7]Bartel D P.MicroRNAs:target recognition and regulatory functions[J].Cell,2009,136(2):215-233
[8]Park JE,Heo I,Tian Y,Simanshu DK,et al.Dicer recognizes the 5'end of RNA for efficient and accurate processing[J].Nature,2011,475(7355):201-205
[9]Vlachos IS,Hatzigeorgiou AG.Online resources for miRNA analysis[J].Clin Biochem,2013,46(10-11):879-900
[10]Griffiths-Jones S,Saini HK,van Dongen S,et al.miRBase:tools for microRNA genomics[J].Nucleic Acids Res,2008,36(Database issue):154-158
[11]高青,鞠志花,王长法,等.miRBase-microRNA序列数据库[J].家畜生态学报,2011,(32):101-104
[12]Flicek P,Ahmed I,Amode MR,et al.Ensembl[J].Nucleic Acids Res,2013,41(Database issue):48-55
[13]Fernandez-Suarez X M,Schuster M K.Using the ensembl genome server to browse genomic sequence data[J].Curr Protoc Bioinformatics Chapter,2010,Chapter 1:Unit1.15.doi:10.1002/0471250953.bi0115s30
