马铃薯StSnRK2.1基因克隆与生物信息学分析
2013-01-16刘思妍毛娟范阿棋张俊莲王蒂白江平
刘思妍 毛娟 范阿棋 张俊莲 王蒂 白江平
(甘肃省作物遗传改良与种质创新重点实验室 甘肃省干旱生境作物学重点实验室 甘肃农业大学农学院,兰州 730070)
蔗糖非发酵相关蛋白激酶家族(SnRKs)在植物许多生理过程中起着重要的作用,根据基因序列的相似性和基因结构的不同将其分为3个亚家族,分 别 是 SnRK1,SnRK2 和 SnRK3[1-3]。SnRK2 亚 家族是植物中所特有的一类,参与植物逆境胁迫,对植物抵御不良环境具有一定的调节作用,已在拟南芥、水稻、玉米中克隆并鉴定了其功能[4,5]。
马铃薯作为粮菜兼用型作物,在作物生产中占有极其重要的作用,尤其对我国西部干旱地区。但是马铃薯对干旱、高温等非生物性胁迫的响应遗传机理与种质的改良还都处于初始阶段[6-8],其原因一方面是植物响应逆境胁迫的数量遗传背景以及生理机理的复杂性使得抗旱育种的进展非常缓慢[9,10],另一方面是因为马铃薯多倍体遗传背景的复杂性限制了对马铃薯功能基因组的研究和生物技术在马铃薯种质改良中的应用[11-17]。SnRK2家族基因被认为与植物响应逆境胁迫有紧密联系[18,19],甘肃主栽品种陇薯3号与其他品种相比具有明显的抗旱性。本研究从陇薯3号中分离SnRK2基因家族并对其功能进行分析预测,拟使其对改善植物抵抗逆境胁迫研究具有积极地推动作用。
1 材料与方法
1.1 材料
马铃薯试管苗(陇薯3号)由甘肃省遗传改良与种质创新重点实验室保存。
E. coliDH5α,本实验室保存;载体pGEMTeasy,购自Promega公司;RNA提取试剂盒、琼脂糖凝胶DNA回收试剂盒,购自TIANGEN公司;M-MuLV第一链cDNA合成试剂盒、DreamTaqTMGreen PCR Master Mix(2X)及引物设计由上海生工生物工程有限公司承担。
1.2 方法
1.2.1 总RNA的提取和cDNA第一链的合成 按照TIANGEN公司的RNA提取试剂盒操作说明书提供的方法提取马铃薯试管苗总RNA。参照上海生工的反转录试剂盒的操作说明得到反转录产物并保存在-20℃下备用。
1.2.2 RT-PCR法扩增StSnRK2.1基因 根据SnRK2同源序列设计引物:上游引物为:5'-GCTCTAGAATGGAGCGTTATGAGATAGTGAAG-3'(下划线部分为XbaⅠ酶切位点);下游引物为:5'-GCGAGCTCTC ACAGTAAACCAGCAAAATCAG-3'(下划线部分为SacⅠ酶切位点)。扩增条件为:95℃ 1 min,95℃ 30 s,55℃ 1 min,72℃ 1 min,40个循环,72℃ 10 min。PCR产物经胶回收试剂盒纯化后保存在-20℃下备用。
1.2.3 载体pGEM-Teasy的构建 将纯化后的PCR产物与T-esay载体相连接,并将连接产物转化入感受态细胞。感受态细胞的制备参照《分子克隆实验指南》[19]。
1.2.4 重组质粒的鉴定及测序 将转化细胞涂布在含25 μL(20 mg/mL)IPTG,15 μL(50 μg/mL)X-gal,100 μL(25 μg/mL)Ampr的 LB 平板上进行蓝白斑筛选,随机挑选白色菌斑 加入含Ampr抗生素的LB液体培养基中摇荡过夜进行菌液PCR检测阳性克隆。然后再通过质粒滞后及酶切鉴定后送测序。
1.2.5 生物信息学分析 将含有目的片段的菌液送生工测序。通过NCBI网站上的blast搜索引擎对测序结果进行同源性分析;利用ExPASy网站对StSnRK2.1基因的编码蛋白进行氨基酸组成、等电点等理化性质及亲/疏水性进行分析;利用CBS网站上提供的软件进行跨膜分析;利用WoLF PSORT网站对基因编码蛋白进行亚细胞定位的预测;利用DNAMAN软件完成对分子进化树的构建;利用PredictProteinPHD信息库对蛋白质序列进行二级结构预测;利用NetPhos2.0 Server进行蛋白磷酸化预测。
2 结果
2.1 马铃薯总RNA提取及RT-PCR扩增目的基因
提取马铃薯试管苗总RNA,结果如图1所示,提取的RNA28S,18S,5S条带清晰、完整,说明RNA没有发生降解,能够用于反转录。以反转录产物为模板,采用RT-PCR扩增目的基因,结果如图2所示,扩增出与预期大小相符的特异性条带,在1 000 bp左右。
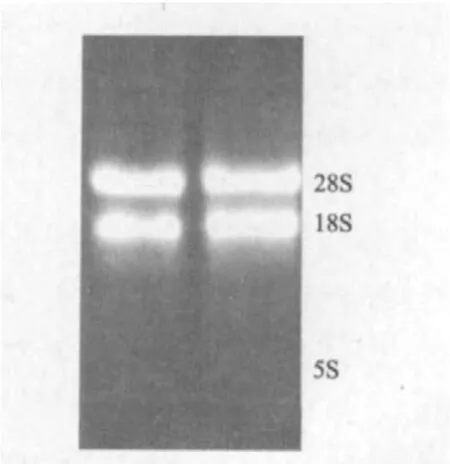
图1 马铃薯总RNA电泳图
2.2 目的基因的克隆与鉴定
将扩增得到的目的基因片段回收并且与pGEMTeasy 连接并转化到大肠杆菌DH5α中,通过蓝白斑筛选,挑选白色重组质粒进行菌落PCR鉴定,结果如图3所示,能够扩增出大小为1 000 bp左右的预期片段;用EcoR I 酶切质粒,能够切下预期的片段(图4),说明可能获得了马铃薯StSnRK2.1的cDNA片段。
2.3 StSnRK2.1基因cDNA序列分析
将重组质粒送上海生工进行测序,测序结果如图5所示,该基因开放阅读框全长1 008 bp,编码335个氨基酸,将基因命名为StSnRK2.1。
2.4 StSnRK2.1基因系统发生树的构建
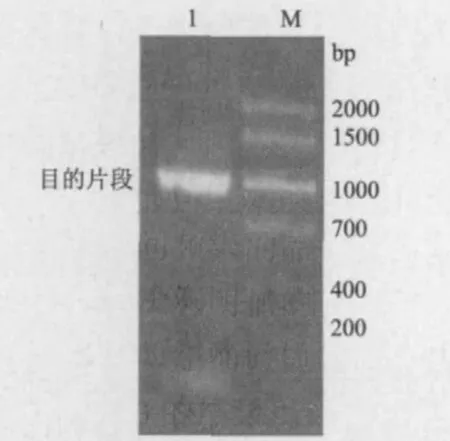
图2 StSnrk2.1基因扩增结果
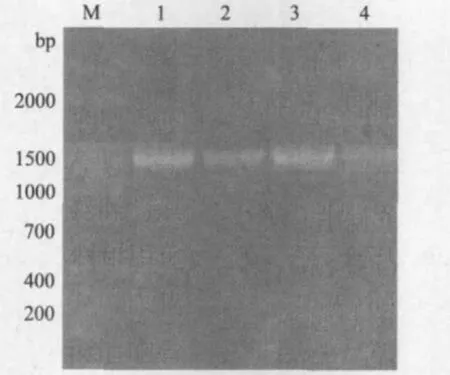
图3 StSnrk2.1基因菌液PCR结果

图4 酶切StSnRK2.1

图5 StSnRK2.1基因全长序列及编码氨基酸序列
利用DNAMAN软件构建了SnRK2基因的系统发生树,结果如图6。利用该软件对其进行多重序列比对,结果表明,StSnRK2.1与蒺藜苜蓿的SnRK基因相似性最高,为77.65%;与蓖麻SnRK基因相似性次之,达77.22%;与高粱SnRK基因相似性也较高,达75%。为了更好地了解不同物种中SnRK2的同源关系,将StSnRK2.1与其他植物一起进行基因的同源性分析,结果如图7所示。从图7可以看出,该基因家族在N端相对保守,而在C端则特异性极其明显。

图6 不同植物SnRk2基因的系统发生树
2.5 StSnRK2.1基因编码蛋白的一级结构及亲水性分析
对编码该基因的氨基酸种类及数量统计,如表1所示。氨基酸的亲水性分析数据表明该基因编码蛋白在第50个氨基酸附近具有最大亲水位置,在第200个氨基酸附近有最大疏水位置,且在较多位置上均表现出疏水性,可见该基因可能属于疏水性(图8),该基因的跨膜分析结果表明该肽链有3个跨膜区域,但是蛋白并未跨膜。
2.6 StSnRK2.1基因编码蛋白的二级结构分析及亚细胞定位
利用SOPMA网站预测马铃薯StSnRK2.1基因编码蛋白的二级结构,发现其二级结构主要有4种类型,分别为α-螺旋,β-折叠,延伸链和无规则卷曲,其中主要以α-螺旋为主,β-折叠最少,具体数据如表2所示。对于该基因的亚细胞定位分析是通过PSORT网站完成,由结果(表3)可知该基因出现在细胞质及微体中的可能性较大。
2.7 StSnRK2.1基因编码蛋白的三级结构预测及磷酸化位点分析
利用ExPASy网站上提供的软件对该基因的编码蛋白进行三级结构的预测,可以清楚地看到该蛋白具有丰富的α-螺旋结构,无规则卷曲也较明显(图9),从而验证了SOPMA网站对其二级结构的预测。该基因磷酸化位点的分析结果(图10)显示该肽链有7处丝氨酸磷酸化位点,2处苏氨酸磷酸化位点,以及3处酪氨酸磷酸化位点。
3 讨 论
在不同的生态条件下,植物体逐渐形成了不同的生理反应机制,尤其是对逆境胁迫的抵抗。研究表明,SnRK2家族基因在逆境环境中起重要作用[20-24]。本研究利用同源克隆的方法从马铃薯试管苗RNA中成功克隆得到SnRK2的同源基因—StSnRK2.1,将测序所得碱基序列与预测碱基序列通过DNAMAN软件进行比对,发现二者相似性达98.61%,其中只有13个碱基发生变化,发生这种现象的原因可能是因为克隆时所使用的模板与网站上提交的序列不是来自同一品种马铃薯;另一种原因可能是在扩增过程中碱基发生了突变。但是当对两个序列的氨基酸进行比对时发现二者相似度高达100%,因此可以猜测碱基不同是由于马铃薯品种不同而使同一氨基酸的密码子不同导致的。此外,将StSnRk2.1基因的编码蛋白与另外10个物种的蛋白质序列进行同源性比较时发现,这些基因在N端具有一定的保守性,而在C端则高度特异,据此推测该基因在进化过程中是比较保守的,对逆境胁迫的响应可能与SnRK2家族中的其他基因一样具有一定的一致性。
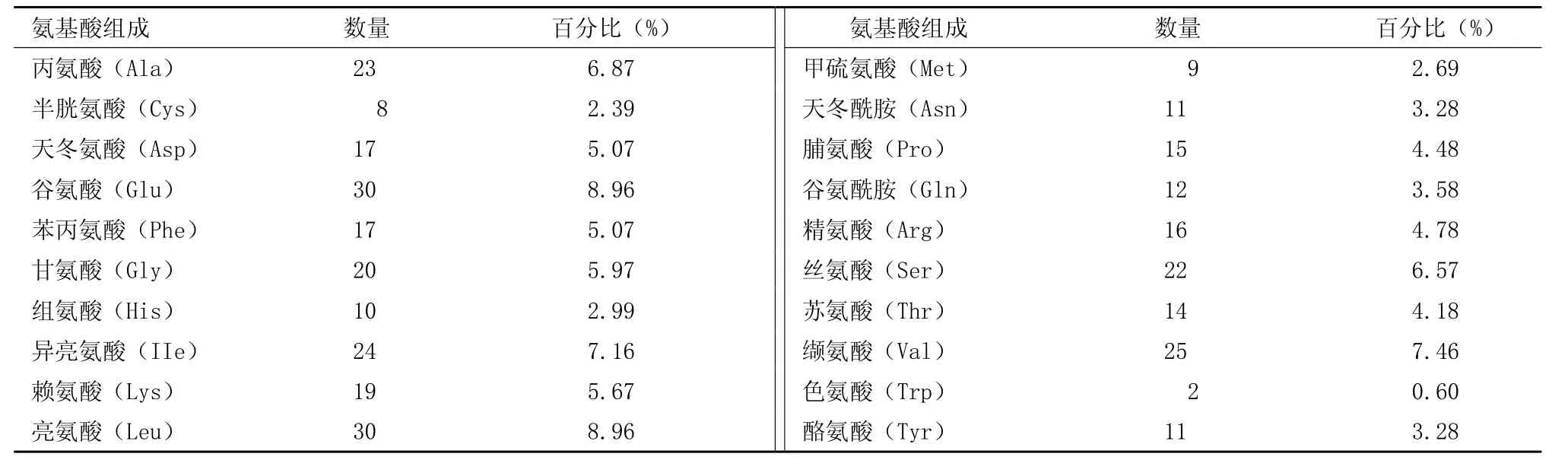
表 1 StSnrk2.1基因编码蛋白的氨基酸含量
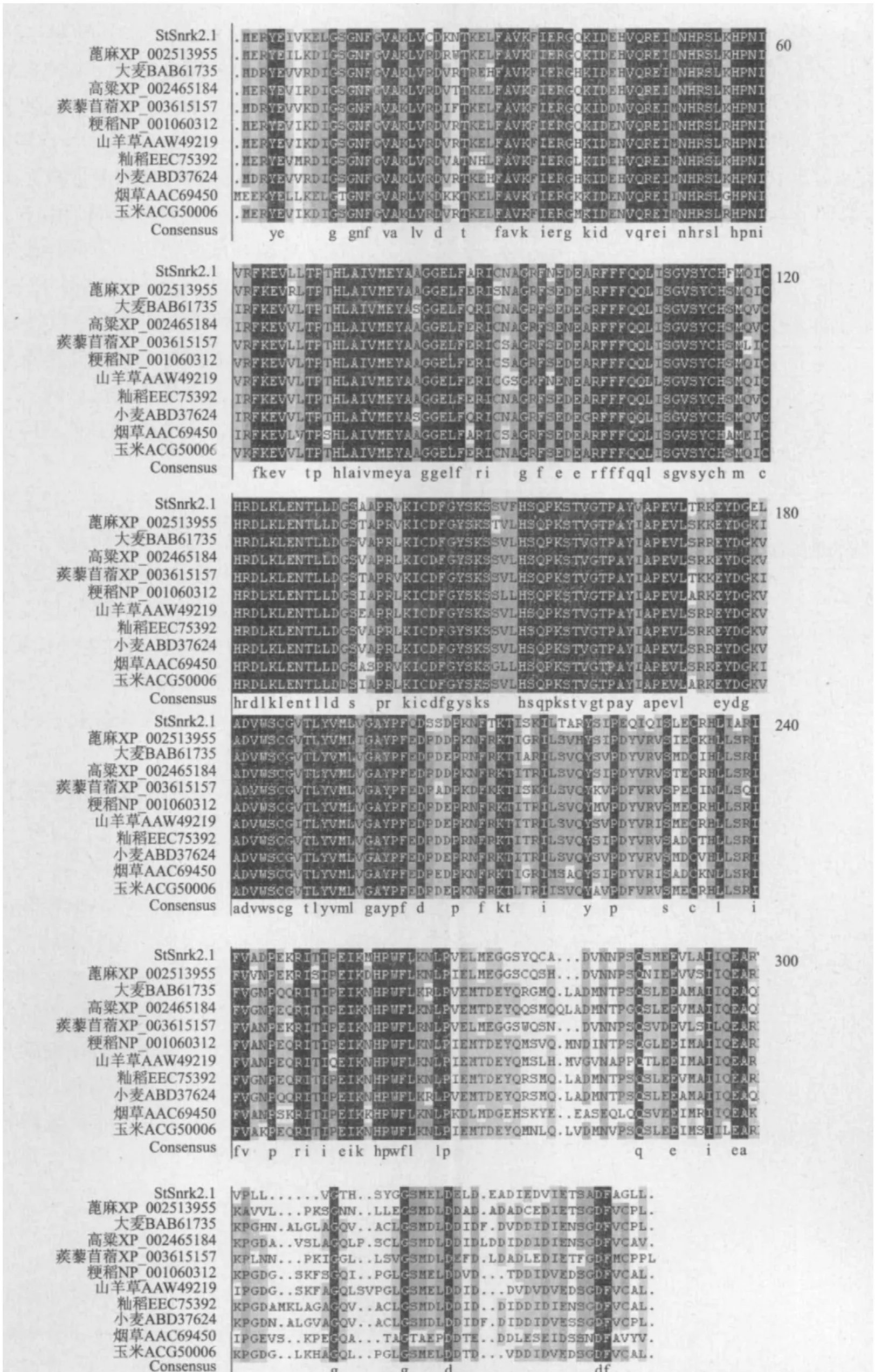
图7 StSnRK2.1基因与其他物种中Snrk2家族基因进行多序列比对
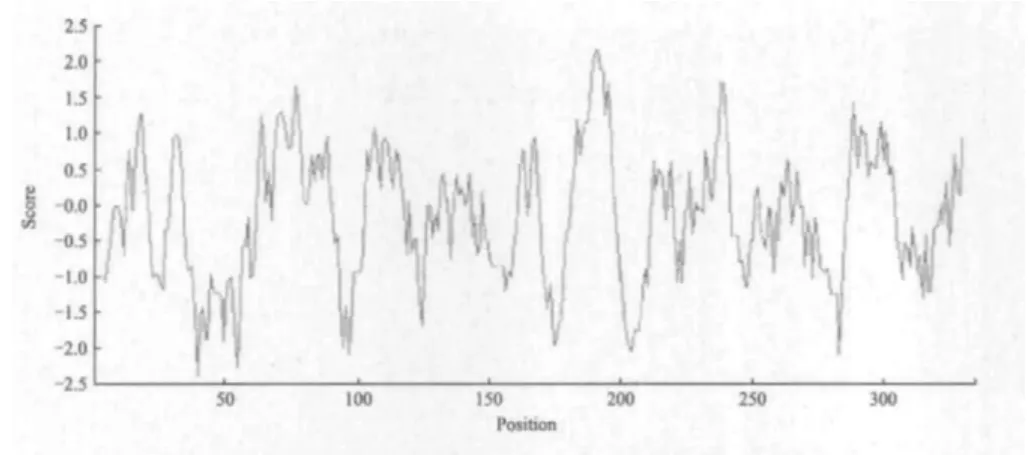
图8 StSnRK2.1基因编码蛋白的亲水性分析

表2 StSnRK2.1基因编码蛋白的二级结构分析

表3 StSnRK2.1基因编码蛋白的亚细胞定位分析
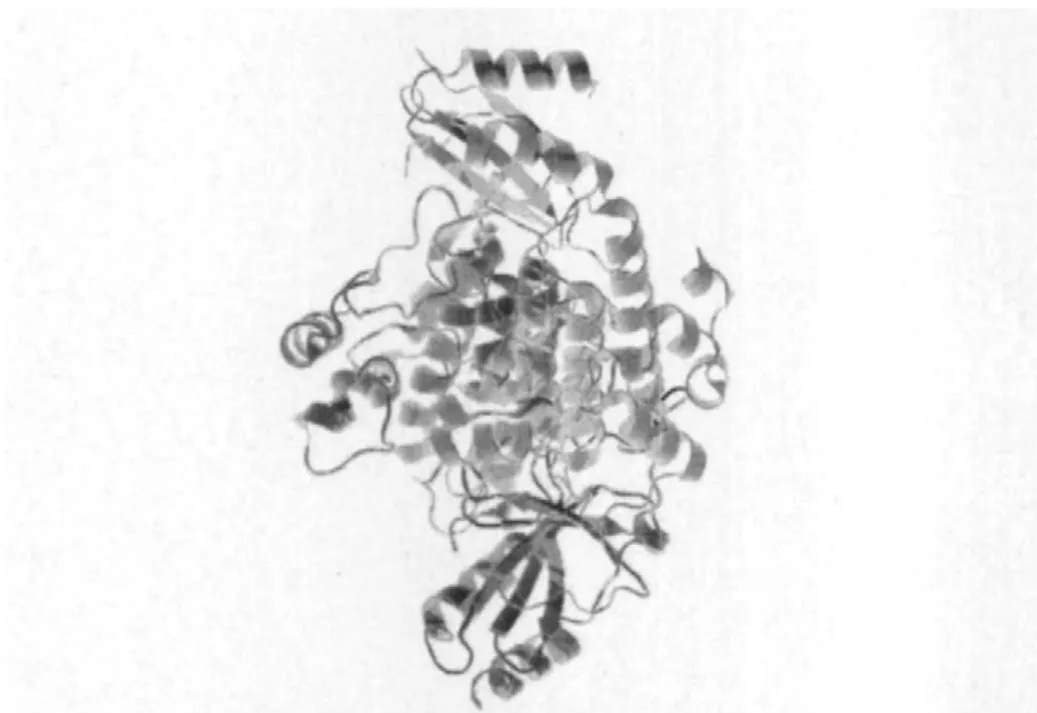
图9 StSnRK2.1基因编码蛋白的三级结构
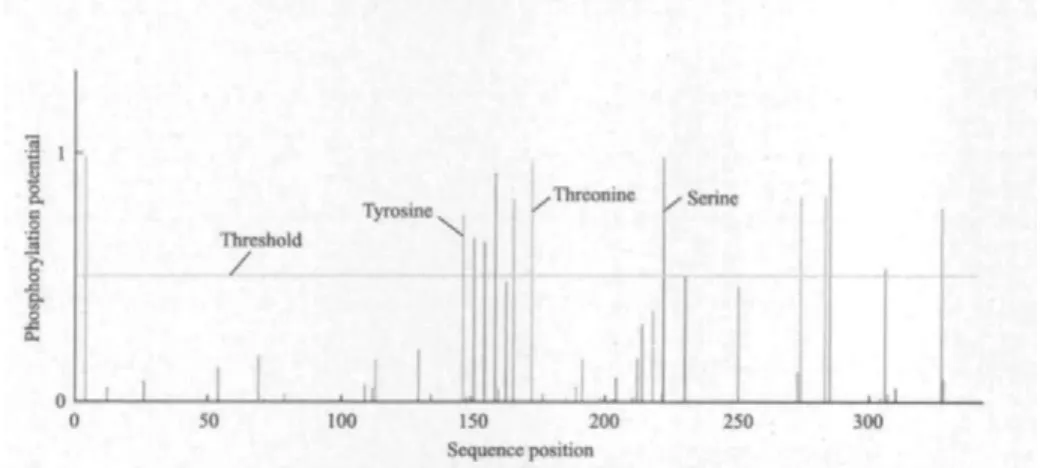
图10 StSnRK2.1基因磷酸化位点分析
蛋白质的磷酸化是最普遍、最重要的一种蛋白翻译后修饰方式。它是植物在逆境胁迫条件下体内能量代谢和信号传递中的重要生化反应[25]。一般来说,多肽链的氨基酸潜在的磷酸化位点越多,发挥更多功能的可能性就越大。从分析所得的数据中,该肽链有7处丝氨酸磷酸化位点,2处苏氨酸磷酸化位点,以及3处酪氨酸磷酸化位点可知该肽链潜在的磷酸化位点较多。由此也可推测,StSnRK2.1可能也是通过可逆的磷酸化反应对逆境胁迫进行感知和应答的。
4 结论
本研究从马铃薯试管苗中克隆得到1个SnRK2.1基因,命名为StSnRK2.1。通过生物信息学分析,得到了该基因开放阅读框全长及其编码氨基酸,蛋白质分子量、等电点、蛋白质二级结构以及亲疏水性,通过分析认为其为膜内蛋白,亚细胞定位显示该基因出现在细胞质及微体中的可能性较大。肽链有7处丝氨酸磷酸化位点,2处苏氨酸磷酸化位点,以及3处酪氨酸磷酸化位点,推测该基因在植物抗逆中起一定作用。
[1] Umezawa T, Yoshida R. SRK2C, a SNF1-related protein kinase 2,improves drought tolerance by controlling stress-responsivegene expression inArabidopsis thaliana[J] . PNAS, 2004, 101(49),17306-17311.
[2] Boudsocq M, Lauriere C. Osmotic signal in plants multiple pathways mediated by emerging kinase families [J] .Plant Physiology, 2005,138:1185-1194.
[3] Kobayashi Y, Yamamoto S, Minami H, et al. Differential activation of the rice sucrose non fermenting l-related protein kinase2 family by hyperosmotic stress and abscisic[J].The Plant Cell, 2004, 16:1163-1177.
[4] Huai JL, Wang M, He JG, et al. Cloning and characterization of the SnRK2 gene family fromZea mays[J]. Plant Cell Reports, 2008,27:1861-1868.
[5] Cohen P. Protein phosphorylation and hormone action[J]. Proceedings of the Royal Society B.Biological Sciences, 1988, 234:115-144.
[6] Jaglo-Ottosen KR, Gilmour SJ, ZaRKa DG et al.ArabidopsisCBF1 over expression induces COR genes and enhances freezing tolerance[J]. Science, 1998, 280(5360):104-106.
[7] Passioura JB. Environmental biology and crop improvement[J].Functional Plant Biology, 2002, 29:537-546.
[8] Ingram J, Bartels D. The molecular basis of dehydration tolerance in plants[J]. Annu Rev Plant Physiol Plant Mol Biol, 1996, 47:377-403.
[9] Schafleitner R, Gutierrez Rosales RO, Gaudin A. Capturing candidate drought tolerance traits in two native Andean potato clones by transcription profiling of field grown plants under water stress[J].Plant Physiol Biochem, 2007, 45(9):673-690.
[10] Fujita Y, Fujita M, Satoh R, et al. AREB1 Is a transcription activator of novel ABRE-dependent ABA signaling that enhances drought stress tolerance inArabidopsis[J]. Plant Cell, 2005, 17(12):3470-3488.
[11] Karim S, Aronsson H, Ericson H, et al. Improved drought tolerance without undesired side effects in transgenic plants producing trehalose[J]. Plant Mol Biol, 2007, 64(4):371-386.
[12] Gosal SS, Wani SH, Kang MS. Biotechnology and drought tolerance.Journal of Crop Improvement, 2009, 23:19-54.
[13] 唐先兵, 赵恢武, 林忠平. 植物耐旱基因工程研究进展[J].首都师范大学学报:自然科学版, 2009, 23(3):47-51.
[14] 李娜, 李莉, 陈光辉. 作物抗旱机理及其相关基因的研究进展[J].长江大学学报:自然科学版, 2011, 8(3):239-243.
[15] Chaves MM, Pereira J, Maroco J. How plants cope with water stress in the field?[J]. Photosynthesis and Growth. Ann Bot, 2002, 89(7):907-916.
[16] Xu ZZ, Zhou GS, Shimizu H. Plant responses to drought and rewatering[J]. Plant Signaling & Behavior, 2010, 5(6):649-654.
[17] 张树珍, 王自章. 物耐旱的分子基础及植物耐旱基因工程的研究进展[J].生命科学研究, 2001, 5(3):134-140.
[18] 刘欣, 李云. 转录因子与植物抗逆性研究进展[J].中国农学通报 , 2006, 22(4): 61-65.
[19] Sambrook J, Fritsh EF, Maniatis T.分子克隆实验指南[ M] .第2版.北京:科学出版社, 1992.
[20] 吴峻岩, 王茂广, 吴能表, 等. 植物中的CDPK/SnRK蛋白激酶家族[J].激光生物学报, 2004, 13(4):265-269.
[21] 王永波, 高世庆, 唐益苗, 等.植物蔗糖非发酵. 1相关蛋白激酶家族研究进展[J].生物技术通报, 2010(11):7-17.
[22] 李利斌, 开昌, 李现刚. 三个新的玉米SnRK2基因的鉴定和特征分析[J]. 山东农业科学, 2009, 12:7-11.
[23] Amaguehi K, Shinmkik K. Transcriptional regulatory networks incellular responses and tolerance to dehydraliotl and cold stress).Annu Rev Plant Bio, 2006, 57:781-803.
[24] 连魏卫, 唐益苗, 高世庆, 等.小麦TaSnRK2.9 蛋白激酶基因克隆与生物信息学分析[J]. 中国农学通报, 2011 , 27(33): 6-12.
[25] 徐蓓, 郭丽香, 赵昌平, 等. 长穗偃麦草EeSnRK2.6基因克隆及生物信息学分析[J]. 麦类作物学报, 2012, 32(1):36-43.
