观光木ISSR-PCR反应体系的建立及优化
2012-12-29吴雪琴徐刚标王家庆
吴雪琴,徐刚标,梁 艳,王家庆
观光木ISSR-PCR反应体系的建立及优化
吴雪琴1,徐刚标1,梁 艳1,王家庆2
(1.中南林业科技大学 生命科学与技术学院,湖南 长沙 410004;2.韶关国有河口林场,广东 韶关 512532 )
利用正交设计实验对影响观光木ISSR-PCR扩增的主要因素(模板DNA、引物、dNTPs、Mg2+的浓度,TaqDNA聚合酶的用量以及退火温度)进行优化, 建立观光木ISSR-PCR反应的最佳体系。结果表明:最佳反应体系(20 μL)为:模板DNA 50 ng,引物0.4 μmol/L,dNTPs 0.25 mmol/L,MgCl22.5 mmol/L,Taq DNA聚合酶1.25 U。扩增反应程序为:94 ℃ 预变性5 min;94 ℃变性 1 min,55 ℃ 退火 30 s,72 ℃ 延伸 90 s,35个循环;72 ℃ 延伸7 min;4 ℃ 保存。该反应体系的建立,为观光木遗传多样性分析提供了客观可靠的方法。
观光木;正交优化; ISSR-PCR;遗传多样性
观光木Tsoongiodendron odorum Chun为木兰科观光木属常绿大乔木,是单种属古老孑遗树种,是国家二级重点保护植物[1],对研究古代植物区系、古地理、古气候都有重要的科学价值[2]。该种分布区很广,湖南、江西南部、福建、广东、广西、云南东部、海南等地均有零星分布,其中,以南岭山地种群数量相对较多。近年来,观光木研究主要集中在生物学特性、育苗造林技术、生态学特性等方面[3],仅见黄久香等[4-5]采用RAPD(randomly amplified polymorphic DNA)研究观光木遗传多样性报道,但种群数量少,研究结果难以代表整个物种的变异水平。
基于PCR技术的ISSR( inter simple sequence repeat)标记已成功地应用于种群遗传结构分析,与RAPD标记相比,ISSR比RAPD重复性好[6]。为了建立观光木ISSR-PCR反应的最佳体系,本研究利用正交设计实验对影响观光木ISSR-PCR扩增的重要因素进行优化,旨在为采用ISSR标记研究观光木种群遗传多样性奠定实验基础。
1 材料与方法
1.1 材 料
材料于2011年4月底采集于贵州省黎平县。采回的观光木新鲜叶片置于-70 ℃冰箱中保存。参照加拿大哥伦比亚大学(UBC)2006年公布的ISSR引物序列,由上海英俊生物技术有限公司合成。经初步筛选出的854号引物,即(TC)8RG作为此次反应体系优化实验的固定引物。
1.2 方 法
1.2.1 基因组DNA提取与检测
观光木叶片基因组DNA提取,采用改良的CTAB法及参考文献[4-8]并略作改动。用Eppendorf公司的Biophotometer核酸蛋白分析仪检测DNA含量和纯度,用0.8﹪琼脂糖凝胶电泳检测提取的总DNA质量,用G-BOX紫外凝胶成像系统观察DNA是否污染并拍照记录。
1.2.2 ISSR-PCR扩增
以提取的观光木 DNA样品为模板,进行ISSR-PCR扩增。扩增程序参考相关文献[7]:94 ℃预变性 5 min,接着进行 35个循环:94 ℃变性 1 min,53 ℃退火 30 s,72 ℃延伸 90 s;循环结束后,72 ℃延伸 7 min,4 ℃保存。
反应体系总体积为20 μL,其中包括1×PCR Buffer,其它成分(模板DNA、dNTPs、Mg2+、引物、TaqDNA聚合酶)按照试验设计所分析的浓度加入,不足20 μL用超纯水补足。
1.2.3 ISSR-PCR扩增体系的单因素试验设计
为保证试验的准确性、可重复性和ISSR-PCR扩增效果,本实验对模板DNA,Taq DNA聚合酶、Mg2+、dNTPs、引物等 5个主要影响因子进行优化筛选,通过单因素实验设计(模板DNA10~60 ng、Taq DNA聚合酶0.5~1.75 U、Mg2+1.0~3.5 mmol/L、dNTPs 0.1~0.35 mmol/L、引物0.1~0.6 μmol/L),分别设置6个梯度,以基本扩增体系为基础,每次改变一个因素,以确定该因素对ISSR结果的影响。
1.2.4 ISSR-PCR扩增体系的正交试验设计
根据单因素实验的结果,设计正交试验方案,最终确定ISSR-PCR扩增的最佳反应体系。
1.2.5 ISSR-PCR扩增产物的检测
扩增产物采用1.5﹪琼脂糖凝胶电泳检测,缓冲液1×TAE,电压5 V/cm。当溴酚蓝指示剂距离前沿约2~3 cm时停止电泳,用G-BOX紫外凝胶成像系统观察并拍照记录。
2 结果与分析
2.1 DNA纯度与浓度
将提取的基因组 DNA稀释 100倍,在UnicoTM UV-2102PC型紫外分光光度计上检测260 nm和 280 nm的吸光值,计算所提取 DNA的纯度和浓度。结果如表1所示,所提取的DNA的OD260nm/OD280nm值在1.6和2.0之间,浓度和纯度都符合ISSR标记技术对DNA质量的要求[8]。
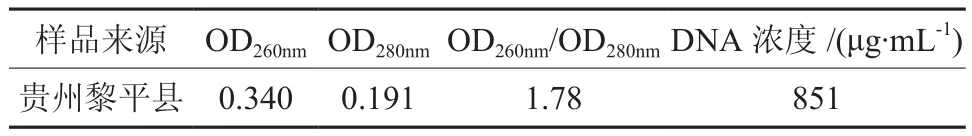
表1 DNA的紫外检测结果Table 1 The ultraviolet absorption test of DNA
2.2 模板DNA用量对ISSR- PCR扩增结果的影响
20 μL PCR反应体系,设定6个模板DNA浓度 (ng/20 μL)梯度 (10、20、30、40、50、60),扩增检测结果(如图1)表明:在其它条件一致的情况下,DNA模板量对PCR扩增的影响较小,模板DNA在10~60 ng内均能扩增出条带。在浓度低于和等于40 ng时,扩增的条带亮度相对较低,清晰度不高且条带较少。模板浓度50 ng与60 ng相比,50 ng/20 μL扩增出的条带清晰明亮,多态性最高。因此适宜的模板DNA浓度为 50 ng/20 μL。

图1 不同模板 DNA浓度对ISSR-PCR的影响Fig 1 Effects of different template concentrations on ISSR-PCR
2.3 Taq DNA聚合酶浓度对ISSR-PCR扩增结果的影响
Taq DNA聚合酶的用量对扩增反应起着决定性的影响作用。本实验设置了 6个Taq DNA聚合酶浓度(U)梯度 (0.5、0.75、1.0、1.25、1.5、1.75),结果(见图2)表明:在 20 μL反应体系中,Taq DNA聚合酶在6个递度范围内均得到清晰的扩增条带,酶浓度在 1.25U时扩增得到的条带最多,因此适宜的 Taq DNA聚合酶浓度为 1.25 U/20 μL。
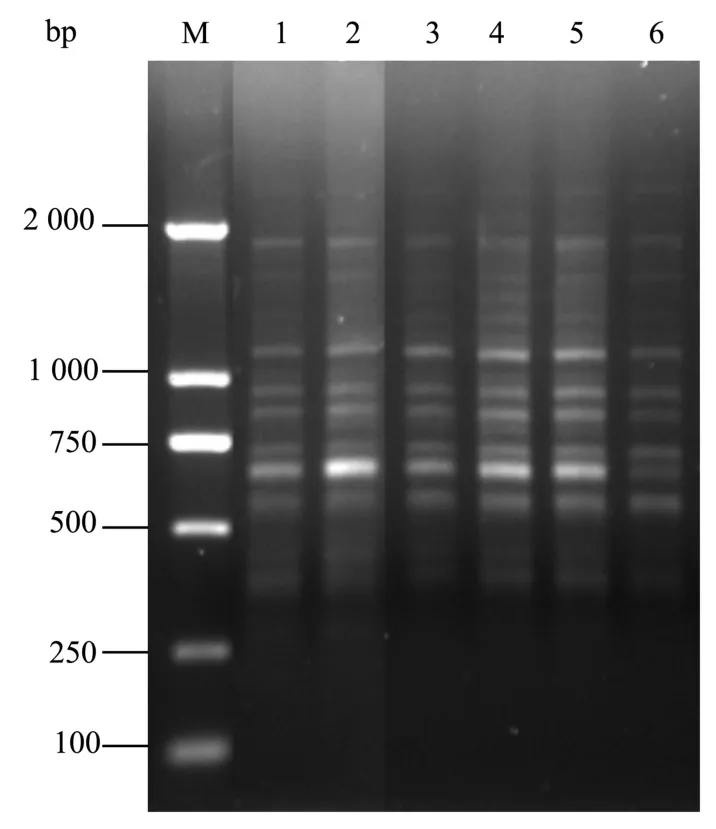
图2 不同 Taq DNA聚合酶浓度对 ISSR-PCR的影响Fig.2 Effects of different Taq DNA polymerase concentrations on ISSR-PCR
2.4 Mg2+浓度对ISSR-PCR扩增结果的影响
Mg2+是Taq DNA聚合酶的激活剂,酶对 Mg2+的浓度非常敏感,其浓度将直接影响Taq酶的活性,本实验设置了6个Mg2+浓度(mmol/L)梯度(1.0、1.5、2.0、2.5、3.0、3.5),由扩增结果(如图 3)可知:在不同浓度下扩增出多态性条带有明显差异,浓度在2.0 mmol/L时,扩增的条带最多且清晰明亮;浓度大于2.0 mmol/L或小于2.0 mmol/L时,均有部分条带缺失,因此选择适宜的Mg2+浓度为 2.0 mmol/L。

图3 不同 Mg2+浓度对 ISSR-PCR的影响Fig.3 Effects of different Mg2+ concentrations on ISSR-PCR
2.5 dNTPs浓度对ISSR-PCR扩增结果的影响
dNTPs是PCR反应的原料,其浓度取决于扩增片段的长度。本实验设计了6个浓度(mmol/L)梯度 (0.1、0.15、0.2、0.25、0.3、0.35),扩增结果(如图4 )表明:在不同浓度下,扩增的结果有较大差异。浓度越高的扩增出的条带越不清晰,浓度过低的扩增的条带也不理想,浓度为0.20 mmol/L时,扩增的条带相对较多且清晰。因此选择适宜的dNTPs反应浓度为0.20 mmol/L。

图4 不同 dNTPs浓度对 ISSR-PCR的影响Fig.4 Effects of different dNTPs’s concentrations on ISSR-PCR
2.6 引物浓度对 ISSR-PCR扩增结果的影响
引物用量对扩增产物的质量有着至关重要的影响。本实验设计了6个引物浓度(μmol/L)梯度(0.1、0.2、0.3、0.4、0.5、0.6), 扩 增 结 果 (见图5)表明:不同的引物浓度对扩增结果有较大的影响,浓度越低扩增条带越不清晰,且有部分条带缺失,浓度在0.4~0.6 μmol/L时,扩增效率逐渐增高,条带较清晰,引物浓度为0.6 μmol/L时,条带相对最多最清晰,因此适宜的引物浓度为 0.6 μmol/L。
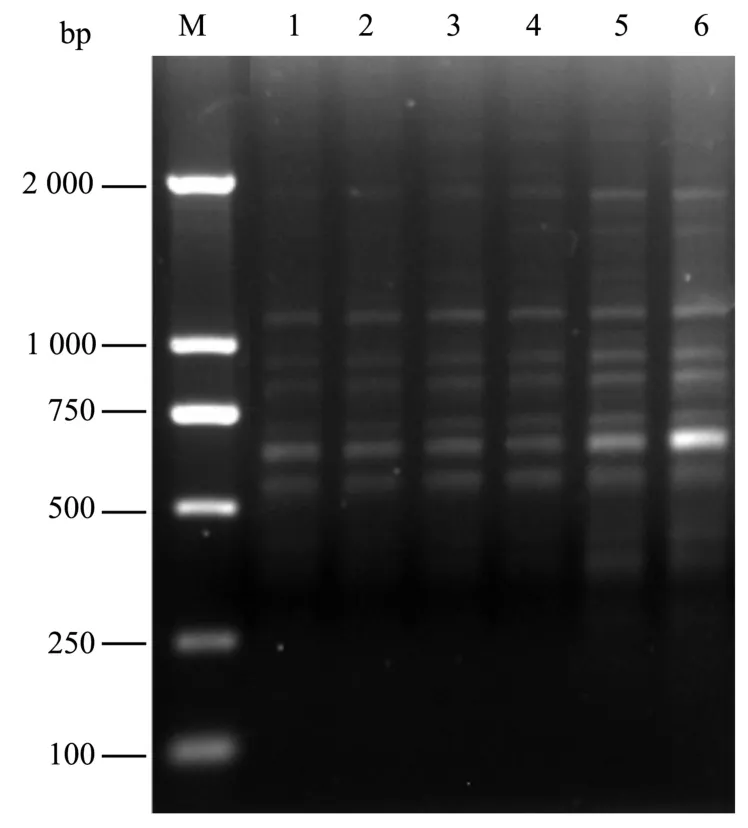
图5 不同Primer浓度对 ISSR-PCR的影响Fig.5 Effects of different primer concentrations on ISSR-PCR
2.7 ISSR-PCR正交实验结果
由单因素实验结果,按照正交试验表L18(35)设计5因素3水平实验进一步优化反应体系,结果如图6所示。在单因素优化的基础上,进行正交设计实验能够扩增出较清晰的条带。在18个处理组中,第8组设计的实验数据扩增的效果最好,谱带最多而且清晰明亮,多态性高。
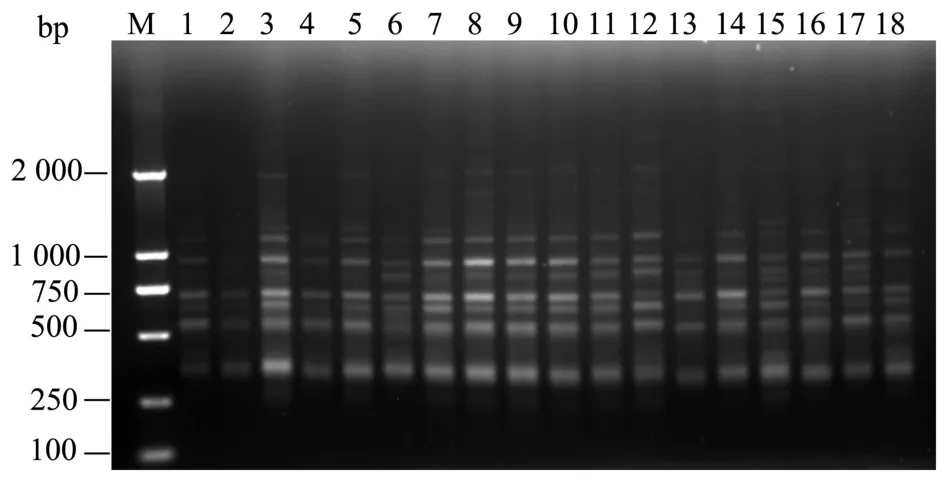
图6 ISSR-PCR正交设计电泳图谱Fig.6 Band patterns of orthogonal design of optimal ISSRPCR system
2.8 退火温度对ISSR-PCR的影响
不同的引物有不同的退火温度,引物UBC854以其Tm(Tm:56)值为中心,设置6个退火温度梯度(53、54、55、56、57、58 ℃)进行PCR扩增,由结果(如图 8)得出,引物UBC854在55 ℃时,扩增出的条带多而亮,而低于或者高于 55 ℃,扩增的条带相对较少且亮度浅。因此,退火温度的高低也直接影响着扩增反应的结果,观光木ISSR反应体系中引物UBC854最适宜的退火温度为 55 ℃。
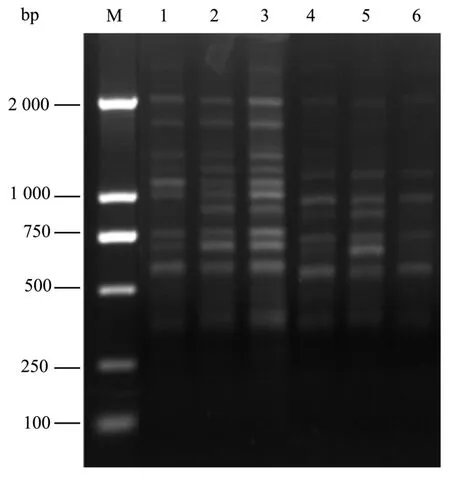
图7 不同退火温度对 ISSR-PCR的影响Fig.7 Effects of different annealing temperatures on ISSR-PCR
2.9 最优体系的验证
为了验证优化后的体系,选取观光木的另外13个样品所提取的DNA采用L18(35)中第8组设计的数据进行PCR扩增,扩增结果如图8所示。从图8可知:条带的多态性与清晰度好,说明该体系能很好地满足观光木的ISSR遗传多样性研究。
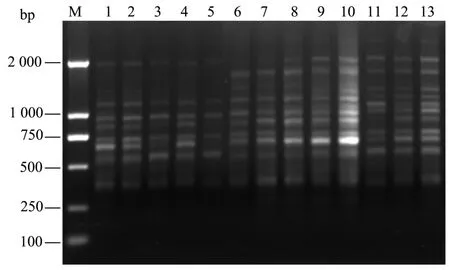
图8 其他13个样品扩增结果Fig.8 Electrophoresis of PCR products of all 13 plant materials
3 结 论
ISSR扩增受模板、TaqDNA聚合酶、Mg2+、dNTPs、引物等因素影响,退火温度的高低也直接影响着扩增反应的结果,不同引物的最适退火温度不同。本研究通过正交试验设计对影响观光木ISSR-PCR扩增的重要因素进行优化,建立了观光木ISSR-PCR反应的最佳体系(20 μL):模板DNA 50 ng、 TaqDNA聚合酶1.25 U、MgCl22.5 mmol/L、dNTPs 0.25 mmol/L、引物0.4 μmol/L。扩增反应程序:94 ℃ 预变性5 min;94 ℃变性 1 min,55 ℃ 退火 30 s,72 ℃ 延伸 90 s,35个循环;72 ℃ 延伸7 min;4 ℃ 保存。该反应体系的建立为观光木种质资源分类、遗传多样性分析以及分子谱系地理学研究提供了更客观可靠的方法。
[1] 傅立国,金鉴明.中国植物红皮书— —稀有濒危植物:第一册[M].北京:科学出版社,1992:454-455.
[2] 郭承则.珍稀濒危树种观光木的引种研究[J].湖南林业科技,1996,23(3):67-68.
[3] 黄松殿,覃 静,秦武明,等.珍稀树种观光木生物学特性及综合利用研究进展[J].南方农业学报,2011,42(10):1251-1254.
[4] 黄久香,庄雪影.观光木种群遗传多样性研究[J].植物生态学报, 2002, 26 (4) 413-419.
[5] 黄久香, 庄雪影.华南三地观光木遗传多样性的RAPD 分析[J].华南农业大学学报:自然科学版,2002,23(2):53-57.
[6] Arcade A F, Anselin P, Faivre Rampant, et al.Application of AFLP,RAPD and ISSR markers to genetic mapping of Euro-pean and Japanese larch [J]. Theor. Appl. Genet., 2000,100:299-307.
[7] 张维铭.现代分子生物学实验手册[M].北京:科学出版社,2003.
[8] 徐刚标, 禹玉婷, 申响保. 湘鄂西地区珙桐天然群体遗传结构的研究[J].中南林业科技大学学报,2006,26(6):5-9.
Establishment and optimization of Tsoongiodendron odorum ISSR-PCR reaction system
WU Xue-qin1,XU Gang-biao1,LIANG Yan1, WANG Jia-qing2
(1.School of Life Science and Technology,Central South University of Forestry and Technology,Changsha 410004,Hunan,China;2. State-owned Hekou Forest Farm, Shaoguang 512532, Guangdong, China)
Through the orthogonal design experiments, the important factors of the inter-simple sequence repeat PCR (ISSR-PCR)amplification system, such as DNA template,primers,dNTPs,Mg2+concentration,dose of TaqDNA polymerase and annealing temperature fit for Tsoongiodendron odorum were optimized. The results show that the optimal reaction system by the volume of 20 μL was established as follows: 50 ng DNA template,0.4 μmol/L primers,0.25 mmol/L dNTPs,2.5 mmol/LMgCl2,1.25 U Taq DNA polymerase. The PCR procedure was: pre-denaturing at 94 ℃ for 5 minutes,35 cycles of denaturation at 94 ℃ for 1 minute,annealing at 55 ℃ for 30 seconds and extension at 72 ℃ for 90 seconds,with 7 minutes final extension at 72 ℃ and then stored at 4 ℃ .The orthogonal-based ISSR-PCR reaction system provided a reliable method for genetic diversity analysis in Tsoongiodendron odorum.
Tsoongiodendron odorum;orthogonal design;ISSR-PCR; genetic diversity
S718.46
A
1673-923X (2012)07-0076-04
2012-03-14
国家林业公益性行业科研专项(201104033)
吴雪琴(1987—),女,四川乐山人,硕士研究生;主要从事分子种群遗传学研究
徐刚标(1965—),男,安徽枞阳人,教授,博士;主要从事森林遗传学及林业生物技术研究
[本文编校:吴 毅 ]
