嗜盐菌群对菲的降解及萘双加氧酶基因的表达规律
2012-12-25王立华宁大亮清华大学环境学院北京00084河北北方学院应用化学研究所河北张家口075000
何 芬,王立华,宁大亮,郭 光,王 慧* (.清华大学环境学院,北京 00084;.河北北方学院应用化学研究所,河北 张家口 075000)
嗜盐菌群对菲的降解及萘双加氧酶基因的表达规律
何 芬1,王立华2,宁大亮1,郭 光1,王 慧1*(1.清华大学环境学院,北京 100084;2.河北北方学院应用化学研究所,河北 张家口 075000)
从胜利油田富集了一个能够降解多环芳烃的嗜盐菌群,通过克隆文库技术解析了菌群萘双加氧酶(ndo)基因的多样性.结果表明,该嗜盐菌群有6种ndo基因型,其中3种主要基因型(占总克隆子92.7%)与经典nah-like基因的相似度为89%.采用real-time RT-PCR等技术分析了不同盐度下菲降解过程中ndo基因的表达量、降解速率、生长曲线以及菲的生物可利用度,探索了菌群对菲的降解及ndo基因的表达规律.结果表明,当盐度由10%升高到20%,菌群完全降解100mg/L菲的时间从6d延长到9d,菌群生长的迟滞期由1d延长为3d.溶解菲的浓度在菌群生长过程中呈增加的趋势.Ndo基因的表达量在降解过程中呈先降低后随溶解菲浓度升高而升高的趋势,表明高浓度菲在初始阶段可能抑制ndo基因的表达.单因素方差分析(n=3, P>0.05)表明溶解菲浓度在10%和20%盐度下没有显著差异,表明一定范围的盐度不会影响菌群降解过程中菲的溶解度.菌群ndo基因的相对表达量在10%盐度下较在20%盐度下高,说明盐度对嗜盐菌群功能基因的表达具有抑制作用.
嗜盐菌群;基因表达;生物可利用度;菲的溶解度;萘双加氧酶
多环芳烃(PAHs)由于具有潜在致畸性、致癌性和遗传毒性而受到广泛关注[1].PAHs的污染常常伴随着盐环境和大幅盐度波动[2-3].高盐度和盐度的波动往往导致非嗜盐微生物的修复效率明显降低,甚至丧失修复能力[2,4].利用嗜盐微生物降解或修复盐环境下PAHs污染是一条可行的解决途径.研究发现,较高的盐度会限制菌群降解石油、苯和甲苯等污染物的能力[3,5-6],但是长时间暴露在污染下,微生物对污染物的降解会增加[3,5].PAHs的生物降解往往受到其生物可利用度的限制(溶解度低或者易于被土壤吸附),而盐度会影响PAHs的溶解度[7].Kleubsteuber等[3]发现,随着盐度在 0~20%间波动,以石油为底物的菌群的代谢多样性会改变,优势菌属也会随之改变.可见,PAHs的生物降解是一个易受盐度影响的复杂过程.但是目前缺乏对高盐环境下 PAHs的生物降解过程及其分子机制的深入探索.
本文采用克隆文库和real-time RT-PCR等方法,旨在解析嗜盐菌群中萘双氧酶(ndo)基因的多样性,并通过分析不同盐度下菲降解过程中萘双氧酶(ndo)基因的表达量、降解速率、生物量以及溶解态菲浓度变化的动力学关系,探讨了嗜盐菌群对菲的生物降解及 ndo基因的表达规律,为建立和发展复杂环境中PAHs生物降解动力学及生物修复方法和策略提供理论依据.
1 材料与方法
1.1 材料
菲 (98%纯,Aldrich Chemical Co., USA),菌株E.coli DH5α和感受态细胞PUC19(天根生化科 技 ),质 粒 pMD®19-T Simple Vector (TAKARA),反转录试剂盒(TAKARA),Real-time SYBRGreen染料(TAKARA),Real-time 仪器(Bio-Rad, America),高效液相色谱(岛津,10Avp).
油污染土壤样品采自中国山东省胜利油田,-80℃贮存.
按照Zhao等[8]的方法配制不同盐度的无机盐培养基(SSDM,sea salt-defined media).当SSDM 培养基中添加 0.01%的酵母粉定义为SSDMY培养基.菌株E.coli DH5α生长于LB培养基上.
1.2 菌群的富集
按照Zhao等[8]的方法采用添加400mg/L菲和10%盐度的SSDM培养基富集中度嗜盐菌群.
1.3 降解曲线和溶解菲的测定
将20mL富集的菌群在6000r/min、常温离心10min后接种到灭菌的SSDMY培养基中,同时设置加灭活菌液的对照样(100mg/L菲)进行降解实验.2组降解实验条件分别是 100mg/L菲,10%盐度和100mg/L菲,20%盐度.在降解过程中定时取样同时测菌群的OD值、菲的浓度和基因表达量.每组实验设3个平行样.
总菲测定:从培养液中均匀取出 1000µL菌液,加入2倍体积的二氯甲烷进行萃取.萃取液用0.22µm滤膜过滤后,采用HPLC测定萃取液中菲浓度.
溶解菲测定:采用带注射器的 0.22µm的纤维素膜过滤样品以去除微粒菲,使用前过滤同样的样品 2mL[9-10].去除微粒菲的样品按照总菲测定的方法处理.
HPLC条件:色谱柱为 C18反相柱(依利特,Hypersil BDS C18,250mm×4.6mm×5μm),测定时间 20min;流动相为 90%甲醇;流速为1.0mL/min;柱温为 35℃;进样量为 20μL;检测波长254nm.测定结果为3个平行样的平均值.
1.4 菌群的生长量
取 4mL菌液检测,采用紫外分光光度仪(UV-2401PC,岛津,日本)在600nm下检测嗜盐菌群的生长.菌群的OD600采用式(1)计算:

式中,样品 OD600指所取菌液的 OD600,样品上清液OD600指菌液离心后上清液的OD600.把培养基中菲浓度带入菲浓度与 OD600标准曲线中计算获得样品菲 OD600.10%盐度和 20%盐度下菲浓度与OD600标准曲线分别是:

式中: y为OD600值,x为菲的浓度.测定结果为3个平行样的均值.
1.5 Ndo基因的克隆文库
收集富集的菌液,经过DNA提取,PCR及产物纯化、连接至质粒pMD®19-T Simple Vector、转化获得单克隆.扩增引物是简并引物 NAH-F和 NAH-R[11],反应体系为 50μL:25μL r-Taq (TAKARA),模板 DNA 1μL,引物(10μmol/L)各1μL,ddH2O 22μL.PCR 程序如下:95℃预变性10min;95℃变性 1min,49℃退火 1min,72℃延伸2min,30个循环;最后72℃延伸10min.采用蓝白斑和菌落PCR筛选含有目的基因的单克隆.将含有ndo基因片段的71个克隆子送往北京三博远志生物技术公司测序,构建克隆文库.采用Mega4.0构建系统发育树,构建方法为Neighborjoining.
1.6 标准质粒的构建
参考Cebron等[12]方法构建2个标准品质粒,分别为目标基因ndo和内参基因16S rDNA.从ndo基因的克隆文库中挑选覆盖率最大的单克隆扩大再培养后提取质粒.利用 NanoDrop分光光度计测定质粒的质量浓度(单位为ng/μL)后,计算质粒的拷贝数,将该质粒按10的比例逐级稀释10个梯度,分装后-20℃保存.
内参基因16S rDNA采用E.coli DH5α构建标准质粒,构建方法参考 ndo基因.扩增引物为968f和 1401r[13].PCR 体系:25μL r-Taq,模板DNA1µL, 引物各1µL,ddH2O 22µL.PCR程序:95℃预变性 5min,94℃变性1min,56℃退火 45s,72℃延伸 1min,30个循环,最后72℃延伸10min.将含16S rDNA基因片段的克隆子3个送出测序.测序结果与E.coli DH5α菌株的16S rRNA基因的对应位置序列一致即可用做标准质粒的构建.
1.7 目标基因ndo拷贝数的检测
提取和纯化:采用TRIZOL方法提取总RNA.参考说明书,采用DNaseI(TAKARA)去除总RNA中的基因组DNA.纯化后的总RNA同时采用1% Agarose电泳(120V,30min)和Nanodrop检测.
反转录和检测:反转录后的 RNA,采用real-time PCR在同一批次中测定同一样品中的ndo和16S rRNA基因的绝对拷贝数.同时设空白对照并做标准曲线.目标基因 ndo的 Real-time PCR的反应体系为:SYBR®Premix Ex TaqTM10μL,上下游引物各0.6μL,反转录的cDNA 1μL,双蒸水 7.8μL.反应程序如下:1个循环:95℃, 30s;40 个循环:95℃,1min,47℃,1min,72℃, 2min(收集荧光);熔解曲线的过程为:95℃,1min,从60℃开始每30s温度升高0.5℃,总共进行71个循环,结束温度为95℃,反应结束之后4℃保存.内参基因16S rRNA Real-time PCR的反应体系为:SYBR®Premix Ex TaqTM10μL,上下游引物各0.4μL,反 转 录 的 cDNA 1μL,双 蒸 水8.2μL.Real-time PCR 反应程序如下:1个循环:95℃,30s;50个循环:95℃,30s,56℃,30s,72℃, 30s(收集荧光);熔解曲线的过程同ndo基因.参考Praffl[14]的方法,用同一样品中的 ndo和 16S rRNA绝对拷贝数的比值作为ndo的相对表达量.采用方差分析real-time PCR结果的差异性(n=3,P=0.05), Student-Newman-Keuls (SNK)法用于检验一组数据间的差异.方差分析和SNK检验均采用SPSS软件.
2 结果与分析
2.1 生长曲线和降解曲线
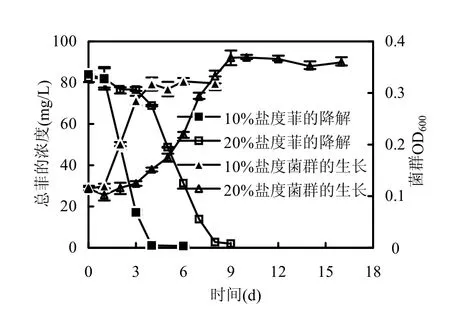
图1 初始菲浓度100mg/L,不同盐度下菲的降解和菌群的生长曲线Fig.1 The biodegradation and growth curve of the bacterial consortium growing with 100mg/L phenanthrene at different salinity
本文从胜利油田富集了一个中度嗜盐菌群,16S rRNA PCR-DGGE技术解析结果表明优势菌属与Halomonas, Alcanivorax, Marinobacter , Idiomarina和 Martelella 这些嗜盐菌属相似度最高(>96%),另外还有不可培养细菌(数据未发表).图1表明,该菌群的生长过程包括迟滞期、对数期和稳定期.在菲初始浓度为 100mg/L,10%盐度下,经过1d的迟滞期,菌群进入对数生长期,到第4d菌群进入生长稳定期.在20%盐度下,迟滞期延长到3d,菌群在第4d进入对数生长期,第9d进入稳定期.盐度从10%增加到20%,对数期间菌群的生长速率由 0.051d-1(R2=0.978)降低到0.028d-1(R2=0.972),菌群的生物量分别增加了0.198和0.207.
在迟滞期,菲的浓度略有下降,这可能是由微生物细胞对菲的降解或细胞膜对菲的吸附引起的[15].在对数期,10%和 20%盐度下,菲的降解动力学呈线性关系,动力学方程分别是 y=-27.39x+ 105.8(R2=0.978)和 y=-13.96x+127.0 (R2=0.965),平均降解速率分别是 27.39,13.96mg/(L·d).菲分别在6d内被降解了98.9%,9d内被降解了97.6%.表明盐度从10%增加到20%,中度嗜盐菌群需要更长时间适应环境,降解速率和生长速率均受到抑制.
2.2 溶解菲浓度的变化
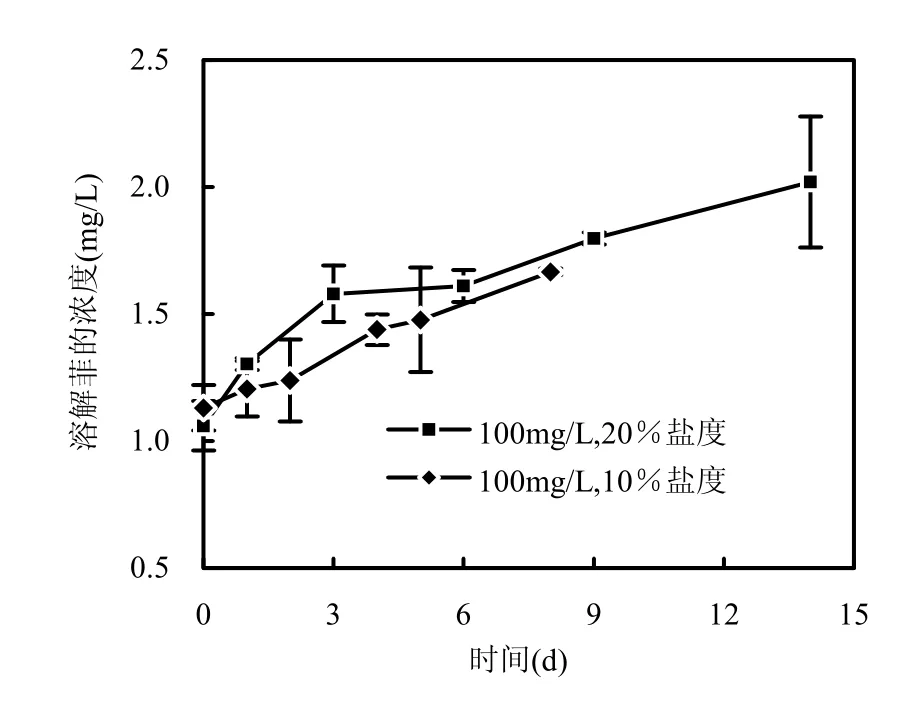
图2 10%和20%盐度下菌群降解100mg/L菲过程中溶解菲浓度的变化曲线Fig.2 The evolution of dissoluble phenanthrene concentration during biodegration at 10% and 20% salinity
由图2可见,盐度由10%升高到20%,初始溶解菲的浓度由1.13mg/L降低到1.06mg/L.这是因为随着盐度升高,菲的溶解度降低.在菌群生长的迟滞期和对数期,初始溶解菲的浓度在10%盐度和20%盐度下分别增加了30%和49%,且溶解菲的动态变化均符合一级动力学,拟合方程为y = 0.235ln(x+1)+1.064(R2= 0.924)和y=0.330ln (x+1) +1.064 (R2=0.970).Reddy等[9]研究短芽孢杆菌PDM-3对菲的降解,发现菌株在降解过程中产生糖脂类的表面活性剂,通过结合分析上清液的表面张力、乳化指数EI24和菲的降解过程,推测所研究的菌株在利用水溶性的菲的同时向细胞外分泌了表面活性剂,而表面活性剂进一步促进菲的溶解,提高菲的溶解度,进而加强菲的生物可利用度.同样,本研究中溶解菲的浓度在菌群生长过程中呈缓慢增加的趋势,但单因素方差分析(n=3,P>0.05)表明对数期溶解菲浓度在10%和20%盐度下没有显著差异,表明一定范围的盐度不会影响菌群降解过程中菲的生物可利用度.
2.3 Ndo基因多样
Ndo(萘双加氧酶基因)是编码催化PAHs降解起始步骤的多环芳烃双氧酶基因[16],它的种类和活性直接影响生物降解的速率.因此,本文构建了中度嗜盐菌群的 ndo基因文库,并进一步研究了菌群ndo基因的多样性以及降解过程中盐度对其表达的影响.在测序的71个含有ndo基因的克隆子中,序列比对表明仅有 1个克隆子与 GenBank中等位基因相似度低于 50%,可能由扩增错误引起.按照1个氨基酸序列的差异将70个克隆子分为6个OUT(代表1个基因型),克隆文库的覆盖率达到 90%.每个基因型挑选一个氨基酸序列构建系统发育树(图3).从图3可以看出,该中度嗜盐菌群中ndo基因均为nah-like基因.其中,序列A,B和 C与菌株 Pseudomonas aeruginosa Pak1和Pseudomonas stutzeri AN10中的经典nah-like基因最相似,相似度达到89%.A,B和C之间的两两相似度高于97.3%.序列A,B和C为主要基因型,占总克隆子 92.7%,并且单独聚为一类,表明 A,B和 C有可能是新的萘双加氧酶基因.一些海洋菌中 ndo基因的同源性分析也表明其与菌株Pseudomonas stutzeri AN10和Pseudomonas sp. C18中nah-like基因最相似[17-18].
2.4 降解过程中ndo基因的表达规律
本研究表明菌群中主要基因型序列 A、B和C的相似度大于97.3%,为经典nah-like基因(丰度超过92.7%),因此,本研究以该ndo基因为目标基因,利用Real-time PCR实时监测该基因在降解过程中表达量的变化.由图 4可以看出,在10%和20%盐度下,相对菌群初始基因表达水平,ndo的表达量均在迟滞期降低1个数量级,在对数期增加1个数量级,到稳定期时增加了约2个数量级.Pearson相关性分析表明 ndo基因表达量与溶解菲的浓度正相关(P=0.05,双边检验,R2=0.712).可见,高浓度菲在初始阶段可能抑制ndo基因的表达,经过一段迟滞期后,ndo基因的表达量会被溶解菲诱导而逐渐升高.同时,在对数期ndo基因表达量与总菲浓度呈负线性相关(图 5),这说明,在总菲浓度远远超过菲溶解度的条件下,即使是固态菲也可能对菌群功能基因的表达产生抑制作用.在相同生长时间,菌群ndo的表达量在10%盐度下比20%盐度下高,表明盐度升高同样可能抑制功能基因的表达,降解速率的测定结果也表明了高盐度对降解有明显抑制作用.即使在对数期,ndo表达量与降解速率也不存在相关性,可能是由于功能基因的表达量只能精确指示翻译过程中操纵子的起始转录水平,不会必然反映降解酶的活性和数量[19];另一方面,降解酶的活性和数量也可能只是影响生物降解速率的一个主要因素,同时还可能存在其他影响因素.

图3 根据菌群ndo基因克隆文库的70个氨基酸序列(110残基)构建的系统发育树Fig.3 Phylogenetic tree based on the analysis of the 70 deduced amino acid sequences (110 residues) of the amplified ndo gene fragments in the clone libraries
3 讨论
本研究中,在嗜盐菌群降解菲的过程中菌群ndo基因的绝对拷贝数与16S rRNA基因绝对拷贝数的比值保持在10-6和10-4之间波动.Cebron等[12]研究土壤和沉积物中 PAH-环羟基化双加氧酶基因(PAH-RHDα)的扩增和定量的特异性和准确性,在基因组水平上,PAH-RHDα与 16S rRNA基因的比值在PAHs污染水平高的样品中波动范围是1×10-5到9.22×10-3,比无PAHs污染的样品至少高 1个数量级[12].采用 2-ΔΔCT方法计算添加蚯蚓粪的PAHs污染土壤中ndo相对表达量,结果表明经过1个月的孵化后ndo的拷贝数增加了2~3个数量级[20].虽然由于分析方法和样品不同,功能基因的相对表达量并不适合与其他研究比较.但是,这些数据的大小和波动范围与本文检测结果一致.
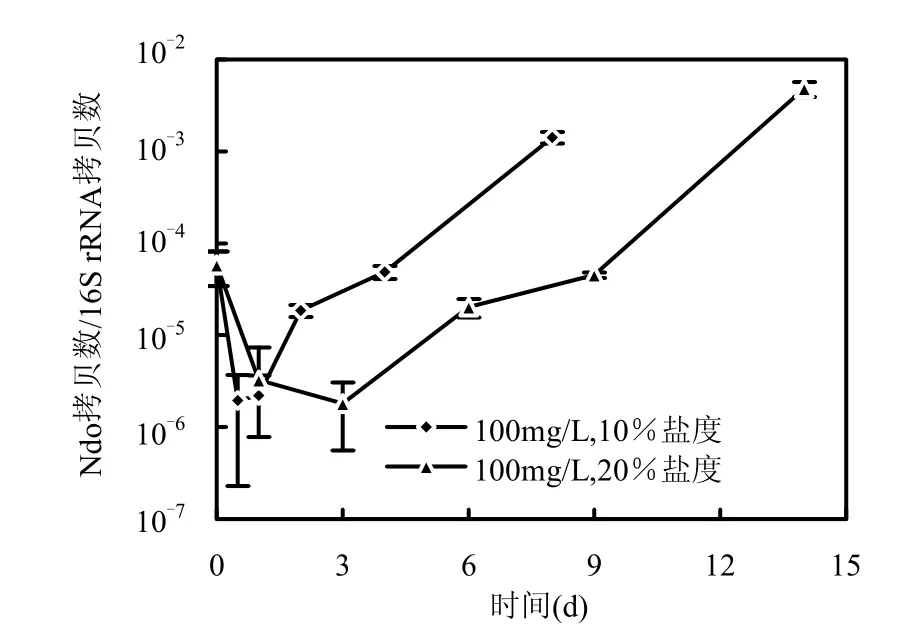
图4 10%和20%盐度下,100mg/L菲降解过程中菌群ndo的表达Fig.4 The expression of ndo genes in the bacterial consortiums growing at 10% and 20% salinity with 100mg/L phenanthrene
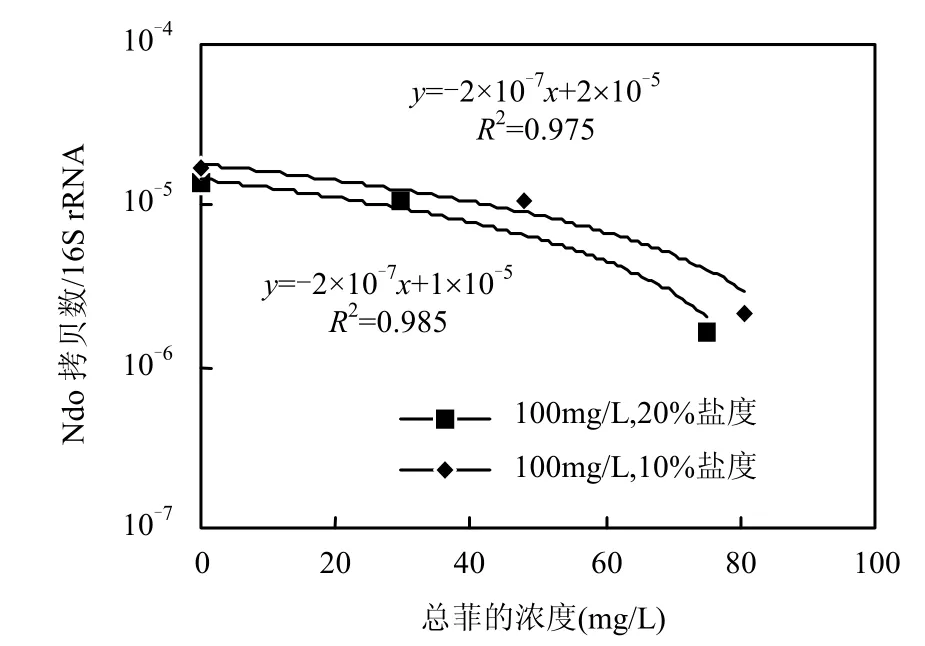
图5 对数生长期内菌群ndo基因的表达量与总菲浓度的关系Fig.5 The correlations between total phenanthrene concentration and ndo gene expression in the bacterial consortium growing at 10% and 20% salinity with 100mg/L phenanthrene at logarithmic phase
基因的表达量与微生物生理状态、外界环境、有毒中间代谢产物的积累、基因调控方式等有着密切而复杂的联系.Corbella等[19]分析了 2株 纯 菌 铜 绿 假 单 胞 菌 株 (Pseudomonas aeruginosa) 142和JB2的上、下游关键降解基因ohbA和hybA分别在迟滞期末期和稳定期基因表达量的比值,结果发现2个基因表达量在不同生长阶段的平均比值分别为 1.17±0.9和 0.07± 0.02.Marlowe等[21]则发现表面活性剂可以影响菲的降解速率和萘双加氧酶基因(nahAc)的表达量,在有表面活性剂时nahAc表达呈双峰模式.从这些研究可以看出,降解基因的调控是精致的,不同菌属的不同功能的基因表达规律不同,并同时受很多环境因素的影响.本研究发现混合菌群的降解基因表达量的变化趋势与 Corbella等[19]在纯菌中的观察到的结果相似,都是在迟滞期降低,对数期升高,稳定期持续升高,说明当环境因素变化时,降解微生物会从最初的受抑制到适应再到响应(如通过改变降解酶表达量及调控模式)形成应付环境变化的生存能力,这可能是一种环境(以混合菌群为主)中较为普遍的现象.
在盐度和污染物浓度的影响面,Minai-Tehrani等[22]在研究0~5%NaCl盐度对PAHs降解的影响时,发现盐度增加会降低土壤中石油和PAHs的降解速率.Zhao等[8]则发现在 10%盐度下从石油土壤中富集的中度嗜盐菌群在 20%盐度下基本丧失了降解菲的能力.本研究的实验结果更为明确地表明了高浓度污染物和盐度均可能抑制ndo基因的表达和生物降解速率.一般认为盐度对生物降解可能有双重影响:一方面,盐度可以通过渗透压等因素影响微生物的生理活性;另一方面,盐度会降低污染物的可溶解性从而影响污染物的生物可利用度.本研究发现,在一定的范围内,盐度的增加确实降低了菌群生物降解活性,但对污染物的生物可利用度并没有显著的影响,反而发现固态的污染物对降解酶基因的表达有抑制作用.Di Gennaro等[20]发现萘污染土壤中低污染水平(10μg/g)比高污染(100μg/g)下ndo基因表达量高,这与本研究发现在超过污染物饱和溶解度情况下,关键降解酶基因ndo的表达量与总菲浓度负相关的结果一致.当盐度从 10%升高到20%,ndo的表达量降低了.但10%和20%盐度下溶解菲的浓度并没有显著差异,说明盐度可能是通过影响其它因素而不是诱导底物的浓度抑制ndo基因的表达.因此,功能基因ndo的表达调控可能受底物的诱导和其它因素的共同作用.此外,普遍认为关键降解酶基因表达量能够反映生物降解本质,并能指示生物降解效率和潜能.但是在本研究条件下,关键功能基因的表达量与生物降解速率没有明显的相关性.因此,在复杂环境条件下以及复合微生物菌群中,如何使用功能基因表达量来反映生物降解的本质,指示生物降解的效率还需要进一步探讨,而盐度对生物降解的影响机制仍需要深入研究.
4 结论
4.1 从石油烃污染盐碱土壤中富集出一个中度嗜盐菌群,该菌群有6种ndo基因型,均与nah-like基因最相似.其中 3种主要基因型(占总克隆子92.7%)单独聚为一类,与菌株 Pseudomonas aeruginosa Pak1和Pseudomonas stutzeri AN10中的经典nah-like基因最相似,相似度达到89%.
4.2 溶解菲的浓度在菌群生长过程中呈增加的趋势.单因素方差分析(n=3,P>0.05)表明溶解菲浓度在10%和20%盐度下没有显著差异,表明一定范围的盐度不会影响菌群降解过程中菲的溶解度.Ndo基因的表达量在降解过程中呈先降低后随溶解菲浓度升高而升高的趋势,表明高浓度菲在初始阶段可能抑制ndo基因的表达,经过一段迟滞期后,ndo基因的表达量会被溶解菲诱导而逐渐升高.盐度由10%升高到20%,ndo基因表达量降低,但溶解菲浓度没有显著差异,说明盐度可能通过影响其它因素而不是诱导底物的浓度抑制ndo基因表达.
4.3 在 10%和 20%盐度下,嗜盐菌群分别在 6d内和9d内降解了97.6%菲,相应的菌群生长的迟滞期由 1d延长为 3d,表明盐度升高会抑制菌群的生长速率和菌群对菲的降解速率.
[1] Santodonato J. Review of the estrogenic and antiestrogenic activity of polycyclic aromatic hydrocarbons: relationship to carcinogenicity [J]. Chemosphere, 1997,34(4):835-848.
[2] Diaz M P, Boyd K G, Grigon S J, et al. Biodegradation of crude oil across a wide range of salinities by an extremely halotolerant bacterial consortium MPD-M, immobilized onto polypropylene fibers [J]. Biotechnology and Bioengineering, 2002,79(2):145-153.
[3] Kleinsteuber S, Riis V, Fetzer I, et al. Population dynamics within a microbial consortium during growth on diesel fuel in saline environments [J]. Applied and Environmental Microbiology, 2006,72(5):3531-3542.
[4] Margesin R, Schinner F. Biodegradation and bioremediation of hydrocarbons in extreme environments [J]. Applied Microbiology and Biotechnology, 2001,56(5/6):650-663.
[5] Riis V, Kleinsteuber S, Babel W. Influence of high salinities on the degradation of diesel fuel by bacterial consortia [J]. Canadian Journal of Microbiology, 2003,49(11):713-721.
[6] Nicholson C A, Fathepure B Z. Aerobic biodegradation of benzene and toluene under hypersaline conditions at the Great Salt Plains, Oklahoma [J]. FEMS Microbiology Letters, 2005, 245(2):257-262.
[7] Tremblay L, Kohl S D, Rice J A, et al. Effects of temperature, salinity, and dissolved humic substances on the sorption of polycyclic aromatic hydrocarbons to estuarine particles [J]. Marine Chemistry, 2005,96(12):21-34.
[8] Zhao B, Wang H, Mao X, et al. Biodegradation of phenanthrene by a halophilic bacterial consortium under aerobic conditions [J]. Current Microbiology, 2009,58(3):205-210.
[9] Reddy M S, Naresh B, Leela T, et al. Biodegradation of phenanthrene with biosurfactant production by a new strain of Brevibacillus sp. [J]. Bioresource Technology, 2010,101: 7980-7983
[10] Story S P, Kline E L, Hughes T A, et al. Degradation of aromatic hydrocarbons by Sphingomonas paucimobilis strain EPA505 [J]. Archives of Environmental Contamination and Toxicology, 2004,47(2):168-176.
[11] Baldwin B R, Nakatsu C H, Nies L. Detection and enumeration of aromatic oxygenase genes by multiplex and real-time PCR [J]. Applied and Environmental Microbiology, 2003,69(6):3350-3358.
[12] Cebron A, Norini M P, Beguiristain T, et al. Real-Time PCR quantification of PAH-ring hydroxylating dioxygenase (PAH-RHDalpha) genes from Gram positive and Gram negative bacteria in soil and sediment samples [J]. Journal of Microbiological Methods, 2008,73(2):148-159.
[13] Felske A, Akkermans A D L, DE VOS W M. Quantification of 16S rRNAs in complex bacterial communities by multiple competitive reverse transcription PCR in temperature gradient gel electrophoresis fingerprints [J]. Applied and Environmental Microbiology, 1998,64(11):4581-4587.
[14] Pfaffl M W. A new mathematical model for relative quantification in real-time RT-PCR [J]. Nucleic Acids Research, 2001,29(9): 2002-2007.
[15] Tian L, Ma P, Zhong J J. Kinetics and key enzyme activities of phenanthrene degradation by Pseudomonas mendocina [J]. Process Biochemistry, 2002,37(12):1431-1437.
[16] Habe H, Omori T. Genetics of polycyclic aromatic hydrocarbon metabolism in diverse aerobic bacteria [J]. Bioscience, Biotechnology, and Biochemistry, 2003,67(2):225-243.
[17] Ferrero M, Llobet-brossa E, Lalucat J, et al. Coexistence of two distinct copies of naphthalene degradation genes in Pseudomonas strains isolated from the western Mediterranean region [J]. Applied Environmental Microbiology, 2002,68(2):957-962.
[18] Hedlund B P, Geiselbrecht A D, Staley J T. Marinobacter strain NCE312 has a Pseudomonas-like naphthalene dioxygenase [J]. FEMS Microbiology Letters, 2001,201(1):47-51.
[19] Corbella M E, Puyet A. Real-time reverse transcription-PCR analysis of expression of halobenzoate and salicylate catabolism-associated operons in two strains of Pseudomonas aeruginosa [J]. Applied Environmental Microbiology, 2003,69(4): 2269-2275.
[20] Di Gennaro P, Moreno B, Annoni E, et al. Dynamic changes in bacterial community structure and in naphthalene dioxygenase expression in vermicompost-amended PAH-contaminated soils [J]. Journal of Hazardous Materiais, 2009,172(23):1464-1469.
[21] Marlowe E M, Wang J M, Pepper I L, et al. Application of a reverse transcription-PCR assay to monitor regulation of the catabolic nahAc gene during phenanthrene degradation [J]. Biodegradation, 2002,13(4):251-260.
[22] Minai-tehrani D, Minoui S, Herfatmanesh A. Effect of salinity on biodegradation of polycyclic aromatic hydrocarbons (PAHs) of heavy crude oil in soil [J]. Bulletin of Environmental Contamination and Toxicology, 2009,82(2):179-184.
Phenanthrene biodegradation and dynamic change of expression of naphthalene dioxygenase (ndo) genes in a halophilic bacteria consortium.
HE Fen1, WANG Li-hua2, NING Da-liang1, GUO Guang1, WANG Hui1*(1.School of Environment, Tsinghua University, Beijing 100084, China;2.Institute of Applied Chemistry, Hebei North University, Zhangjiakou 075000, China). China Environmental Science, 2012,32(9):1662~1669
A halophilic bacterial consortium capable of degrading phenanthrene was developed from Shengli Oilfield in China. The diversity of naphthalene dioxygenase (ndo) genes in the bacterial consortium was analyzed by the method of clone library. Consortium consist of six nah-like genes. Three of them (covered 92.7% of the total clones) related to the classic nah-like genes with identity of about 89%. Using real-time reverse transcription PCR and other techniques, this study investigated the dynamic expression of ndo genes, biodegradation rate, biomass and bioavailability of phenanthrene in bacterial consortium growing at different salinities. As the salinity increased from 10% to 20%, the lag phase increased from 1 day to 3 days, the time needed for degrading 100mg/L phenanthrene increased from 6 days to 9 days. Both of the dissolved phenanthrene concentrations at 10% and 20% salinity increased constantly during the biodegradation, and One-way ANOVA(n=3, P>0.05) analysis showed there were no significant difference between them. A certain range of salinity probably not affected the bioavailability of phenanthrene. The expression of ndo genes at 20% salinity was lower than that at 10% salinity, while both of them decreased at the lag phase, and then increased constantly. Increasing salinity and high concentration of phenanthrene inhibited the expression of ndo genes.
halophilic bacteria consortium;gene expression;bioavailability;solubility of phenanthrene;naphthalene dioxygenase gene
2012-02-18
国家自然科学基金资助项目(30970098)
* 责任作者, 教授, wanghui@mail.tsinghua.edu.cn
X172
A
1000-6923(2012)09-1662-08
何 芬(1987-),女,湖北武汉人,清华大学环境学院硕士研究生,研究方向为生物修复.发表论文3篇.
