30份野牛草种质的遗传多样性
2012-12-24成凯凯魏小兰张蕴薇
成凯凯,魏小兰,杜 鹃,张蕴薇
(中国农业大学草地研究所,北京100193)
野牛草(Buchloedactyloides)为多年生暖季型C4草本植物,是生长在北美草原上最古老的禾本科植物之一,起源于美洲中南部[1],自然群落主要分布在美国、加拿大、墨西哥中部的干旱地带[2],常与格兰马草(Boutelouagracilis)等一起构成草原景观[3]。因其具有很多优良特性及较好的坪用性状(如耐旱、植株低矮、叶片纤细柔软等)而逐渐被用于草坪种植,又因具有优越的抗病虫能力、抗旱性及适应性,日益成为我国华北、东北、西北等地区园林绿化、环境保护、水土保持的主要草种之一。
简单重复序列区间扩增(Inter-Simple Sequence Repeat,ISSR)是近年来一种以微卫星为引物的新型PCR分子标记技术[4]。目前常用的ISSR引物是由加拿大哥伦比亚大学(University of British Columbia,UBC)设计的96个ISSR 引物[5]。近年来ISSR技术已在品种鉴定[6-7]、遗传作图[8-9]和居群遗传学[10]等研究领域中被广泛应用。
本研究通过对30份不同地理来源的野牛草种质资源坪用性状的测定以及ISSR分子标记,分析供试材料的遗传多样性,检测材料的遗传结构及亲缘关系,通过聚类分析初步将种质归类,选择性状优良且亲缘关系较远的种质作为将来育种的材料,同时为野牛草种质资源的保护及利用提供依据。
1 材料与方法
1.1 供试材料 本试验的30份野牛草种质搜集于美国和中国,地理来源不同,2007年移栽至中国农业大学上庄试验站。地理位置115°50′E,40°02′N,海拔50m。暖温带大陆性季风气候,年平均气温11.4℃,绝对最低温-21.7℃,绝对最高温为41.6℃,年平均降水量628mm。全年无霜期211d。由于特殊原因,具体地理来源已不明。
1.2 田间观测 于2010年7月-2011年7月进行田间观测。从开花期开始测定的形态学指标有叶长、叶宽、株高、茎粗、匍匐茎长度、匍匐茎数目和匍匐茎节数;成熟期测定的生理指标包括叶色和叶片枯黄程度。其中选中的叶片在单株中用线和标牌分别进行标记,以便进行连续观测。
叶长:开花期,随机选取任意分蘖枝下端第2片叶片用直尺测定其长度,重复3次。
叶宽:开花期,用游标卡尺测定以上选取的叶片中部的宽度,重复3次。
株高:用直尺测定野牛草的绝对高度,从开花期开始连续测定3次,每次间隔10d。
茎粗:开花期,随机选取任意分蘖枝,用游标卡尺测定其茎基部的直径,同一植株3个重复。
分蘖数:在分蘖期、开花期和结实期各测定1次。
匍匐茎长度:用直尺测定,从开花期开始连续测定3次,每次间隔10d。
匍匐茎节数:统计测定的最长匍匐茎的节数,从开花期开始连续统计3次,每次间隔10d。
匍匐茎数目:从开花期开始连续测定3次,每次间隔10d。
叶色:在野牛草成熟期(7和8月),每株随机选取任3个叶片采用叶绿素测定仪(SPAD仪)测定SPAD值,并对选定叶片做记号,重复3次。
叶片枯黄程度:2010年10月4日、10月14日和10月24日每个单株标记3个叶片,分别测定叶片总长度和枯黄部分的长度,以枯黄部分占叶片总长度的比值表示叶片枯黄程度。
1.3 野牛草基因组DNA的提取 选取野牛草健康幼叶,用CTAB法[11]提取其基因组DNA,通过琼脂糖凝胶电泳和紫外分光光度计检测其浓度和纯度,合格的DNA样品于4℃冰箱内保存备用。
1.4 ISSR-PCR体系的建立及优化 根据之前发表的ISSR-PCR反应体系[12-17],经优化后得到下述体系:总体积25μL,其中1×buffer 1.5μL、dNTP 0.3mmol·L-1、Taq酶1.0U、Primers 0.6 μmol·L-1、Mg2+1.0mmol·L-1、DNA 模板50 ng,双蒸水补足25μL。
1.5 引物筛选和PCR反应 本试验中ISSR反应采用的引物序列均由加拿大哥伦比亚大学提供,由上海生物工程公司合成,共33条[18]。
PCR扩增程序:94℃预变性3min;94℃变性30s,48~60℃退火30s,72℃延伸1min,共35个循环;最后72℃延伸10min,4℃保存[12]。
1.6 电泳检测 本试验采用的是12%的聚丙烯酰胺凝胶电泳,以盛旭百川生物科技公司生产的DL2000为Marker,110V电压下电泳4h,停止电泳后在0.1%硝酸银溶液中银染,NaOH溶液中显影,然后拍照,供数据分析。
1.7 数据分析 田间试验数据采用SAS 8.2进行差异显著性分析和聚类分析。
分子试验数据分析方面,对获得的清晰可重复的DNA条带进行统计,在相同迁移位置上,根据条带的有无记为1或0,构成原始数据矩阵。
根据原始矩阵,统计得到的总条带数和多态性条带数,通过计算多态性位点百分率(Percentage of Polymorphic Bands,PPB),引物多态性含量(Polymorphism Information Content,PIC)和遗传相似系数(GS)来估计遗传多样性。

式中,TNB指ISSR扩增的总条带数(Total Number of Bands),NPB指多态性条带数(Number of Polymorphic Bands);PIC=1-∑Pi2,Pi为i位点的基因频率。

式中,Nij为材料i和j共有的扩增条带数,Ni为材料i的扩增条带数,Nj为材料j的扩增条带数;遗传距离GD=1-GS。利用NTSYS-PC 2.10软件计算Dice遗传相似系数,根据GS值按不加权成对群算术平均法(UPGMA)对野牛草种质材料进行遗传相似性聚类和主成分分析。
2 结果与分析
2.1 不同地理来源野牛草的坪用性状观测30份不同地理来源的野牛草单株相互之间在基本坪用性状上存在很大差异。叶长、叶宽、株高、匍匐茎长度和匍匐茎节数差异极显著(P<0.01);茎粗、叶色呈显著差异(P<0.05);分蘖数、匍匐茎数目和叶片枯黄程度无显著差异(P>0.05)(表1)。
根据形态学指标可将30份不同地理来源野牛草分为4类,第Ⅰ类材料有16份,包括2、3、4、5、7、8、12、14、16、17、19、21、22、25、28、29;第 Ⅱ 类 材料7份,包括9、10、11、23、24、26、27;第Ⅲ类材料3份,包括1、6、30;第Ⅳ类材料4份,包括13、15、18、20(图1)。

表1 不同地理来源野牛草坪用性状观测Table 1 Observation of turf characteristics of 30different geographical origin Buchloe dactyloides
2.2 供试材料ISSR扩增产物的多态性分析从33条引物中选择了7条[18]扩增条带多态性好、清晰稳定的引物对30份材料进行PCR扩增,通过聚丙烯酰胺凝胶电泳(PAGE)的检测,部分结果见图2。
30份野牛草材料经过7个引物的扩增共获得108条条带,其中多态性条带数目为90条,多态性比率为83.3%,平均每个引物扩增出条带15.4条,PIC值的变化范围为0.78~0.89,平均值是0.85(表2)。所选用的7条引物基本上均能独立地将30份野牛草区分开来,这说明不同地理来源的供试野牛草间具有较大的遗传变异和较丰富的遗传多态性,同时表明了ISSR分子标记可以用来检测野牛草属种间亲缘关系。
2.3 野牛草种质的亲缘关系分析 遗传相似系数和遗传距离是评价群体间、群体内DNA变异水平的重要参数。遗传相似系数越大,说明群体间的亲缘关系越近;遗传相似系数越小,说明群体间的亲缘关系越远。
本试验利用筛选的7条ISSR引物对30份不同地理来源的野牛草基因组DNA进行PCR扩增,共扩出108个位点,通过NTSYS-PC 2.10进行遗传相似系数分析。不同地理来源野牛草种质资源的GS值变化范围为0.546~0.880,其中材料22和23、材料30和37的遗传相似性系数最高,为0.880,遗传距离较近;材料3和材料39的遗传相似性系数最低,只有0.546,亲缘关系最远(表3)。
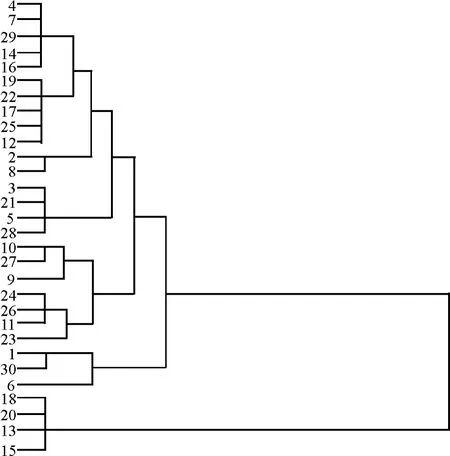
图1 30份不同地理来源野牛草资源的表型聚类图Fig.1 Phenotype UPGMA-derived dendrogram of 30 different geographical origin Buchloe dactyloides
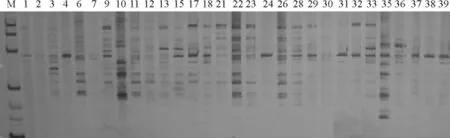
图2 引物TP2聚丙烯酰胺凝胶电泳图谱Fig.2 Results of PCR amplification with primer TP2

表2 用于ISSR分子标记的引物以及扩增结果Table 2 The primers and the results of ISSR amplification
2.4 野牛草种质的聚类分析 利用ISSR分子标记在遗传相似系数为0.690时能将30份野牛草资源分为5类,第Ⅰ大类包括3份材料(1、6、10);第Ⅱ大类包括20份材料:在遗传相似系数为0.710时,第Ⅱ大类材料又分为4个亚类,第1亚类为材料2和材料9,其GS值为0.701,第2亚类共7份材料(3、26、31、32、33、36、38),第3亚类包括8份材料(11、12、13、15、23、24、29、35),第4亚类共3份材料(17、21、28);第Ⅲ大类包括3份材料(4、18、22);第Ⅳ大类包括3份材料(7、30、37);第Ⅴ大类仅为材料39。聚类分析直观地表明了不同野牛草材料之间的
2.5 野牛草材料的主成分分析 基于GS值,利用NTSYS-PC 2.10软件对不同地理来源的野牛草材料进行主成分分析,根据第一、第二主成分作图(图4),得到30份野牛草材料的位置分布情况,位置靠近者表示其具有较近的亲缘关系,位置远离者表示其关系较远。将位置靠近者划在一起,供试野牛草材料大致可以分为5组,即第1组包括材料7、30、37,第2组包括材料4、18、22,第3组包括材料1、3、26、31、32、33、36,第4组包括2、9、11、12、15、17、2 1、23,第5组包括6、10、13、24、28、29、35、38、39。主成分分析结果与聚类分析结果基本一致,并且主成分分析结果在表明不同野牛草材料之间的亲缘关系方面更加直观。

图3 30份材料基于遗传相似系数的UPGMA聚类图Fig.3 UPGMA dendrogram based on GS genetic similarity coefficients4

?
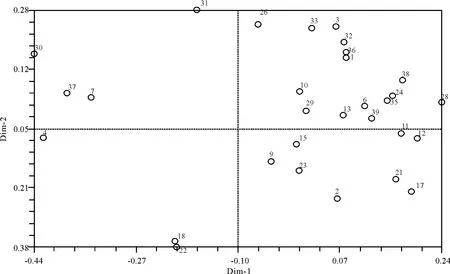
图4 30份野牛草材料基于遗传相似系数的主成分分析Fig.4 The principal coordinate analysis(PCA)based on genetic similarity coefficients
2.6 结合田间试验与分子试验的结果分析结合田间试验与分子试验的聚类结果,下列材料的两种聚类结果一致:材料2与材料9在田间试验中被聚为第Ⅰ大类,在分子试验中聚为第Ⅱ大类中的第一亚类;材料4、18和22在田间试验中聚为第Ⅰ大类,在分子试验中聚为第Ⅲ大类;材料11、12、13、31、32、35和36在田间试验中聚为第Ⅱ大类,在分子试验中31、32和36聚为第Ⅱ大类中的第二亚类,11、12、35和13聚为第Ⅱ大类中的第三亚类;材料1 7、21、24和28在田间试验中聚为第Ⅳ大类,在分子试验中聚为第Ⅱ大类中的第三亚类。
3 讨论
本研究中30份不同地理来源的野牛草单株之间在基本坪用性状上存在很大差异,其中叶长、叶宽、茎粗、株高、匍匐茎长度和匍匐茎节数差异极显著(P<0.01),叶色呈现显著差异(P<0.05),分蘖数、匍匐茎数目和叶片枯黄程度没有显著差异。形态学指标的差异显著性分析结果说明,供试材料间具有丰富的遗传背景,表型多样性较好,根据形态学指标能将所有材料聚为4类,这为初步推断其来源地是否相同提供了直观的信息,同时为选择性状优良植株进行育种提供佐证。
分子试验方面,从ISSR标记的结果来看,一共得到了108条条带,90条为多态性条带,平均多态性信息含量为0.850,说明这30份材料具有较丰富的多态性,同时也说明了ISSR分子标记技术具有高效、遗传多态性高、重复性好等特点,同时也克服了RAPD重复性低、AFLP成本高和SSR需要测定重复序列两端的序列信息等缺点,表明ISSR分子标记技术对于分析野牛草的遗传多样性方面是有效的,可以用作野牛草育种的辅助工具,同时本研究的结论为ISSR分子标记技术在野牛草属植物上进一步研究中的应用奠定了一定的基础,类似的结果也出现在其他植物的研究中[13,19-24]。
从之前的研究中发现,ISSR标记聚类群体与其地理位置的分布有一定的关系,地理位置接近的多数材料都聚成一类[25-26]。遗传距离从一定程度上反映出群体间遗传差异程度。一般认为材料间的遗传距离越大,它们产生的杂种优势就有可能越大,但是对于不同作物和牧草,遗传距离与杂种优势的关系不同且较为复杂。本研究利用ISSR标记对30份不同地理来源野牛草的遗传多样性聚类分析,遗传相似性系数变化范围为0.546~0.880,其中材料22和23、材料30和37的遗传相似性系数最高,为0.880,遗传距离较近;材料3和材料39的遗传相似性系数最低,只有0.546,亲缘关系最远。通过对遗传距离的分析可以得出,供试材料之间具有遗传基础广阔、遗传多样性丰富的特点。这在主成分分析结果中也得到了证实,主成分的结果和聚类结果大体一致,但是有少部分材料通过两种分析的结果不尽相同,大部分材料在空间图上所处的空间位置较远,这可能与野牛草复杂的倍性有关。Johnson等[27]等研究了美国野牛草主要分布区域的染色体水平状况,发现美国主要分布区的野牛草的染色体水平主要有二倍体、四倍体、五倍体及六倍体4种类型。其中,六倍体野牛草(73%)是自然分布的主体类型,在采集地均有分布,四倍体野牛草(23%)主要分布在美国的西部地区,二倍体野牛草(2.6%)的野生种群只分布在墨西哥东部地区和德克萨斯州西北部,在野外也发现了少量的五倍体(1.8%)。遗传距离分析及聚类分析的结果从分子水平进一步说明了供试种质资源间存在丰富的遗传变异。野牛草植物丰富的遗传变异为野牛草属植物的优良品系选育、开发和利用提供了空间,避免了因遗传基础狭窄而给育种工作造成的限制。因此,对野牛草属植物进行深入研究,选育优良的新品种具有资源方面的优势也具有开发潜力。
结合田间试验与分子试验的聚类结果,有些材料在两种聚类分析中都聚为一类,表现出了较好的一致性,这些材料地理来源很有可能相同,该结果为优良野牛草种质资源的筛选提供了可靠的依据,同时揭示了不同地理来源野牛草种质的遗传结构和亲缘关系,为野牛草种质资源的利用以及保护提供了依据。
[1]Quinn J A,Engel J L.Life-history strategies and sex ratios for a cultivar and wild population ofBuchloe dactyloides(Graminean)[J].American Journal of Bot-any,1986,73(6):874-881.
[2]Riordan T.An old grass is getting a new look[J].USGA Green Section Record,1991,29:6-9.
[3]胡林,边秀举,阳新玲.草坪科学与管理[M].北京:中国农业大学出版社,2001.
[4]周延清.DNA分子标记技术在植物研究中的应用[M].北京:化学工业出版社,2005:2-3.
[5]解新明,卢小良.SSR和ISSR标记及其在牧草遗传与育种研究中的应用前景[J].草业科学,2005,22(2):30-37.
[6]孟芳,袁庆华,苏德荣,等.紫花苜蓿抗褐斑病ISSR分子标记研究[J].草地学报,2007,15(6):561-565.
[7]张立荣,徐大庆,杨文香,等.小麦抗叶锈基因Lr37ISSR分子标记的初步研究[J].河南农业大学学报,2004,27(3):69-77.
[8]Pasqualone A,Lotti C,Bruno A,etal.Use of ISSR markers for cultivar identification in durum wheat[J].Ciheam-Options Mediterraneennes,2000(3):157-161.
[9]Dangi R S,Lagu M D,Choudhary L B,etal.Assessment of genetic diversity inTrigonellafoenugraecumandTrigonellacaeruleausing ISSR and RAPD markers[J].BMC Plant Bio1ogy,2004,4(1):13-24.
[10]Barcaccia G,Albertini E,Rosellini D,etal.Inheritance and mapping of 2n-egg production in diploid alfalfa[J].Genome,2000,43(3):528-537.
[11]Ausubel F M.精编分子生物学实验指南[M].金由辛,包慧中,赵丽云,译.北京:科学出版社,1998:58.
[12]Wei X L,Zhang Y W.Establishment and Optimization ofBuchloedactyloidesISSR-PCR Optimal Conditions[R].Buenos Aires,Argentina:The 6th Internationall Symposium on Mollecullar Breeding of Forage and Turf MBFT,2010.
[13]李永祥,李斯深,李立会.披碱草属12个物种遗传多样性的ISSR和SSR比较分析[J].中国农业科学,2005,38(8):1522-1527.
[14]Cupta M,Chyi Y S,Romero-Severson J,etal.Amplification of DNA maker from evolutionarily diverse genomesusing single primers of simplesequence repeats[J].TAG Theoretical Applied Genetics,1994,89:998-1006
[15]钱韦,葛颂,洪德元.采用RAPD和ISSR标记探讨中国疣粒野生稻的遗传多样性[J].植物学报,2000,42(7):741-750.
[16]Nagaoka T,Ogihara Y.Applicability of inter simple sequence repeat polymorphisms in wheat for use as DNA markers in comarison to RFLP and RAPD markers[J].TAG Theoretical Applied Genetics,1997,94:597-602.
[17]Joshi S P,Gupta V S,Aggarwal R K,etal.Genetic dicersity and phylogenetic relationship as revealed by inter simple sequence repeat(ISSR)polymorphiam in the genusOryza[J].Theoretical Applied Genetics TAG,2000,100:1311-1320.
[18]Zhang B Y,Wei X L,Yang F Y,etal.Effects of space flight factors on genetic diversity ofBuchloedactyloidesseeds[J].African Journal of Biotechnology,2011,10(60):12812-12820.
[19]Kostla S,Ruohonen-Lehto M,VäinöläR,etal.Phylogenetic information in inter-SINE and inter-SSR fingerprints of the Artiodactyla and evolution of the BovtA sine[J].Heredity,2000,84:37-45.
[20]汪爱华,丁毅.西藏近缘野生大麦RAPD和ISSR分子标记的遗传多样性[J].武汉大学学报,2007,53(6):723-730.
[21]侯永翠,颜泽洪,兰秀锦,等.利用RAMP和ISSR标记分析大麦种质资源的遗传多样性[J].中国农业科学,2005,38(12):2555-2565.
[22]程春明,石云素,宋燕春,等.ISSR分子标记技术在分析玉米自交系遗传关系研究中的适用性[J].植物遗传资源学报,2005,6(2):172-177.
[23]刘莉,邓春婷,包满珠.野牛草实生群体多样性的表型及ISSR分析[J].草业科学,2008,25(1):100-105.
[24]马艳明,李斯深,范玉顶,等.黄淮麦区小麦品种(系)的ISSR位点遗传多样性分析[J].植物遗传资源学报,2006,7(1):13-17.
[25]彭帅,武贤进,唐冬英,等.17份鱼腥草种质亲缘关系的ISSR分析[J].安徽农业科学,2007,35(12):3484-3486.
[26]Wilson B L,Kitzmiller J,Rolle W,etal.Isozyme variation and its environmental correlates inElymus glaucusfrom the California Floristic Province[J].Canadian Journal of Botany,2001,79:139-153.
[27]Johnson P G,Riordan T P,Johnson-Cicalese J.Lowmowing tolerance in buffalograss[J].Crop Science,2000,40:1339-1343.
