基于COI基因序列的太湖新银鱼遗传多样性*
2012-12-11雷光春王忠锁
张 迪,雷光春,龚 成,王忠锁
(1:北京林业大学自然保护区学院,北京100083)
(2:首都师范大学生命科学学院,北京100048)
(3:天鹅洲白暨豚国家级自然保护区,石首434400)
基于COI基因序列的太湖新银鱼遗传多样性*
张 迪1,2,雷光春1,龚 成3,王忠锁2**
(1:北京林业大学自然保护区学院,北京100083)
(2:首都师范大学生命科学学院,北京100048)
(3:天鹅洲白暨豚国家级自然保护区,石首434400)
利用线粒体细胞色素C氧化酶I(COI)分子标记分析长江中下游太湖新银鱼(Neosalanx taihuensis)8个地理种群132个样本的遗传多样性.该基因630 bp片段的碱基序列共检出8个核苷酸变异位点(变异率1.27%),其中局域性单倍型居多(75%),群体单倍型多样性较高(h=0.576 ±0.036),而核苷酸多样性较低(π =0.00112 ±0.00204).不同地理种群遗传多样性差异显著:有人工移植历史种群遗传多样性较高、隔离度较高的种群遗传多样性较低,但大部分的遗传变异来自于种群内(54.83%),反映出地理隔离和人为干扰对太湖新银鱼遗传格局影响显著.研究表明COI基因适于银鱼科鱼类物种鉴别和系统发育研究,同时可为同种种群间遗传关系分析提供一定的信息.
太湖新银鱼;长江;线粒体COI;遗传多样性;生命条码
太湖新银鱼(Neosalanx taihuensis Chen)是我国特有的淡水鱼类[1-2],隶属于胡瓜鱼目(Osmeriformes)银鱼科(Salangidae)[3].该物种属于一年生鱼类,对水环境变化较敏感[4],同时具有重要的经济价值.其地理分布区位于我国黄河及其以南的东部河流中下游与河口区[5].长江中下游及其附属湖泊是太湖新银鱼的核心分布区,是该物种资源保护的关键区域[6].
近30~50 a来,太湖新银鱼天然资源呈现大范围、长时间持续衰退趋势.1980s后,种群退化速度加快.现今,除长江和淮河中下游少数大中型湖泊外,其它绝大部分湖泊内的天然太湖新银鱼已经消失或失去经济价值[7-8].与此同时,大范围的太湖新银鱼人工移植改变了该物种的空间分布格局[9].新建于上游水体的移植种群会因个体入侵而对下游天然种群造成基因污染,影响物种遗传多样性空间结构的保护和进化演化信息的保存[7].因此,选择合适的分子标记研究太湖新银鱼天然种群遗传多样性格局为其种质资源保护的当务之急.
太湖新银鱼遗传多样性的研究已有较好的基础,同工酶[10]、RAPD[11]、AFLP[12]和线粒体 Cytb、COII[13-14]等分子标记相继用于其遗传多样性分析.Zhao等基于Cytb基因遗传多样性分析了包括长江流域在内的3个流域13个太湖新银鱼地理种群的遗传关系[15],郭立等以线粒体的4个常用基因分析了包括太湖新银鱼在内的银鱼科鱼类的系统发育关系[16].已有的研究报道为银鱼科鱼类系统发育关系的确定提供了重要信息,但对太湖新银鱼种群遗传结构和空间分布格局的探索尚显不足.
近年来,DNA条形码(DNA barcode)技术发展迅速[17].线粒体DNA的COI基因因其长度适宜、进化速率慢、富含系统发育信息以及易被通用引物所扩增等特点而被广泛用于条形码分析[17-20].基于COI基因655 bp碱基片段的分析,Ward等有效地区分了澳大利亚207种海洋鱼类[19],Schlei等对鲑科鱼类一个亚科的3个属进行鉴定[21],王中铎等有效鉴别了南海40种硬骨鱼类[22].郭立等研究中所涉及的太湖新银鱼COI基因多样性分析为本研究提供了重要启示[16].本研究选用COI基因标记,分析了长江中下游8个太湖新银鱼地理种群的遗传结构,目的在于:1)从种下水平探索COI分子标记在银鱼科鱼类中作为DNA条形码的可行性;2)分析太湖新银鱼遗传多样性分布格局,探索地理种群形成机制,并为制定银鱼资源保护对策提供科学依据.
1 材料和方法
1.1 研究区域及样点位置
共采集长江中下游天然银鱼分布区8个地理种群的太湖新银鱼,其中长江中游和下游各4个种群(图1).由于鄱阳湖南北狭长、水面宽阔,调查样点分散,图1中标出了主要样点的分布,但数据处理时将罗溪湾、金溪湖、珠湖、龙潭湖坝下及新妙湖样品归为鄱阳湖种群.共分析太湖新银鱼样本132尾,其中洞庭湖16尾,华容东湖16尾,天鹅洲故道4尾,梁子湖20尾,鄱阳湖28尾,泊湖16尾,巢湖16尾,南漪湖16尾.
1.2 COI序列分析
以专业银鱼拖网采集银鱼,所获银鱼用95%乙醇分类保存.DNA提取时,取乙醇保存的太湖新银鱼背鳍前方背部肌肉组织约 30 mg,置于 1.5 ml离心管中,用 500 μl匀浆缓冲液(1 mol/L Tris-HCl 25 μl,0.5 mol/L EDTA 50 μl,5 mol/L NaCl)浸泡过夜,以充分溶解去除组织中的乙醇.常规SDS蛋白酶K消化,酚、氯仿-异戊醇(24:1)各抽提1次,无水乙醇沉淀,70%酒精洗涤获得基因组DNA.

图1 研究区域和采样湖泊分布Fig.1 Study area and sampling sites of Yangtze River basin
扩增mtDNA COI基因5'端630 bp碱基序列,采用以下引物序列[19]:FishF1:5'-TCAACCAACCACAAAGACATTGGCAC-3';FishR1:5'-TAGACTTCTGGGTGGCCAAAGAATCA-3'.
PCR 反应体系 25 μl:10 × buffer 2.5 μl,dNTPs(2.5 mM)2 μl,FishF1、FishR1(10 μM)各 0.5 μl,Taq 酶(2.5U)0.2 μl,反应模版为 0.1 ~ 1.0 μg 基因组 DNA,加超纯水补至 25 μl.PCR 反应程序:95℃ 预变性2 min;循环40次,94℃变性30 s,54℃退火30 s,72℃延伸1 min;最后72℃延伸10 min.扩增产物经1%琼脂糖凝胶电泳检测后送博迈德生物有限技术公司测序,测序引物与扩增引物相同.序列比对使用Bioedit 7.0软件完成[23].
1.3 数据分析
使用Bioedit 7.0软件对COI基因序列进行编辑、排序及比对,利用Mega 4.0软件对序列的碱基组成、密码子各位置碱基组成、替换频率、转换颠换比进行计算[24].运用Dnasp 4.0软件对所有序列进行单倍型分类,统计各单倍型频率,同时分析8个群体的遗传多样性参数[25].在TCS 1.21中以最大简约法构建单倍型网络图[26].使用Arlequin 3.1软件中的分子方差分析(AMOVA)方法评价太湖新银鱼不同地理种群的遗传变异水平[27].在MEGA 4.0软件中,基于Kimura双参数模型计算各地理种群内及种群间的遗传距离.
2 结果与分析
2.1 太湖新银鱼群体COI基因序列多态性
太湖新银鱼132个样本COI基因630 bp碱基序列中 A、T、G、C的平均含量分别为20.2%、25.5%、19.8%、34.5%,其中 A+T(45.7%)比例略小于 G+C(54.3%).
序列比对结果显示COI基因630 bp序列共有8个变异位点,约占全序列的1.27%,其中7个变异位点发生在密码子第三位点(占可变位点数的87.5%),仅有一个发生在密码子第一位点(占可变位点数的12.5%).简约信息位点4个,碱基的转换/颠换比(Ts/Tv)为46.67,转换频率大于颠换频率(表1).

表1 太湖新银鱼mtDNA COI基因突变位点及单倍型Tab.1 Polymorphic locus and haplotype of mtDNA COI gene of N.taihuensis
2.2 单倍型分析
132尾太湖新银鱼样本共检出8个单倍型(表2),其中有5个共享单倍型,3个单倍型为单个地理种群所特有.TCS最大简约单倍型网络图显示HapI、HapII个体为较原始的单倍型,其他单倍型多由该单倍型经过一次或两次变异产生(图2).在8个单倍型中,HapI和HapII为明显的优势单倍型,占样本总数的86.36%.其中 HapI占59.09%(78 个样本)、HapII占27.27%(36 个样本,表2).
单倍型发生率和单倍型频率呈显著正相关(R=0.931,P=0.001).HapI和HapII发生率分别为87.50%和75.00%,表现为流域广布型.HapVI发生率为37.50%,HapV发生率为25.00%,其它单倍型仅见于单个种群,发生率为12.50%(图3).种群单倍型丰度(单倍型数)彼此差异悬殊,华容东湖和巢湖种群单倍型最丰富(5个单倍型),其次为鄱阳湖(3个单倍型),而梁子湖则没有单倍型分化(表2).

图2 太湖新银鱼单倍型网络关系Fig.2 Genetic relationships of the eight haplotypes
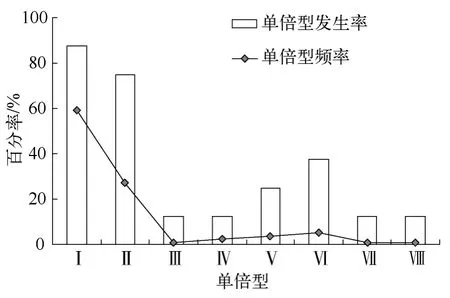
图3 单倍型频率及种群发生率Fig.3 Frequency and occurrence incidence of the COI haplotypes

表2 太湖新银鱼线粒体COI基因单倍型分布Tab.2 Distribution of the eight haplotypes of N.taihuensis mtDNA COI gene
2.3 种群遗传多样性分析
8个种群总的单倍型多样性(h)较高,介于0~0.667之间,均值为0.576±0.036.核苷酸多样性(π)较低,介于0.00037~0.03976之间,均值为0.00112±0.00204(表3).种群多样性系数最高的为巢湖和华容东湖,其次为天鹅洲故道和鄱阳湖,而相对较低的是洞庭湖和梁子湖种群.

表3 太湖新银鱼各种群的单倍型多样性和核苷酸多样性Tab.3 Haplotype diversity(h)and nucleotide diversity(π)of N.taihuenesis populations
2.4 种群遗传距离和AMOVA分析
长江中下游8个太湖新银鱼群体的遗传距离在0.00020~0.00265之间,其中华容东湖和巢湖遗传差异最大,达到0.00252;梁子湖和泊湖间遗传距离最小,仅为0.00020(表4).

表4 太湖新银鱼种群遗传距离Tab.4 Genetic distance among eight N.taihuenesis populations
AMOVA分析结果表明长江中下游太湖新银鱼之间有明显的遗传分化,但这种变异主要来源于种群内个体间,而各地理种群之间的分化不明显(P>0.05).这一结果与局域性单倍型丰富现象相一致(表5).

表5 太湖新银鱼mtDNA COI基因序列Ф-统计量AMOVA分析Tab.5 AMOVA on mtDNA COI gene of N.taihuensis based on Ф-statistics
3 讨论
经济鱼类捕捞产量下降是其资源衰退的直接表现,而资源衰退的深层次影响则反映在遗传多样性的损失和种质退化上.如果说渔业储量(fishery stock)可以通过人工增殖等手段来恢复的话,种群遗传多样性的损失却难以弥补,而长期进化形成的物种空间遗传结构的破坏则根本无法重建.因此,对退化物种种质状况的监测和遗传保护显得尤为重要,但这一点并未受到应有的重视,既有的研究基础也非常薄弱[6].太湖新银鱼为传统银鱼业的主要捕捞对象之一,具有较高的经济价值.长江流域是太湖新银鱼的传统产区,具有悠久的商业生产历史和最高的商业产量.但近30~50 a来,该物种持续衰退,产量持续下滑,局域经济绝灭频发,资源衰退极其严重[7].自1980s初起孙帼英等报道了长江口和长江中下游主要湖泊的银鱼资源衰退问题[28-30],2000s初王忠锁等从流域尺度上开展了天然银鱼资源的调查和研究,并系统报道和分析了长江流域淡水银鱼资源状况及其环境因子相关性[31].但这些工作基本停留在渔业产量和物种多样性水平,基本没有涉及银鱼的种质衰退问题.
太湖新银鱼活动能力较弱,基本属于静水定居性鱼类,易受堤坝涵闸的阻碍而形成地理隔离.已有研究发现长江流域的大量水工建筑导致的生境破碎和种群隔离对银鱼空间格局产生显著影响,是导致银鱼资源退化的重要因素[32].本研究进一步证实这些物理障碍是导致银鱼遗传结构发生变化的重要因素.长江流域太湖新银鱼8个遗传单倍型中局域性单倍型比例较高,占单倍型总数的75%,其中单种群独有单倍型达4个,同时隔离度较高的洞庭湖和梁子湖种群单倍型丰度较低.这些结果既印证了太湖新银鱼天然群体间存在较高的地理隔离,彼此间基因流不畅,又反映出局域种群特有遗传类型丰富的遗传多样性空间格局.这与Zhao等基于线粒体Cytb基因的分析结果相一致[15].因此,与其它鱼类相比太湖新银鱼局域种群绝灭所造成的遗传多样性损失应该更大.遗憾的是太湖新银鱼天然资源已经从长江、淮河以外的流域内消失,即便在江淮流域其残存的有效种群也不过10个[7].由此推断我国太湖新银鱼遗传资源已经遭受了严重损失,其空间遗传结构也在天然种群大量绝灭过程中遭到相当严重的破坏.
在天然种群衰退的同时,太湖新银鱼被人工移植到本无银鱼分布的数千个水库或湖泊[9].新建于江河上游水体的太湖新银鱼移植种群会因个体顺水下流而入侵下游的天然种群,并可能对天然种群遗传结构和遗传多样性格局构成威胁.本研究中有太湖新银鱼人工移植历史的巢湖和华容东湖两种群的COI单倍型达到5个,显著高于其它未受移植影响的种群的单倍型数(不超过3个,表1).由此推断,人工移植过程对天然太湖新银鱼种群的遗传结构确实产生一定的影响.Zhao等[15]对太湖新银鱼流域内种群间遗传距离高于流域间的报道应该在一定程度上与人工移植的影响有关,因为珠江流域的太湖新银鱼基本都是1980s后人工移植新建的,其遗传结构更类似于源种群(source population),不具备珠江流域的地理区域代表性.由于案例较少,本文关于移植种群影响太湖新银鱼遗传多样性空间格局的推断有待进一步深入研究.
作为DNA条形码研究的目的基因应具有相对保守性以便于用通用引物扩增,同时应具有足够的变异以确保物种或种群间的区分度[17].核基因由于太过保守,在物种层次进行鉴别较有难度.线粒体12S和16S核糖体基因中存在大量的插入和缺失现象,使序列比对受到障碍,容易造成错误的比对[33].而线粒体基因组基因插入、删除很少,线粒体13个蛋白编码基因中仅COI、线粒体细胞色素b(Cytb)基因拥有适合的长度和慢的进化速率[34-35].相比之下,COI基因序列变化比Cytb慢,蕴含简明的系统发育信息,更适合解析亲缘关系密切的分类类群,因而被较广泛地应用于动物物种鉴定和系统发育关系的生命条码研究[17-20].Hebert等[17,34]认为当种间的遗传距离大于种内的遗传距离,且距离差异大约10倍时,相关目的基因可以有效鉴别物种.本例研究结果显示太湖新银鱼COI基因种内平均遗传距离为0.125%,而与同科的大银鱼(Protosalanx hyalocranius)、短吻间银鱼(Hemisalanx brachyrostralis)及寡齿新银鱼(N.oligodontis)等种间遗传距离平均值为4.443%(未发表).种间遗传距离为种内遗传距离的35倍.相比鲚属(Coilia)、鲌属(Culter)、大马哈鱼(Oncorhynchus sp.)以及班鳟属(Salmo)等鱼类,其种内的遗传距离更小,且远远小于Herbert建议的1%的水平[34,36].综合本结果和郭立等[16]的研究结论,我们认为线粒体COI基因非常适合银鱼科鱼类的物种鉴别和系统发育研究.
就种下水平的研究而言,COI基因蕴含的信息量小于线粒体Cytb基因.同期测序结果显示,太湖新银鱼Cytb基因的变异率约为COI基因的3倍(未发表),Zhao等的研究也给出了近似的结果[15].
总之,COI基因适宜银鱼科鱼类DNA条形码分析及对银鱼科鱼类进行快速有效的物种鉴定.而在同种不同地理种群间遗传关系研究中应结合Cytb等基因一并分析,以获取足够量的信息,服务于太湖新银鱼等银鱼的种质资源评估、动态监测和科学管理.
致谢:本研究的样品采集得到黄德利、许隆君、研究地区各级渔业主管部门的大力支持和湖区渔民的鼎立帮助,遗传多样性分析实验得益于张爱兵教授的指导,在此一并致以诚挚的感谢.
[1]陈宁生.太湖所产银鱼的初步研究.水生生物学集刊,1956,(2):324-335.
[2]张玉玲.中国新银鱼属Neosalanx的初步整理及其一新种.动物学研究,1987,8(3):277-286.
[3]Nelson JS.Fishes of the world:4th edition.Hoboken:John Wiley and Sons,2006:601.
[4]Xu J,Xie P,Zhang M et al.Icefish(Salangidae)as an indicator of anthropogenic pollution in freshwater systems using nitrogen isotope analysis.Bulletin of Environmental Contamination and Toxicology,2007,79(3):323-326.
[5]张玉玲.银鱼科鱼类系统生物地理学初步研究.见:张广学.系统进化动物学论文集:第二集.北京:中国科学技术出版社,1993:65-77.
[6]王忠锁,傅萃长,雷光春.中国银鱼的多样性及其保护对策.生物多样性,2002,10(4):416-424.
[7]Wang ZS,Shi JQ,Xu CR et al.Degradation of icefishes(Salangidae)in the Yangtze River basin of China:threats and strategies.Environmental Biolgy of Fishes,2009,86(3):109-117.
[8]Dou SZ,Chen DG.Taxonomy,biology and abundance of icefishes,or noodlefishes(Salangidae),in the Yellow River estuary of the Bohai Sea,China.Journal of Fish Biology,1994,45(5):737-748.
[9]胡传林,刘家寿.我国银鱼研究概况及其移植的生态管理准则.湖泊科学,2001,13(3):204-210.
[10]张 颖,董 仕,王 茜等.大银鱼和太湖新银鱼同工酶遗传组成的研究.大连水产学院学报,2005,20(2):111-115.
[11]夏德全,曹 萤,吴婷婷等.用RAPD方法分析太湖大银鱼、太湖新银鱼和寡齿新银鱼的亲缘关系.中国水产科学,2000,7(1):12-15.
[12]Kim JK,Kai Y,Nakabo T.Genetic diversity of Salanx ariakensis(Salangidae)from Korea and Japan inferred from AFLP.Ichthyollgical Research,2007,54(4):416-419.
[13]Zhang J,Li M,Xu MQ et al.Molecular phylogeny of icefish Salangidae based on complete mtDNA cytochrome b sequences,with comments on estuarine fish evolution.Biological Journal of the Linnean Society,2007,91(2):325-340.
[14]张际峰.2种银鱼线粒体COⅡ及侧翼tRNA基因的测定分析及其亲缘关系研究.中国海洋大学学报,2008,38(3):424-428.
[15]Zhao L,Zhang J,Liu ZJ et al.Complex population genetic and demographic history of the Salangid,Neosalanx taihuensis,based on cytochrome b sequences.BMC Evol Biol,2008,8:201(10.1186/1471-2148-8-201).
[16]郭 立,李 隽,王忠锁等.基于四个线粒体基因片段的银鱼科鱼类系统发育.水生生物学报,2011,35(3):449-459.
[17]Hebert PDN,Ratnasingham S,de Ward JR.Barcoding animal life:cytochrome coxidase subunit 1 divergences among closely related species.Proceedings of the Royal Society B:Biological Sciences,2003,270(Suppl 1):96-99.
[18]Vences M,Thomas M,Bonett RM et al.Deciphering amphibian diversity through DNA barcoding:chances and challenges.Philosophical Transactions:Biological Sciences,2005,360(1462):1859-1868.
[19]Ward RD,Zemlak TS,Innes BH et al.DNA barcoding Australia's fish species.Philosophical Transactions:Biological Sciences,2005,360(1462):1847-1857.
[20]Witt JDS,Therlof DL,Hebert PDN.DNA barcoding reveals extraordinary cryptic diversity in an amphipod genus:implications for desert spring conservation.Molecular Ecology,2006,15(10):3073-3082.
[21]Schlei OL,Crête-Laf renire A,Whiteley AR et al.DNA barcoding of eight North American coregonin species.Mol Ecol Res,2008,8(6):1212-1218.
[22]王中铎,郭昱嵩,陈荣玲等.南海常见硬骨鱼类COI条码序列.海洋与湖沼,2009,40(5):608-614.
[23]Hall TA.BioEdit:A user-friendly biological sequence alignment editor and analysis program for Windows 95/98/NT.Nucleic Acids Symposium Series,1999,41:95-98.
[24]Tamura K,Dudley J,Nei M et al.MEGA4:Molecular Evolutionary Genetics Analysis(MEGA)software version 4.0.Molecular Biology and Evolution,2007,24:1596-1599.
[25]Rozas J,Sánchez-Delbarrio JC,Messeguer X et al.DnaSP,DNA polymorphism analyses by the coalescent and other methods.Bioinformatics,2003,19:2496-2497.
[26]Clement M,Posada D,Crandall KA.TCS:a computer program to estimate gene genealogies.Molecular Ecology,2000,9(10):1657-1660.
[27]Excoffier L,Laval G,Schneider S.Arlequin ver.3.0:An integrated software package for population genetics data analysis.Evol Bioinformatics Online,2005,(1):47-50.
[28]孙帼英.长江口及其邻近海域的银鱼.华东师范大学学报:自然科学版,1983,(1):111-119.
[29]朱成德.太湖银鱼产量与水位关系的数理统计分析.淡水渔业,1982,(4):40-42.
[30]张开翔,高礼存,张 立.洪泽湖所产太湖短吻银鱼的初步研究.水产学报,1982,6(1):9-16.
[31]Wang ZS,Lu C,Hu HJ et al.Freshwater icefishes(Salangidae)in the Yangtze River basin of China:spatial distribution patterns and environmental determinants.Environmental Biology of Fishes,2005,73(3):253-262.
[32]王忠锁,吕 偲,许崇任等.江湖阻隔对短吻间银鱼空间发生格局的影响.生物多样性,2005,13(5):407-415.
[33]肖金花,肖 晖,黄大卫.生物分类学的新动向——DNA条形编码.动物学报,2004,50(5):852-855.
[34]Hebert PDN,Cywinska A,Ball SL et al.Biological identifications through DNA barcodes.Proceedings of the Royal Society B:Biological Sciences,2003,270(1512):313-321.
[35]周晓犊,杨金权,唐文乔等.基于线粒体COI基因DNA条形码的中国鲚属物种有效性分析.动物分类学报,2010,35(4):819-826.
[36]彭居俐,王绪祯,王 丁.基于线粒体COI基因序列的DNA条形码在鲤科鲌属鱼类物种鉴定中的应用.水生生物学报,2009,33(2):271-276.
Genetic diversity of Neosalanx taihuensis based on mitochondrial COI sequences
ZHANG Di1,2,LEI Guangchun1,GONG Cheng3& WANG Zhongsuo2
(1:School of Nature Conservation,Beijing Forestry University,Beijing 100083,P.R.China)
(2:College of Life Science,Capital Normal University,Beijing 100048,P.R.China)
(3:Swan Oxbow National Reserve for Lipotesvexillifer,Shishou 434400,P.R.China)
Neosalanx taihuensis is an annual fish with commercial importance.This species is endemic to China,occurring in inland waters affiliated to the eastern-flowing rivers southern to the Yellow River,e.g.the rivers of Yellow,Huaihe,Yangtze,Ou and Pearl etc.Among these river basins,the Yangtze River tests to be the most important region for this species with the highest fishery stocks and the most populations,and thus to be the core area for its conservation and sustainable exploitation.In the near decades,however,N.taihuensis decreased severely with most of the natural populations extinguished economically or even ecologically.The local population extinction infers that the species'genetic diversity must have suffered bad influence and some genetic types must have lost as result.To test this presumption,we conducted an icefish-specific investigation in the mid-lower reaches of Yangtze River during August,2006.Totally eight populations were sampled and used in the followed experiment on their genetic diversity.Based on the gene diversity of mitochondrial cytochrome oxidase subunit I(COI),we mainly focus on two points:1)to find out the genetic diversity in single population and among populations for understanding the genetic status of wild N.taihuensis in the river basin,2)to check the suitability of COI gene for DNA barcoding study in the family of Salangidae.We selected 132 specimens,representing eight different geographic populations in the mid-lower Yangtze River basin,and analyzed their base sequences of COI gene.Among the 630 bp nucleotide sequence of COI gene,eight(1.27%)was variation sites,contributing to eight gene haplotypes.Although the number of gene haplotype and the nucleotide diversity index(π =0.00112 ±0.00204)were not high,haplotype diversity(h)was fairly rich(h=0.576 ± 0.036)in the populations at river basin scale.Taking spatial distribution pattern into account,most gene haplotypes distributed in spatially narrow region with quite low occurrence incidences of 12.5%or 25.0%,occurred in one or two lakes respectively.Haplotype richness in each population varied significantly from each other,with a high fixation index.The least haplotypes occurred in the two highly isolated lakes of Dongting and Liangzi,and the highest haplotype richness occurred in the two lakes with icefish introduction histories.This result indicates that a high inter-population variation existed in the studied region.So we concluded that wild populations of N.taihuensis have suffered great ecological isolation,although they are geographically near to each other.Comparing with several other salangid fishes,e.g.Protosalanx hyalocranius,Hemisalanx brachyrostralis and N.oligodontis,the inter-specific genetic distance was 4.443%,amounting to some 35 times of that in N.taihuensis populations(0.125%).Compared with other fish taxa,such as Coilia,Culter,Oncorhynchus and Salmo,the intra-specific genetic distance was much smaller within N.taihuensis populations(<1%).In conclusion,the COI gene is good and suitable to DNA barcode study in species identifying and systematic study,and can be useful in the assessment,monitoring and conservation management of icefish genetic diversity.
Neosalanx taihuensis;Yangtze River;COI;genetic diversity;DNA barcode
* 国家自然科学基金项目(30570290)、北京市自然科学基金项目(8112010)和北京市教委科技计划项目(KM201110028011)联合资助.2011-05-03收稿;2011-08-15收修改稿.张迪,女,1988年生,硕士研究生;E-mail:zhangdibluesky@163.com.
** 通信作者;E-mail:zhongsuo@yahoo.com.cn.
