中药资源研究常用生物信息学技术
2012-11-07谢腾王升马炯郭兰萍
谢腾,王升,马炯,郭兰萍*
(1.西南交通大学生命科学与工程学院,四川 成都 610031;2.中国中医科学院中药研究所,北京 100700;3.北京大学深圳研究生院环境与能源学院,广东 深圳 518055)
中药资源研究常用生物信息学技术
谢腾1,2,王升2,马炯3,郭兰萍2*
(1.西南交通大学生命科学与工程学院,四川 成都 610031;2.中国中医科学院中药研究所,北京 100700;3.北京大学深圳研究生院环境与能源学院,广东 深圳 518055)
生物信息学的兴起和快速发展在全球取得的巨大进展和成就,为中药资源研究提供了新的研究方法技术和思路,并且极大地推动了中药资源的研究和产业发展。综述中药资源研究常用的生物信息学技术,以期为中药资源研究提供基础信息和方法。
中药资源;生物信息学技术;综述
中药资源(Chinese Medicinal Resources),是在一定空间范围内可供作为中药使用的天然药物及其蕴藏量的总和[1],包括植物类资源、动物类资源和矿物类资源,是中药产业发展的物质基础。其中,中药植物资源是自然界来源最广泛的中药资源。
作为一门新的交叉学科和工具学科,生物信息学技术在揭示中药次生代谢产物的产生、解释道地药材形成的分子机制、选育抗胁迫的药用植物和中药鉴定新标准的建立等中药资源研究中应用广泛。本文将对目前中药资源研究常用的生物信息学技术进行综述。
1 生物信息数据库技术
数据库是生物信息学的主要内容和研究基础,通过对这些数据的分类、收集、整理,产生了成千上万的数据库,几乎涉及了生命科学的各个研究领域,如核酸序列数据库,蛋白质序列数据库,蛋白质、核酸、多糖的三维结构数据库,基因组数据库,文献数据库和其他种类数据库。目前应用比较广泛的数据库有:GenBank、EMBL、DDBJ等核酸序列数据库和 PIR、MIPS、Swiss-Prot等蛋白质序列数据库。
以药用植物名为检索词,在GenBank(http://www.ncbi.nlm.nih.gov/genbank/)检索了截至 2012年5月64个常见中草药品种,于表1记录了核苷酸序列注册情况,并举例了生物信息研究常见的核酸序列、GenBank登录号以及序列的长度。

表1 GenBank数据库中64个常见中草药品种核苷酸序列注册情况和序列举例

续表1
2 生物芯片
生物芯片是指通过微电子和微加工技术在固相基质表面构建微型生物化学分析系统,通过检测集成在芯片上的分析微阵列,来分析细胞及其他的生物分子,快速获取样品的生物信息,也称为微点阵(Micro Array)技术[2]。生物芯片技术具有高信息量、快速、微型化、自动化、成本低、污染少、用途广等特点[3]。常见的生物芯片有:基因芯片、蛋白质芯片、细胞芯片、组织芯片、糖芯片和芯片实验室等[4]。
目前生物芯片技术已经应用于包括拟南芥、水稻、玉米、番茄、草莓、松树、大豆、白杨等植物物种中[5],新的芯片技术例如质子束直写技术[6]以及新型生物芯片例如数字微流控芯片[7-8]、三维生物芯片[9]和低成本的一次性塑料生物芯片[10]等是近年生物芯片研究的热点。
生物芯片技术虽尚处于幼年阶段,仍然有许多需要改进的地方,但芯片技术以其连续化、集成化、微型化的优势为科学家们所青睐,在植物科学领域迅速应用。
生物芯片在植物方面的应用主要集中在:基因表达检测与新基因的发现;植物分子病理中的研究[11];植物逆境和营养方面的研究;转基因植物检测及植物检疫等方面,也为道地药材科学内涵的阐述提供了强有力的工具。丹参道地药材cDNA芯片的构建是以传统药用植物和道地药材为研究主题的首张芯片,得到的114个在丹参毛状根不同时期差异表达的独立基因(Unigone)中有52个为丹参中首次报道[12]。在药材的鉴定方面,李绍平等[13]对川贝母、浙贝母、皖贝母和湖北贝母4种主要贝母品种进行序列分析发现,以川贝母特有碱基序列cTITFGTcGQ TcA为探针制成的芯片,可准确鉴定川贝母[14]。
3 测序技术
DNA测序技术是现代生物学研究中基本的手段之一,它的出现极大地推动了生物学的发展。表2是对三代测序技术比较全面的概括和比较。

表2 三代测序技术的比较
DNA是生命遗传的物质基础,包含着生命发生发展的遗传特征,是道地药材分子生物学研究的基础[15]。小分子RNA如miRNA和siRNA等介导的转录后基因调控是植物中的一种新型基因调控机制,在植物生长、发育以及应答外界胁迫等方面起到重要作用[16]。植物叶绿体基因组独立于核基因组,属于母系遗传,具有高度的保守性和生物转化的性状稳定性,物种进化和系统发育分析研究也是道地药材研究的重要方向。
作为最重要的分子生物学分析方法之一,高通量测序技术在植物DNA、小分子RNA以及叶绿体DNA等方面的测序发展快速,表3中收录了近年已发表的采用高通量测序的部分物种,其中多种物种基因组序列密码解析过程中都采用了新一代测序技术[17]。
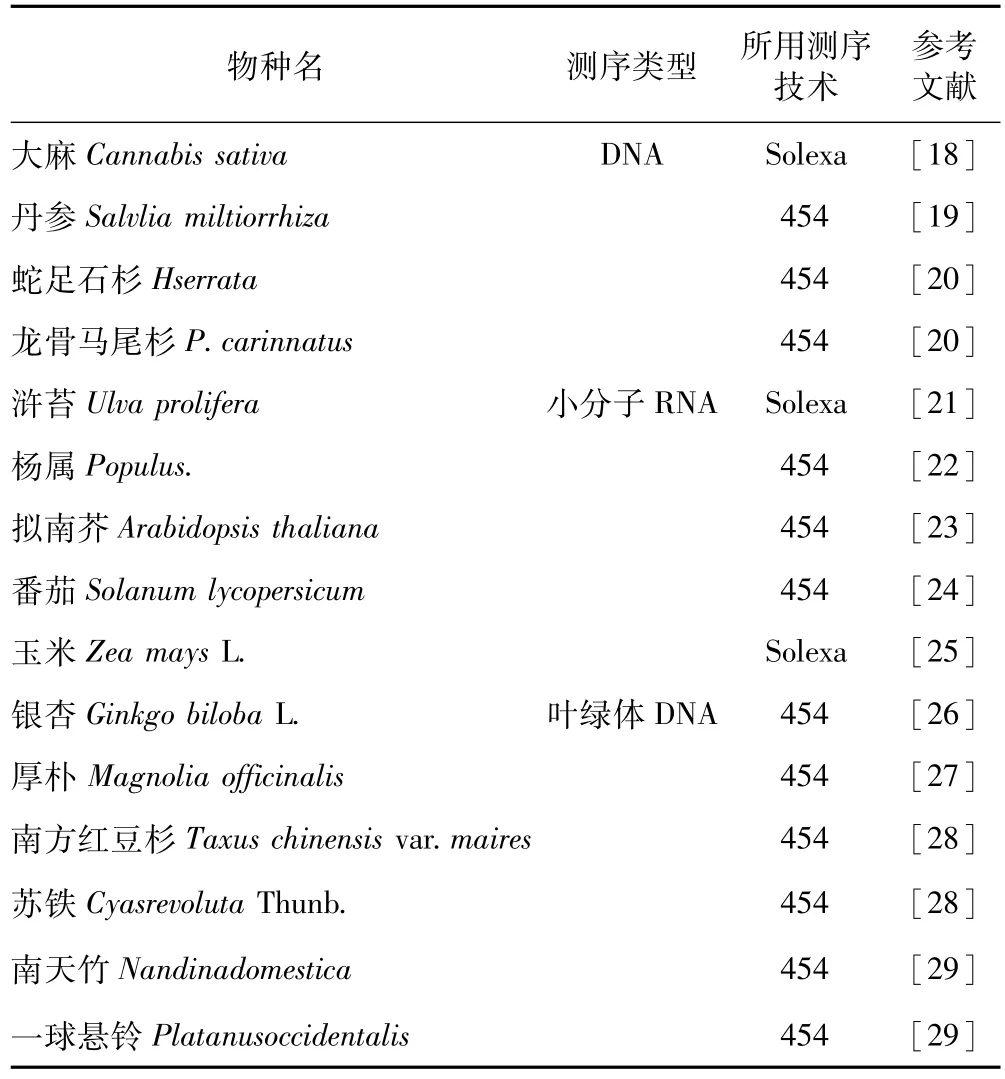
表3 使用高通量测序的部分药用植物和常见植物
4 EST
表达序列(Expressed Sequence)是指基因组表达为RNA的序列,ESTs(Expressed Sequence Tags)是从已建好的cDNA库中随机取出一个克隆,从5’末端或3’末端对插入的cDNA片段进行一轮单向自动测序,所获得的约60~500 bp的一段cDNA序列。ESTs是cDNA克隆的一部分序列,携带了表达基因的部分遗传序列,具有组织特异性。EST获得快速、简便且廉价,是研究基因表达最有用的材料和基因组学的主要内容,广泛地应用于基因组物理图谱的绘制、基因识别、基因表达谱的构建和新基因的发现等研究方向[30]。为了方便研究,表4给出了常用的EST数据库及其网站。

表4 常用的EST数据库
对EST数据的分析包括聚类前处理(pre-Processing)、聚类(Clustering)、拼接(Assembly)和分析Analysis)。数据聚类分析对于EST的功能识别、剪接产物的区分、基因表达谱的分析都有很大的帮助,经聚类和装配得到的唯一序列代表了对所研究的物种(组织)在某一特定时间所表达的基因的随机采样的结果[31]。为了方便研究,表5给出了常用EST聚类和拼接软件,并从四个方面作了比较。此外 SequencherTM、Repeatmasker、Seqclean、Esttrimmer、Contig Express等也用来做EST聚类。

表5 常用EST聚类和拼接软件比较
EST技术作为基因组学、功能基因组学与后基因组学之间的桥梁,已成为分子生物学研究的有力工具。EST技术最基本的方法就是建立EST数据库。利用ESTs技术建立基因数据库的其他药用植物还有人参、薄荷、丹参、银杏、甘草、长春花[32]、藏红花[33]等。
EST数据库在药用植物研究方面的另一重要用途是鉴别和发现药用植物代谢途径中的新基因为道地药材遗传多样性、性状关联基因标记、药材真伪和品质优劣的鉴别[34]等提供新的技术手段。
EST的测序方法存在一定的误差(精确度为97%),目前dbEST数据库中EST并不精确,EST技术所获得的也只是一种模拟序列,只能作为参考,对结果的最终确认还要通过实验验证。尽管如此,EST仍然是获取生物信息快捷和经济的方法。图1给出了截至2012年5月,对30个常见中药药用植物以药用植物名为检索词,检索并统计其在dbEST( http://www.ncbi.nlm.nih.gov/projects/dbEST/)中EST的注册情况。

图1 部分中药药用植物在dbEST中注册EST的情况
5 序列比对
生物信息学分析方法种类繁多,但最基本的分析方法是以数据库为基础的序列比对、新序列的识别、结构和功能的预测以及软件的研制等[35]。序列的比对是生物学中最为基础的一个部分。最常见的比对是蛋白质序列之间或核酸序列之间的两两比对。
通过分析序列之间的相似和差异,寻找二者可能的保守区域、位点,探索产生共同功能的序列模式。常用的序列比对方法有双序列比对和多序列比对。表6提供了常用的序列比对软件和网址,且所提供的软件均适用于蛋白质和DNA序列的比对。
随着DNA测序技术的不断发展,DNA序列分析在药用植物的分类和系统关系、鉴定、道地性以及序列变异与药用成分的关系等方面开始得到了应用,在推进中药现代化等诸多方面展示出广阔的应用前景,为道地药材的研究注入了新的活力[36]。

表6 常用序列比对分析软件
基于DNA测序和序列比对的DNA条形码(DNA barcoding)是近年来分子生药鉴定的研究热点和方向。DNA条形码最早是2003年由加拿大动物学家Hebert提出[37],陈士林等[38]认为 DNA条形码技术在药用植物鉴定中的成功应用是中药鉴定方法的革命性突破。目前,道地药材分子鉴定涉及到栝楼[39]、川乌[40]、草乌[40]、人参[41]、柴胡[42,43]、木瓜[44]、地龙[45]、威灵仙[46]、芍药[46]、土茯苓[46]、浙贝母[47]、丹皮[48]、金银花[49]、金钗石斛[50]、小茴香[51]、山 药[52]、京 大 戟[53]、金 钱 草[54]、天 胡荽[54]、过 路 黄[54]、 阳 春 砂[55]、 草 果[55]、 黄 花蒿[56]、天门冬[57]、石豆兰属[58]、广藿香[59]等诸多生药。
陈士林等学者[60-61]提出了将ITS2作为药用植物鉴定的通用条形码序列的观点,并在后续的研究中得到不断论证。金钱白花蛇、乌梢蛇及酒乌梢蛇、蕲蛇及酒蕲蛇的分子鉴定方法应用于2010年版 《中国药典》[62],这是 《中国药典》首次采用分子标记方法鉴定药材。
在蛇床子[63]6个居群的叶绿体matK基因测序分析研究中发现蛇床子居群间的亲缘关系与地理分布及所含香豆素化学型呈明显的相关性。对广藿香[64]基因序列(18SrDNA、matK基因)分析也得到类似的相关性。李萍等[49]对金银花不同居群5SrDNA基因间区的测序分析结果与邢俊波等[65]对绿原酸含量的分析结果相一致,认为基因的差异是金银花药材道地性的因素之一。蔡朝晖等[47]发现不同产地浙贝母的5SrDNA基因间区具有相同的碱基序列,总生物碱含量相当且含有相同的单体生物碱,因此认为不同产地浙贝母的差异不是由碱基序列引起,而是小环境因素造成的。
此外,ITS序列分析也被用于分析不同居群的遗传差异,铁皮石斛[66]、山参[67]、太子参[68]、阳春砂[69]等的DNA序列分析结果显示ITS序列差异与居群类型存在相关性。
6 生物云计算
生物云计算(Biological Cloud Computing)是基于云计算(Cloud Computing)的新兴生物信息学工具,将生物信息计算能力作为一种商品通过互联网进行流通,为庞大的生物信息计算提供了强大的计算能力,在降低计算成本的同时也提高了研发效率。
集成云计算面向服务的多自主体系统(CISM,Cloud Computing Integrated into Service-Oriented Multi-Agent)架构允许发展动态、分布式和高度可扩展的生物信息软件[70]。MapReduce、MPI和云计算在Alu重复序列和EST构建中的表现得出:云技术在处理大型数据集、移动数据等方面可以达到简化的效果[71]。此外,生物云计算平台的搭建也有相关的报道,如基于水稻功能组学研究的TTRSIS的建设[72]和基于植物科学 iPlant Atmosphere的提出[73]等。
目前比较成熟而实用的云计算产品有IBM蓝云、亚马逊Amazon EC2、谷歌Google A PPEngine、微软Windows Azure,以及后起之秀红帽云计算服务。
7 系统发育分析
系统发育学也称系统发生学(Phylogeny),是基于分子数据差异(不同的基因或DNA片段它们之间存在的进化速率差异)来研究物种的形成或进化过程和机制,以及物种之间的进化关系的一门学科,是生物信息学的一个重要分支。在中药资源的研究中通过系统发育研究,可以探讨植物的分类群间的系统发育关系,以及进化的过程和机制,可以了解物种间的进化关系和历史,探索物种的起源和未知基因的功能,进行药材差异性研究和微生物生态学的研究。
基于分子水平的系统发育树构建方法可以分为四大类:(1)基于字符序列的方法:最大简约法/M P;最大似然法/ML);贝叶斯法/Bayesian。(2)基于距离的算法是目前应用最广的一类系统发育树构建方法。其理论基础是最小进化原理(mini-mum evolution,ME)。两个经典算法是平均连接聚类法/UPGMAU)和邻接法/NJ。此外还有FM法。(3)神经网络法不再以距离矩阵为输入数据,而是以序列为输入数据,通过网络对序列地不断学习,自适应生成系统发育树。(4)基于图形表示的系统发育树的构建方法。表7给出了常用的几种系统发育分析软件以及构建系统发育树的方法。
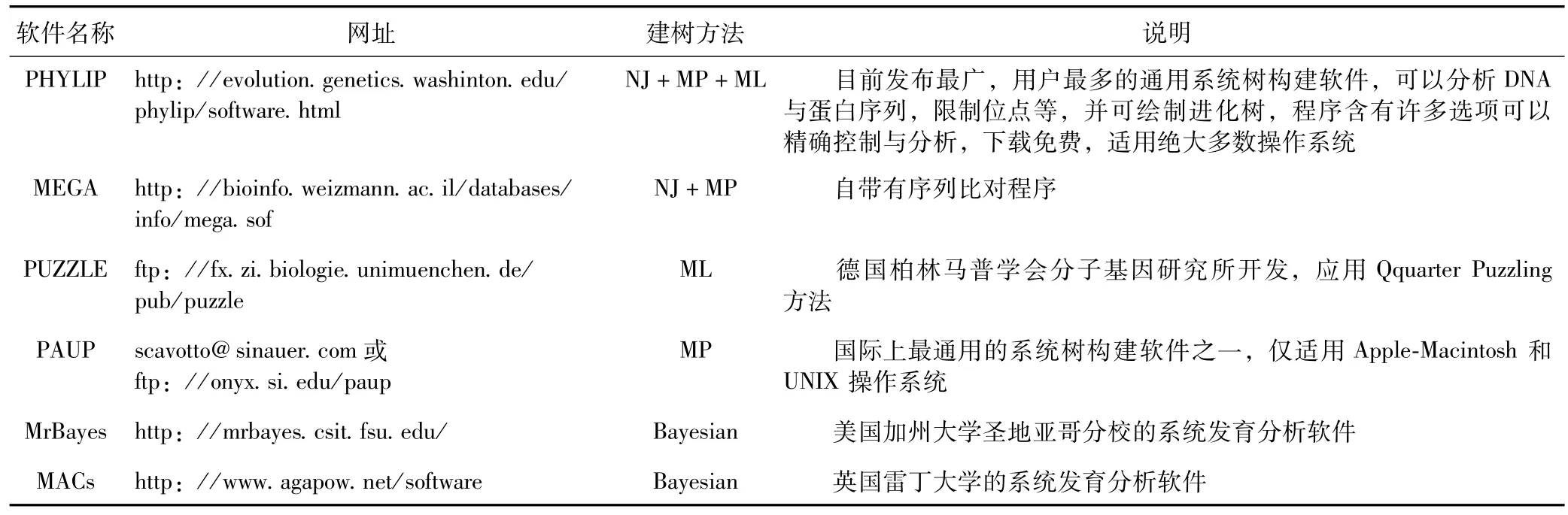
表7 常用系统发育分析软件
系统发育分析后,通过一种分支图表即进化树来反映同一谱系的进化关系。目前经常使用到的部分系统发育分析软件如表7所示。可以根据不同的研究目的,采用不同的序列片段来探究系统发育问题,但是通过计算机所构建的系统发育树只是一个物种进化的模型,并不一定完全符合自然界真实的物种进化规律。表8列举了近年进行的系统发育分析部分的植物,以及所采用的系统发育方法和软件。

表8 植物系统发育分析研究举例
生物信息学技术在中药资源研究中应用日趋广泛和深入,不过生物信息学技术在中药资源领域的应用总的来说还未成熟,只是处在初生阶段,相关研究还是零散的,并不成系统,目前也没有中药资源的生物信息数据库的建立,不过生物信息学技术未来在揭示中药资源生物学规律将发挥更大作用。
[1]杨世海.中药资源学[M].北京:中国农业出版社,2006:1-15.
[2]李瑶.基因芯片技术[M].北京:化学工业出版社,2004:1-13.
[3]胡业华.生物芯片技术[J].江西教育学院学报,2004,24(3):24-30.
[4]周冬生,杨瑞馥.生物芯片分类及其技术原理[J].微生物学免疫学进展,2002,30(3):101-107.
[5]倪进斌,吕德方,高亚梅,等.生物芯片在植物科学中应用的研究进展[J].现代化农业,2010,(7):1-3.
[6]S Homhuan,B Zhang,F-S Sheu,et al.Single-cell electroporation using proton beam fabricated biochips[J].Proc of SPIE,2010,7716:1-9.
[7]Krishnendu Chakrabarty,Yang Zhao.Digital Microfluidic Biochips:A Vision for Functional Diversity and More than Moore[R].NC,USA:IEEE,2010.
[8]Wajid Hassan Minhass,Paul Pop,Jan Madsen.System-Level Modeling and Synthesis of Flow-Based Microfluidic Biochips[R].NY,USA:IEEE,2011.
[9]Xiangdong Xue,Silvia Marson,Mayur K.Patel,et al.Progress towards the design and numerical analysis of a 3Dmicrochannel biochi Pseparator[J].Int JNumer Meth Biomed Engng,2011,27:1771-1792.
[10]Ishtiaq Saaem,Kuo-Sheng Ma,S.Munir Alam,Jingdong Tian.Fabrication of plastic biochips[J].JVac Sci Technol A,2010,28(4):963-968.
[11]马新颖,汪琳,任鲁风,等.10种植物病毒的基因芯片检测技术研究[J].2007,37(6):561-565.
[12]崔光红.丹参道地药材cDNA芯片构建及毛状根基因表达谱研究[D].北京:中国中医科学院,2006.
[13]李绍平.生物芯片技术在中药鉴定研究中的应用与展望[J].世界科学技术——中药现代化,2000,2(3):158.
[14]孙科,杨培,闰微.药用植物在生物技术领域中的研究及应用[J].现代农业科技,2007,(19):115-117,119.
[15]王兴春,杨致荣,王敏,等.高通量测序技术及其应用[J].中国生物工程杂志,2012,32(1):109-114.
[16]卫波,张荣志,李爱丽,等.利用高通量测序技术发现植物小分子 RNA研究进展[J].中国农业科学,2009,42(11):3755-3764.
[17]岳桂东,高强,龙海,等.高通量测序技术在动植物研究领域中的应用[J].中国科学,2012,42(2):107-124.
[18]Van Bakel H,Stout J M,Cote A G,et al.The draft genome and transcriptome of Cannabis sativa. [J].Genome Biol,2011,12:R102.
[19]李滢,孙超,罗红梅,等.基于高通量测序454 GS FLX的丹参转录组学研究[J].药学学报,2010,45(4):524-529.
[20]李滢.基于高通量测序技术的药用植物表达序列标签系统的构建与应用[D].北京:中国医学科学院北京协和医学院,2010.
[21]黄爱优,王广策,何林文,等.浒苔小 RNA高通量测序及相关生物信息学分析[J].科学通报,2011,56(24):1993-1998.
[22]Barakat A,Wall PK,Diloreto S,et al.Conservation and divergence ofmicroRNAs in Populus[J].BMCGenomics,2007,8:481.
[23]Rajagopalan R,Vaucheret H,Trejo J,etal.A diverse and evolutionarily fluid set ofmicroRNAs in Arabidopsis thaliana[J].Genes&Development,2006,20(24):3407-3425.
[24]Moxon S J R,Szittya G,Schwach F,et al.Dee Psequencing of tomato short RNAs identifies microRNAs targeting genes involved in fruit ripening[J].Genome Research,2008,18(10):1602-1609.
[25]Nobuta K,Lu C,Shrivastava R,et al.Distinct size distribution of endogeneous siRNAs in maize:Evidence from dee Psequencing in the mop1-1mutant[J].Proceedings of the National Academy of Sciences of the USA,2008,105(39):14958-14963.
[26]林小涵.银杏和木兰属植物的转录组-叶绿体基因组及其相关研究[D].北京:中国医学科学院北京协和医学院,2011.
[27]李西文,胡志刚,林小涵,等.基于454FLX高通量技术的厚朴叶绿体全基因组测序及应用研究[J].药学学报,2012,47(1):124-130.
[28]李卿.灯盏细辛血管保护作用及南方红豆杉等叶绿体基因组研究[D].北京:中国医学科学院北京协和医学院,2011.
[29]Moore MJ.Rapid and accurate pyrosequencing of angiosperm plastid genomes[J].BMC Plant Biology,2006,6:1.
[30]何懿菡,孙坤.生物信息学研究进展[J].青海师范大学学报,2011,(3):69-72.
[31]丁克越,沈岩.ESTs数据分析及ESTs数据系统[J].国外医学,2002,24(2):113-116.
[32]Eujayl I,Sorrells ME,Baum M,et al.Isolation of EST-derived microsatellite marker$for genotyping the A and B genomes of wheat[J].Theor Appl Genet,2002,104(2-31):399-407.
[33]Murata J,Bienzle D,Brandle JE,et al.Expressed sequence tags from Madagascar periwinkle(Catharanthusr Oseus)[J].FEBS Lett,2006,580(18):4501-4507.
[34]Yu JK,Dake TM,Singh S,et al.Development and mapping of EST-derived simple sequence repeat markers for hexaploid wheat[J].Genome,2004,47(5):805-818.
[35]胡鸢雷,生物信息学的发展与现状[OL].[2008-11-05]http://home.bbioo.com/thread-30876-1-1.html.
[36]苏雪,孙坤,张建清,等.DNA序列分析在药用植物鉴定中的应用[J].西北师范大学学报,2006,42(4):79-86.
[37]HEBERT PD,CYWINSK A A,BALL SL,et al.Biological identifications through DNA barcodes[J].Proc Biol Sci,2003,207(1512):313-321.
[38]陈士林,宋经元,姚辉.药用植物DNA条形码鉴定策略及关键技术分析[J].中国天然药物,2009,7(5):322-327.
[39]黄璐琦,王敏,杨滨.栝楼农家品种苗期的分子标识鉴别[J].中国中药杂志,1999,34(9):642.
[40]罗艳,杨亲二.川乌与草乌的ITS序列分析[J].中国药学杂志,2008,43(11):820-823.
[41]陈美兰.采用PAPD和PCR-RFLP方法从分子水平鉴定人参[J].国外医学,2002,24(5):305-306.
[42]黄必胜,熊梦晓,杨昊,等.保康柴胡5S-rDNA间隔序列分析及道地性研究[J].现代中药研究与实践,2007,21(4):14-17.
[43]武莹,刘春生,刘玉法,等.5种习用柴胡的ITS序列鉴别[J].中国中药杂志,2005,30(10):732-734.
[44]HAN B X,PENG H S,YAO Q,et al.Analysis of genetic relationships in germplasms mugua in China revealed by internal transcribed spacer and its taxonomic significance[J].Zeitschrift Fur Naturforschung Section C,2010,65(7-8):495-500.
[45]吕国庆,牛宪立,姬可平.动物性中药材地龙 DNA条形码初步研究[J].广东农业科学,2011,17:114-116.
[46]曾旭,李莉,业宁,等.基于ITS2条形码的中药材威灵仙与其易混伪品的鉴定[J].环球中医药,2011,4(4):264-269.
[47]蔡朝晖,李萍,李松林,等.不同产地浙贝母的基因序列及生物碱含量比较[J].中药材,2001,24(3):157-159.
[48]刘海兵,宋社吾,刘道芳,等.丹皮药材及其伪品、混淆品核糖体 rDNA ITS2的序列分析[J].生物学杂志,2008,25(4):33-36.
[49]李萍,蔡朝晖,邢俊渡.5S-rRNA基因间区序列变异用于金银花药材道地性研究初探[J].中草药,2001,32(9):834-837.
[50]葛晓军,肖鲲,李小琼,等.赤水金钗石斛rDNA ITS序列分析[J].江苏大学学报,2008,18(6):510-512,531.
[51]刘义梅,罗焜,陈科力,等.ITS2序列鉴定小茴香及其常见混伪品[J].环球中医药,2011,4(4):260-263.
[52]王东,白雁,陈志红.不同产地山药rDNA ITS区序列的比较[J].时珍国医国药,2007,18(1):54-55.
[53]石林春,宋经元,李莉,等.基于ITS2条形码序列的京大戟及其易混品的DNA分子鉴定[J].环球中医药,2011,4(2):103-108.
[54]庞晓慧,宋经元,孙超,等.中药金钱草及其混淆品的ITS2序列分析与鉴别[J].环球中医药,2011,4(2):95-98.
[55]韩建萍,李美妮,石林春,等.砂仁及其混淆品的ITS2序列鉴定[J].环球中医药,2011,4(2):99-102.
[56]张祖荣,廖志华,彭梅芳.黄花蒿HDS基因的克隆与功能分析[J].中草药,2012,3(1):148-154.
[57]欧立军,佘朝文,许栋,等.天门冬叶绿体2个缺失片段的差异研究[J].时珍国医国药,2011,22(1):167-168.
[58]蒋明,陈贝贝,贺蔡明.石豆兰属植物rDNAITS序列的克隆与分析[J].中草药,2012,43(2):343-349.
[59]张英.广东道地药材广藿香的GC-MS化学指纹图谱和DNA指纹图谱研究[D].北京:北京中医药大学,2007.
[60]刘义梅.杜鹃花属药用植物DNA条形码的鉴定研究[D].武汉:湖北中医药大学,2011.
[61]罗焜,陈士林,陈科力,等.基于芸香科的植物通用DNA条形码研究[J].中国科学,2010,40(4):342-358.
[62]国家药典委员会.中国药典[S].一部.北京:中国医药科技出版社,2010:72.
[63]曹晖,蔡金娜,刘玉萍,等.蛇床子地理分布与叶绿体matK基因序列的相关性分析[J].中国药学杂志,2001,36(6):373-379.
[64]刘玉萍,罗集鹏,冯毅凡,等.广藿香的基因序列与挥发油化学型的相关性分析[J].药学学报,2002,37(4):304-308.
[65]邢俊波,李萍,胡世林.金银花道地药材品质评价研究Ⅰ:HPLC法测定不同产地金银花中绿原酸的含量[J].中国野生植物资源,1999,18(6):44-46.
[66]丁小余,王峥涛,徐珞珊,等.F型、H型居群的铁皮石斛rDNA ITS区序列差异及SNP现象的研究[J].中国中药杂志,2002,27(2):85-89.
[67]马小军,汪小全,肖培根,等.野山参与栽培参rDNA内转录间隔区(ITS)序列比较[J].中国中药杂志,2000,25(4):206-209.
[68]余永邦,秦民坚,梁之桃,等.不同产区太子参的rDNA ITS区序列的比较[J].植物资源与环境学报,2003,12(4):1-5.
[69]王培训,黄丰,周联,等.阳春砂与几种常见姜科伪充品的 RAPD分析[J].中药材,2000,23(2):712-731.
[70]Javier Bajo,Carolina Zato,Fernando de la Prieta,et al.Cloud Computing in Bioinformatics[J].Distrib.Computing&Artif.Intell.AISC,2010,79:147-155
[71]Jaliya Ekanayake,Thilina Gunarathne,Judy Qiu.Cloud Technologies for Bioinformatics Applications[J].Parallel and Distributed Systems,IEEE Transactions on,2011,22(6):998-1011.
[72]Ming-Jen Fan,Su-May Yu,Tzu-Chieh Hung,et al.TTRSIS:A Cloud Computing Platform for Rice Functional Genomics Research through a Reverse Genetics Approach[R].Taichung,Taiwan:IEEE,2011.
[73]iPlant Atmosphere:A Gateway to Cloud Infrastructure for the Plant Sciences[R].NY,USA:ACM,2011.
[74]唐先华,张晓艳,施苏华,等.睡莲类植物ITS+nrDNA序列的分子系统发育分析[J].地球科学,2003,28(1):97-101.
[75]王秀荣,赵杨,骈瑞琪,等.胡枝子属植物ITS序列研究与系统发育分析[J].2008,23(5):70-73.
[76]李悦.中国莴苣属及其近缘属植物系统学关系的初步研究[D].郑州:郑州大学,2010.
[77]马剑.重楼属几种重要植物的遗传多样性和化学指纹图谱研究[D].北京:中央民族大学,2009.
[78]康兆茹,周厚成,赵霞,等.草莓果实MADS-box基因保守片段的克隆与序列分析[J].生物技术通报,2011(9):76-80,89.
[79]李艳萍,魏继承,何淼,等.基于nrDNA ITS序列东北地区丁香属植物的系统发育关系[J].东北林业大学学报,2010,38(8):45-47.
[80]侯鑫,刘俊娥,赵一之.中国锦鸡儿属的分子系统发育[J].植物分类学报,2008,6(4):600-607.
[81]尧金燕,赵南先,郝刚,等.中国榕属榕亚属植物的系统发育初探[J].广西植物,2009,29(1):7-12.
[82]朱颖,董玉芝,昝少平,等.基于ITS序列探讨忍冬属的系统发育关系[J].西北植物学报,2010,30(2):250-254.
[83]曹国艳.木兰科含笑属及其相关属的ISSR系统发育分析[D].昆明:西南林学院,2009.
[84]刘湘志,龙桂友,邓子牛.柑橘Ptcor8基因相关功能的生物信息学分析[J].湖南农业大学学报,2011,37(2):161-165.
[85]许崇梅,曲畅游,于文光,等.基于ITS序列探讨金线草的系统学位置[J].广西植物,2011,31(3):300-303.
[86]江玉梅,夏冰,李晓丹,等.石蒜psaH基因的克隆及分析[J].江苏农业科学,2010(4):29-31.
[87]杨光伟.中国桑属(Morus L.)植物遗传结构及系统发育分析[D].重庆:西南农业大学,2003.
[88]代金霞,王玉炯,郭晶静,等.荒漠植物柠条根瘤菌的抗逆性及其系统发育分析[J].干旱地区农业研究,2011,29(4):223-227.
[89]段双全,陈彬,张正果,等.西藏虫草属真菌生物学特性研究及其无性型 rRNA基因 ITS间隔区系统发育分析[J].西藏大学学报,2011,26(1):7-14.
[90]柴华,沙伟.地衣的ITS序列系统发育分析和 DNA条形码的初探[J].生物技术,2011,21(1):1-4.
[91]王亚玲,李勇,张寿洲,等.用matK序列分析探讨木兰属植物的系统发育关系植物分类学报[J].2006,44(2):135-147.
App lications of Common Bioinformatics Technology in Study of Chinese M edicinal Resources
XIE Teng1,2,WANG Sheng2,MA Jiong3,GUO Lan-ping2
(1.School of Life Science and Engineering,Southwest Jiaotong University,Chengdu610031,China;2.Institute of Chinese Materia Medica,China Academy of Chinese Medical Sciences,Beijing100700,China;3.The School of Environment and Energy,Peking University Graduate School in Shenzhen,Shenzhen518055,China)
Bioinformatics,arising in the late 20th century,is one of themajor frontiers of life sciences and natural sciences of this century.Extensive applications of bioinformatics in life science research field result in huge economic and social benefits.Chinesemedicinal resources including plant,animal,and mineral are the basis of the development of Chinesemedicine industry.Among them,Chinesemedicinal plant resource is natural source and is the most widely used Chinese medicine resources.The rise and rapid development of bioinformatics have made great progress and achievements in theworld,provided new research ideas to the study of Chinesemedicine resources,and greatly promoted the study of Chinese medicine resources and industrial development.This review gives a comprehensive collection and analysis of the application of bioinformatics in the study of Chinese medicine resources and an outlook of bioinformatics,so as to provide the basic information and ideas for the study of it.
Chinesemedicinal resources;Bioinformatics Technology;Summary
*[通讯作者]郭兰萍,Tel:(010)64011944,E-mail:glp01@126.com
