传统发酵酸牛奶中乳酸菌多态性分析
2012-10-25魏冉冉霍贵成
魏冉冉,方 伟,霍贵成
(东北农业大学乳品科学教育部重点实验室,黑龙江哈尔滨 150030)
传统发酵酸牛奶中乳酸菌多态性分析
魏冉冉,方 伟,霍贵成*
(东北农业大学乳品科学教育部重点实验室,黑龙江哈尔滨 150030)
以从内蒙古呼伦贝尔市牧区采集的1份传统发酵酸牛奶样品为研究对象,对其进行乳酸菌的分离鉴定。通过传统纯培养法分离出17株菌,并对17株菌进行16S rDNA序列分析、多位点pheS序列分析和生理生化鉴定,鉴定的结果为11株乳酸乳球菌乳酸亚种、1株格式乳球菌、1株粪肠球菌、2株植物乳杆菌植物亚种及2株弯曲乳杆菌。乳酸乳球菌乳酸亚种为优势菌(占总分离菌株的64.7%)。
酸牛奶,乳酸菌,多态性
内蒙古地区牧民长期饮用的酸牛奶,就是以鲜牛奶为原料,未经任何处理在自然环境中发酵而成的乳制品。发酵的过程不需要商品发酵剂,而是用前一天的酸奶做天然发酵剂接种到新鲜的奶中使其自然发酵成酸奶,其味清酸爽,开胃增食,是农牧民野外、农耕、放牧的理想食品。常和奶油混合用来拌炒米或其他米饭吃。如加入少许白糖,夏季尤为酸甜可口,为牧区夏季日常饮用食品之一[1]。由于其是自然发酵,未经任何处理,因此其微生态环境未遭破坏,而且由于它特殊的制作方法使其形成了自己的特色,使其中的乳酸菌的生物学特性和基因多样性得到了很好的保留,因此这些丰富的乳酸菌资源有待于开发和利用。对这些乳酸菌进行分离鉴定和生物学特性的研究,对传统乳制品中乳酸菌种类的认识、推动乳酸菌的基础研究以及开发应用地域特色的乳酸菌制品都有重要意义。本研究以从内蒙古呼伦贝尔市牧区采集的1份传统发酵酸牛奶样品为研究对象,对其中的乳酸菌多样性进行了研究,不仅能够研究牧区独特自然发酵乳中微生物区系组成,而且能为进一步开发利用内蒙古自治区牧民自制酸牛奶中的乳酸菌奠定基础。
1 材料与方法
1.1 材料与仪器
样品 采自内蒙古呼伦贝尔市牧民家庭自制的1份传统发酵酸牛乳;乳酸菌富集用加富的脱脂乳培养基 10%脱脂乳、0.5%蛋白胨、0.25%酵母粉、1%葡萄糖[2];分离培养 采用MRS培养基分离乳酸杆菌和M 17培养基分离乳酸球菌[3];做生理生化实验所用的各种糖发酵实验的培养基 均为青岛海博有限公司生产的细菌微量生化反应管。
光学显微镜(OLMPUS)、蒸汽灭菌器、恒温培养箱、超净工作台、PCR仪,凝胶成像仪。
1.2 实验方法
1.2.1 样品的采集 采集牧民家庭中的传统发酵酸牛乳于灭菌管中,并封好放入冰盒内,带回实验室于4℃冰箱中保存备用。
1.2.2 乳酸菌的分离、纯化与保存 样品→富集培养→涂布(稀释度为10-5、10-6)→挑取可疑菌落→液体培养基培养→平板划线分纯(分纯三次)→纯培养物→革兰氏染色(G+);过氧化氢实验(-)→甘油保存(-80℃)(50%的甘油,菌液∶甘油为6∶4)。
1.2.3 菌株基因组DNA小量提取 取1m L MRS或M 17液体培养物(过夜培养或培养24h),10000r/m in离心1m in,收集菌体。采用天根公司的细菌基因组DNA提取试剂盒提取DNA。提取的DNA置于-20℃保存。提取的DNA作为16S rDNA序列分析,多位点pheS序列分析的样品。
1.2.4 16S rDNA序列分析 利用引用通用引物27F和1541R[4-5]对菌株的16S rDNA片段进行扩增,正向引物为27F:5’-AGAGTTTGATCCTGGCTCAG-3’,反向引物为1541R:5’-AAGGAGGTGATTCCAGCC-3’。
扩增条件如下:94℃预变性5m in;30个循环:94℃变性1m in,58℃退火1m in,72℃延伸2m in;72℃末端延伸10m in,然后将扩增成功的产物直接送华大纯化和测序。参比序列采用乳酸菌已知模式菌株的16S rDNA序列(这些序列从GeneBank中获取)[6]。序列的对比和系统发育树的构建都通过Mega 4软件(Tamura,et al,2007)[7]完成。系统发育树的构建采用邻比法[8]和maximum composite likelihood model模型。自展分析基于1000次重复。
1.2.5 pheS基因序列分析[9]利用引物pheS-21-F和pheS-23-R对菌株的pheS基因序列片段进行扩增,正向引物为pheS-21-F:5’-CAYCCNGCHCGYGAYATGC-3’,反向引物为pheS-23-R:5’-GGRTGRACCATVCC NGCHCC-3’。
扩增条件如下:94℃预变性5m in;30个循环:94℃变性1min,46℃退火1min 15s,72℃延伸1min 15s;72℃末端延伸7m in,然后将扩增成功的产物直接送华大纯化和测序。参比序列采用乳酸菌已知模式菌株的pheS(这些序列从GeneBank中获取)。序列的对比和系统发育树的构建都通过Mega 4软件完成。系统发育树的构建采用邻比法和maximum composite likelihood model模型。自展分析基于1000次重复。1.2.6 乳酸菌的生理生化鉴定 鉴定乳酸乳球菌,需要做40℃生长实验、4%NaCl生长实验、pH 9.2生长实验和糖发酵实验。糖发酵实验利用细菌微量生化反应管,将活化好的菌液离心后,用生理盐水重新悬浮,注入管内,再放入灭菌大试管中,37℃培养一周后观察结果。实验鉴定结果均参照《乳酸菌分类鉴定及实验方法》[10]、《伯杰细菌鉴定手册》[11]、《常见细菌系统鉴定手册》[12]的标准进行判定,确定其属种。
2 结果与讨论
2.1 各菌株16S rDNA序列分析
各菌株的序列分析结果总结在树状图中(见图1)。
菌株KLDS 6.1101与粪肠球菌(Enterococcus faecalis)系统发育关系最为密切,有99.9%的16S rDNA序列相似性。因而菌株KLDS 6.1101可以初步鉴定为粪肠球菌(Enterococcus faecalis)。

图1 基于16S rDNA序列的树状图Fig.1 Phylogenetic tree based on the 16S rDNA alignment
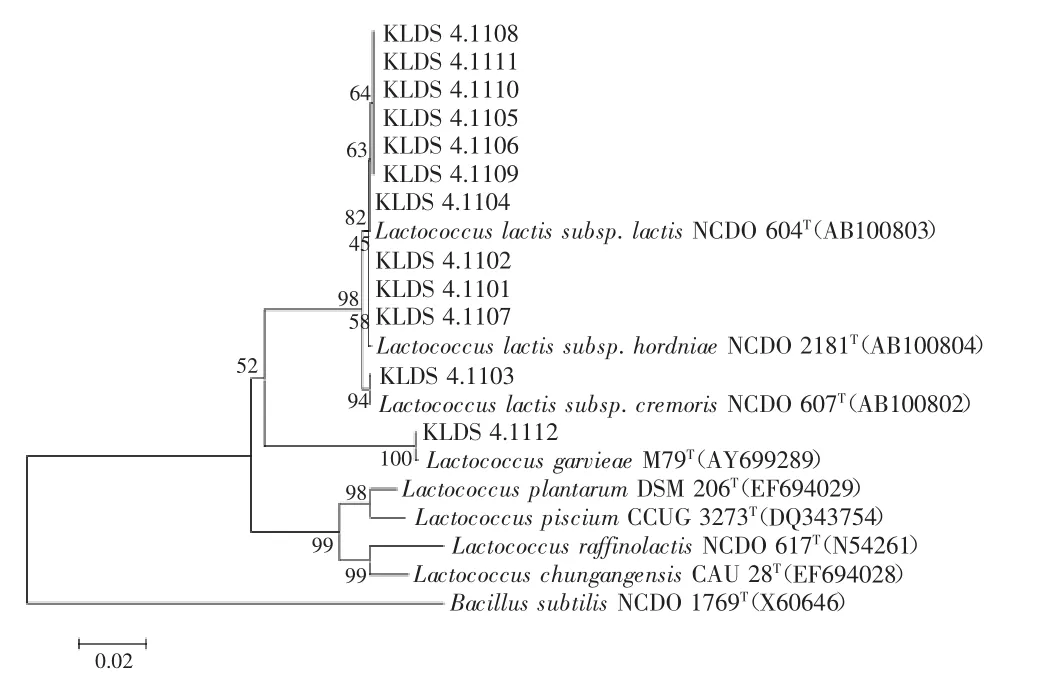
图2 基于16S rDNA序列的树状图Fig.2 Phylogenetic tree based on the 16S rDNA alignment
菌株KLDS 4.1112与格式乳球菌(Lactococcus garvieae)系统发育关系最为密切,有99.9%的16S rDNA序列相似性,因而菌株KLDS 4.1112可以初步鉴定为格式乳球菌(Lactococcus garvieae)。由此树状图可以看出乳酸乳球菌形成单一的DNA同源群,但是按照表型特征足可以将它们分为三个亚种,它们是Lactococcus lactis subsp.cremoris、Lactococcus lactis subsp.hordniae和Lactococcus lactis subsp.lactis。菌株KLDS 4.1101、KLDS 4.1102、KLDS 4.1103、KLDS 4.1104、KLDS 4.1105、KLDS 4.1106、KLDS 4.1107、KLDS 4.1108、KLDS 4.1109、KLDS 4.1110、KLDS 4.1111属于乳酸乳球菌,它们与Lactococcus lactis subsp.cremoris、Lactococcus lactis subsp.hordniae、Lactococcus lactis subsp.lactis的16S rDNA序列相似性都在99%以上。本实验的结果也说明了很难通过16S rDNA全序列测定的方法将它们区分出来,因此它们进一步的分离可以根据生理生化实验的结果。
菌株KLDS 1.1101和KLDS 1.1102属于植物乳杆菌(L.p lantarum)组,与Lactobacillus p lantarum subsp. argentoratensis、Lactobacillus pentosus、Lactobacillus p lantarum subsp.p lantarum、Lactobacillus parap lantarum和Lactobacillus fabifermentans系统发育关系密切,分别有99.6%、99.7%、99.6%、99.7%和99.0%的16S rDNA序列相似性,可以初步鉴定为植物乳杆菌(L.p lantarum),其确切的分类地位还需要其他分类学方法才能确定。

图3 基于16S rDNA序列的树状图Fig.3 Phylogenetic tree based on the 16S rDNA alignment
菌株KLDS 1.1103和KLDS 1.1104属于弯曲乳杆菌(L.curvatus)组,与Lactobacillus fuchuensis、Lactobacillus graminis、Lactobacillus sakei 和Lactobacillus curvatus系统发育关系密切,分别有98.0%、99.4%、99.0%和99.6%的16S rDNA序列相似性,除了Lactobacillus fuchuensis外,菌株KLDS 1.1103和KLDS 1.1104与其他三个菌种的16S rDNA序列相似性都很高,菌株KLDS 1.1103和KLDS 1.1104可以初步鉴定为弯曲乳杆菌(L.curvatus),其确切的分类地位还需要其他分类学方法才能确定。
2.2 pheS基因序列分析
菌株KLDS 1.1101和KLDS1.1102与Lactobacillus p lantarum subsp.argentoratensis、Lactobacilluspentosus、Lactobacillus p lantarum subsp.plantarum、Lactobacillus paraplantarum、Lactobacillus fabifermentans的16S rDNA序列相似性都很高,因此在16S rDNA序列分析的基础上,通过pheS基因序列进一步对菌株KLDS 1.1101和KLDS 1.1102的分类地位进行确定。

图4 基于pheS基因序列树状图Fig.4 Phylogenetic tree based on the pheSgene alignment
菌株KLDS 1.110和KLDS 1.1102与Lactobacillus p lantarum subsp. argentoratensi、 Lactobacillus pentosus、Lactobacillus plantarum subsp.plantarum、Lactobacillus parap lantarum 和 Lactobacillus fabifermentans分别有90%、80.3%、100%、89.5%和77.7%pheS序列相似性,因此菌株KLDS 1.1101和KLDS 1.1102鉴定为植物乳杆菌植物亚种(Lactobacillus p lantarum subsp.p lantarum)。
2.3 乳酸乳球菌生理生化鉴定结果
乳酸菌种的鉴定主要是分离菌株对碳水化合物的发酵产酸实验,即通过对单糖、二糖、三糖、多糖以及糖苷和糖醇类等碳水化合物的利用情况来区分[10-12]。对于某些球菌还有pH 9.2、4%NaCl和40℃的生长实验。对菌株KLDS 4.1101、KLDS 4.1102、KLDS 4.1103、KLDS 4.1104、KLDS 4.1105、KLDS 4.1106、KLDS 4.1107、KLDS 4.1108、KLDS 4.1109、KLDS 4.1110、KLDS 4.1111进行生理生化鉴定,鉴定项目和结果如表1所示,11株乳酸乳球菌能发酵半乳糖、乳糖、麦芽糖和核糖,不能发酵松三糖、蜜二糖和棉籽糖,能在pH 9.2、4%NaCl和40℃条件下生长,其结果和《乳酸菌分类鉴定及实验方法》[10]、《伯杰细菌鉴定手册》[11]、《常见细菌系统鉴定手册》[12]中描述的乳酸乳球菌乳酸亚种的生理生化结果一致,因此将11株菌鉴定为乳酸乳球菌乳酸亚种。
3 结论
3.1 从传统发酵酸牛奶中分离和纯化了17株乳酸菌,对这17株乳酸菌进行16S rDNA全序列分析,可以初步将这些菌株鉴定为以下乳酸菌种:11株乳酸乳球菌、1株格式乳球菌、1株粪肠球菌、2株植物乳杆菌和2株弯曲乳杆菌。

表1 乳酸乳球菌的生理生化鉴定结果Table 1 Identification results of physiological and biochemical test of lactococcus lactis
3.2 对菌株KLDS 1.1101和KLDS 1.1102的确切的分类地位通过pheS序列分析进行了确定,可将菌株KLDS 1.1101和KLDS 1.1102鉴定为植物乳杆菌植物亚种。用16S rDNA全序列分析和生理生化相结合将菌株KLDS 4.1101、KLDS 4.1102、KLDS 4.1103、KLDS 4.1104、KLDS 4.1105、KLDS 4.1106、KLDS 4.1107、KLDS 4.1108、KLDS 4.1109、KLDS 4.1110、KLDS 4.1111鉴定到亚种,这些菌株均为乳酸乳球菌乳酸亚种。
3.3 内蒙古传统发酵酸牛奶中的乳酸菌种类和数量有所不同,呈现出多样性,其中乳酸乳球菌乳酸亚种为优势菌占总分离菌的64.7%,可能正是它们赋予了酸牛奶特殊的风味和质地。
[1]刘计民.内蒙古传统风味民族乳食品简介[J].中国乳品工业,1993,21(5):237-240.
[2]赵蕊,霍贵成.新疆酸奶子中乳酸菌多样性分析[J].山东大学学报:理学版,2008,43(7):18-22.
[3]Amrane A,Prigent Y.Lactic acid production rates during the different growth phases of Lactobacillus helveticus cultivated on whey supplemented with yeast extract[J].Biotechnology Letters,1998,20(4):379-383.
[4]Mauro S,Diego M,Silvia C,et al.Development of genus/ species-specific PCR analysis for identification of carnobacterium strains[J].CurrentMicrobiology,2002,45:24-29.
[5]Liu W,Sun Z,Zhang J,etal.Analysis ofmicrobial composition in acid whey for dairy fan making in Yunnan by conventional method and 16S rRNA sequencing[J].Current Microbiology,2009,59:199-205.
[6]Altschul,Gish,Miller,et al.Basic local alignment search tool [J].JMol Biol,1990,215:403-410.
[7]Tamura K,Dudley J,et al.MEGA 4:molecular evolutionary genetics analysis(MEGA)software version 4.0[J].Mol Biol Evol,2007,24,1596-1599.
[8]Saitou N&Nei M.The neighbor-joining method:a new method for reconstructing phylogenetic trees[J].Mol Biol Evol,1987,4,406-425.
[9]Naser S M,Dawyndt.Identification of lactobacilli by pheS and rpoA gene sequence analyses[J].Int JSyst Evol Microbiol,2007,57,2777-2789.
[10]凌代文.乳酸细菌分类鉴定及实验方法[M].北京:中国轻工业出版社,1999.
[11]R E布坎南,等.伯杰细菌鉴定手册[M].第八版.北京:科学出版社,1984.
[12]东秀珠.常见细菌系统鉴定手册[M].北京:科学出版社,2001.
Diversity analysis of lactic acid bacteria isolated from the traditional fermented m ilk
WEIRan-ran,FANGW ei,HUO Gui-cheng*
(Key Laboratory of Dairy Science,Ministry of Education,Northeast Agricultural University,Harbin 150030,China)
17 isolates of lactic acid bac teria(LAB)were isolated from the trad itional fermented m ilk gathering from the herdmen’s home in Hulunbeier of Inner Mongolia by traditional pure culture method.Based on 16S rDNA sequence analysis,pheS sequence analysis and physiological and biochem ical test,these isolates were identified as 11 strains of Lactococcus lactic.subsp.lactic,1 strain of Lactococcus garvieae,1 strain of Enterococcus faecalis,two strains of Lactobacillus p lantarum subsp.p lantarum and 2 strains Lactobacillus curvatus.Lactococcus lactic.subsp.lactic was the p redom inant strain(64.7%of the total isolates).
fermented m ilk;lactic acid bacteria;d iversity
TS201.3
A
1002-0306(2012)22-0210-04
2012-05-21 *通讯联系人
魏冉冉(1986-),女,硕士研究生,研究方向:乳品科学与微生物。
“乳酸菌代谢调控与发酵剂制造技术”教育部长江学者和创新团队发展计划资助(IRT0959)。
