西伯利亚鲟热休克蛋白HSP70 cDNA的克隆、序列分析和组织分布
2012-09-25田照辉徐绍刚王巍胡红霞董颖宋超
田照辉,徐绍刚,王巍,胡红霞,董颖,宋超
(国家淡水渔业研究中心暨北京市水产科学研究所,北京 100068)
热休克蛋白(Heat shock proteins,HSPs)又称应激蛋白,其中HSP70家族高度保守,当机体遭受应激后会被诱导而迅速表达。HSP70具有分子伴侣功能,可以提高机体对应激因子的抵抗能力,在免疫过程中具有重要作用[1-6]。热休克蛋白因具有广泛的生物学功能而成为研究热点。一些学者对黄颡鱼、团头鲂、剑尾鱼等多种鱼类的HSP70基因进行了克隆和研究[5-9],还有学者建议将HSP70的表达作为潜在性的环境应激和毒性量化指标[10]。
西伯利亚鲟Acipenserbaerii属鲟形目、鲟科、鲟属,为亚冷水性鱼类。该鱼的养殖利润较高,因而成鱼养殖得到迅速推广。在成鱼养殖过程中,养殖户的盲目选址以及为追求产量加大鲟的放养密度,由此而引起的拥挤胁迫和夏季水温过高引起的高温胁迫时有发生,致使西伯利亚鲟抗病力下降,病害增加。本研究中,作者首次克隆了西伯利亚鲟HSP70 cDNA的全长,并对其在各组织中的分布进行了分析,旨在为研究HSP70在西伯利亚鲟抗应激过程中的作用和机理奠定基础。
1 材料与方法
1.1 材料
试验用西伯利亚鲟取自北京市水产科学研究所国家级鲟鱼良种场, 其中1尾(150 g)用于基因克隆试验,3尾(165 g±23.46 g)用于组织分布试验。
RNeasy mini kit提取试剂盒和RNase-Free DNase Set为Qiagen公司产品;Trizol Reagent、DNaseI、RT-PCR试剂盒购于Invitrogen公司;ExTaq、SMART RACE cDNA Amplification Kit(Clontech)和pGM-19T Vector载体连接试剂盒购自TaKaRa公司;胶回收试剂盒为Axygen公司产品;实时定量试剂为AB公司生产的2×Sybrgreen PCR Master Mix。克隆菌株DH10B由北京市水产科学研究所生物技术与育种研究室保存。
1.2 方法
1.2.1 引物合成及测序 所有引物用Primer 5.0设计(表1),其中中间片段引物HSPFP1、HSPRP2根据GenBank中登录的白鲟、鲤、斑马鱼、鲑的HSP70基因保守区设计,其中3′HSP1、3′HSP2为3′RACE特异引物,5′GSP1为5′RACE特异引物,2010722F和2010722R为克隆OFR引物,4f和4r为实时定量引物。所有引物合成及序列测定均由北京擎科生物工程有限公司完成。

表1 试验用引物
1.2.2 HSP70 cDNA全长序列的获取
1)总RNA的提取。取1尾西伯利亚鲟(150 g)于水温为24 ℃的水池中暂养1周,再转入水温为30 ℃的水池中急性热应激1.5 h。取其肝脏,用Trizol法提取总RNA,用BIORAD紫外分光光度计测定RNA的浓度和OD值,根据OD260nm/OD280nm值和琼脂糖凝胶电泳判断RNA的质量。
2)合成cDNA第一链。用Dnase I处理总RNA,按照SuperScript III First-Strand Synthesis System for RT-PCR (Invitrogen)使用说明,以Oligo(dT)20为引物进行反转录,合成cDNA第一链。
3)中间片段的分离。以cDNA第一链为模板,根据GenBank中登录的HSP70保守区设计引物HSPFP1和HSPRP2扩增西伯利亚鲟HSP70中间片段。PCR反应体系包括10×buffer 2 μL、10 mmol/L dNTPs 0.8 μL、10 μmol/L引物各0.8 μL、5 U/μL ExTaq 0.2 μL、模板2.0 μL,加灭菌ddH2O补至20 μL。反应程序为:94 ℃下预变性3 min;94 ℃下变性45 s,55 ℃下退火45 s,72 ℃下延伸2 min,共进行35个循环;最后在72 ℃下再延伸10 min。PCR产物经12 g/L琼脂糖凝胶电泳,用Axygen胶回收试剂盒回收,连接pMD19-T载体,并转化至DH10B大肠杆菌感受态细胞中,进行蓝白斑菌筛选。挑取单个白色菌落过夜培养,通过菌液鉴定挑选阳性菌液进行测序。
4)3′端和5′端扩增。3′HSP1、3′HSP2为根据中间片段序列设计的3′端特异性引物,其中3′HSP2为嵌套引物,AP和AUAP为非特异性引物。第一轮PCR反应以cDNA第一链为模板,利用3′HSP1和锚定引物AP进行扩增;第二轮以第一轮产物为模板,以3′HSP2和AUAP为引物,进行巣式PCR得到特异性单一片段。使用Clontech Race试剂盒对5′RACE进行扩增。扩增产物用琼脂糖凝胶电泳分离,胶回收,连接转化,进行蓝白斑菌筛选和菌液鉴定,对阳性菌液进行测序。
5)HSP70 cDNA全长序列的确定。使用DNAStar中的SeqManⅡ将上述中间片段、3′端和5′端片段拼接成完整的西伯利亚鲟HSP70 cDNA序列。为验证全长拼接的准确性,在OFR两端设计引物2010722F和2010722R,克隆接近OFR的长片段,并与全长进行比对。
6)序列分析、系统发育树构建以及蛋白结构的预测。使用软件DNAstar、ClustalX 1.83、Mega 4、Antheprot 2000以及在线软件SignalP完成。
1.2.3 HSP70 mRNA的组织分布
1)取样。取体质量为(165±23.46)g(n=3)的西伯利亚鲟于17.5 ℃水中暂养一周,试验时转入含1 g/L丁香酚的水中,迅速麻醉后取其肝脏、鳃、脾脏、心脏、肌肉、中肠、性腺、脑8种组织,于液氮中速冻,-80 ℃下保存。
2)RNA的提取与反转录。使用Qiagen公司生产的 RNeasy mini kit试剂盒提取总RNA,在RNase-Free DNase set柱上处理基因组,用紫外分光光度计测定OD值,保证总RNA样本的OD260 nm/OD280nm值为1.9~2.0,用琼脂糖凝胶电泳检验RNA的完整性。
3)荧光实时定量。采用SYBR Green法,将待测样品cDNA经过系列稀释后作为标准品,构建相对标准曲线[10-12]。具体步骤如下:取总RNA 1 μg,用SuperScript III First-Strand Synthesis System for RT-PCR (Invitrogen)进行反转录,并进行无反转录酶(NRT)和无RNA(NOT)对照。实时定量引物4f、4r的扩增片段为100 bp,预先采用普通PCR和琼脂糖凝胶电泳验证引物的特异性。实时定量时取1尾鱼8种组织的cDNA等量混合,梯度稀释30~36倍为模板,假定稀释36倍的模板的copy值为1,作相对标准曲线。将各组织的cDNA稀释16.3倍作为模板,与标准曲线同时定量分析,并与NTC(无模板阴性对照)、NRT、NOT进行对照。反应总体积为20 μL,包括2×Sybrgreen PCR Master Mix 10 μL,10 μmol/L 4f、4r各0.7 μL,模板2 μL,用灭菌ddH2O补至20 μL。反应程序为:95 ℃下预变性10 min;95 ℃下变性15 s,58 ℃下退火15 s,72 ℃下延伸45 s,共进行40个循环,并采集荧光信号。每个模板重复3次,实时定量结束后立即进行熔解曲线分析,熔解曲线从55 ℃到95 ℃,每30 s升高0.5 ℃,采集荧光15 s。
1.3 数据处理
试验数据采用SPSS 11.5软件进行处理,用One-Way进行单因素方差分析,如果差异显著则用Ducan’s法进行多重比较。
2 结果与分析
2.1 总RNA
分离制备的总RNA的OD260 nm/OD280nm值为1.9~2.0,用琼脂糖凝胶电泳检测,28S、18S rRNA条带清晰(图1),说明提取的RNA质量很好,较完整,适合进行反转录。
2.2 HSP70 cDNA全长
用引物HSPFP1、HSPRP2扩增的中间片段经克隆后测序得到1 182 bp片段,3′RACE经克隆后测序为923 bp,5′RACE经克隆后测序为630 bp(图1)。将上述中间片段、3′和5′序列使用DNAStar软件中的SeqManⅡ拼接得到西伯利亚鲟HSP70 cDNA序列全长为2 343 bp,该序列已登录GenBank(登录号为HM348777)。用引物2010722F和2010722R扩增OFR得到的片段为1 958 bp,与设计片段比对,除几处突变外完全一致。
2.3 序列分析
西伯利亚鲟HSP70 cDNA的全序列为2 343 bp,通过DNAstar软件中的Editseq分析,5′非编码区为140 bp,3′非编码区为256 bp,具有AATAA的加尾信号及多聚A尾。可读编码框(ORF)为1 947 bp,编码为648个氨基酸。氨基酸残基中,负电荷残基(D,E)为96个,正电荷残基(K,R)为81个,亲水性残基(A,I,L,F,W,V) 为209个,极性残基(N,C,Q,S,T,Y)为 161个,整个蛋白带负电荷-13.960(pH 7.0),相对分子质量为71 000,等电点为5.21,具有细胞质特征性保守序列EEVD(645~648aa),C端重复序列为GGMP(617~624 aa)。使用Antheprot 2000搜索到该序列中含有HSP70的3个特征序列(图2),分别为IDLGTTYS([IV]-D-L-G-T-[ST]-x-[SC], 11~18 aa)、IFDLGGGTFDVSIL([LIVMF]-[LIVMFY]-[DN]-[LIVMFS]-G-[GSH]-[GS]-[AST]-x(3)-[ST]-[LIVM]-[LIVMFC], 199~212 aa)和IVLVGGSTRIPKIQK([LIVMY]-x-[LIVMF]-x-G-G-x-[ST]-{LS}-[LIVM]-P-x-[LIVM]-x-[DEQKRSTA], 336~350aa)。KRKYKKDISDNKRAVRRL和RARFEEL为推测的热休克蛋白核定位序列。
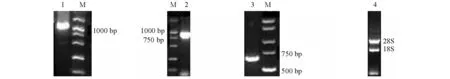
注:1为引物HSPFP1、 HSPRP2扩增的中间片段;2为3′端;3为5′端;4为总RNA。Note:1,the fragments of the primers HSPFP1 and HSPRP2;2,the fragment of 3′ Race; 3,the fragment of 5′ Race; 4,the total RNA.图1 西伯利亚鲟总RNA和HSP70 cDNA的扩增产物Fig.1 The amplification products of total RNA and HSP70 cDNA in Siberian sturgeon Acipenser baerri
2.4 同源性分析
在GenBank中选取部分鱼类、两栖类和爬行类动物的氨基酸序列使用软件ClustalX 1.83进行比对,用Mega 4构建NJ系统发育树,用Bootstrap验证重复1 000次(图3)。从图3可见,

图2 西伯利亚鲟HSP70 cDNA序列及推导出的氨基酸序列Fig.2 The cDNA nucleotide sequence of HSP70 and the amino acids deduced in Siberian sturgeon Acipenser baerii

续图2 西伯利亚鲟HSP70 cDNA序列及推导出的氨基酸序列Cont.Fig.2 The cDNA nucleotide sequence of HSP70 and the amino acids deduced in Siberian sturgeon Acipenser baerii
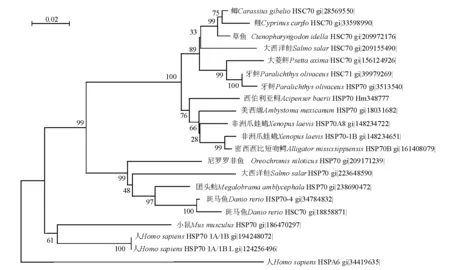
图3 西伯利亚鲟HSP70氨基酸序列系统发育树(NJ树)Fig.3 Phylogenetic tree of HSP70 amino acid sequence in Siberian sturgeon(NJ method)
非洲爪蛙蟾、密西西比短吻鳄、美西螈与西伯利亚鲟的HSP70聚为一支,西伯利亚鲟和美西螈亲缘关系较近。
2.5 蛋白质二级结构和功能预测
用DNAStar软件中的Protean对西伯利亚鲟热休克蛋白的二级结构、抗原位点和亲水性等进行预测 (图4)。经Hydropathy-Kyte-Doolittle预测发现,亲水性区域远远大于疏水性区域,说明该蛋白是亲水性的。经Antigenic-Jameson-Wolf预测潜在的蛋白质抗原决定簇,发现该蛋白的抗原位点丰富,具有良好的抗原性。用Chou-Fasman方案预测该蛋白的二级结构,α-螺旋区占40.7%,β-折叠区占23.4%,转角区占30.2%;用Gamier-Roboson方案预测该蛋白的二级结构,α-螺旋区占47.5%,β-折叠区占35.9%,转角区占6.47%,无规则卷曲区占10.2%。两种方案的预测结果均说明该蛋白的二级结构以α-螺旋为主。利用信号肽预测工具SignalP对西伯利亚鲟HSP70蛋白序列在线搜索,使用神经网络法(NN)和隐马科夫模型(HMM)进行预测,NN预测结果表明,该蛋白没有信号肽位点;HMM预测结果表明,该信号肽概率极低,无信号肽。
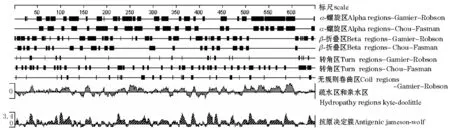
图4 用DNAStar软件预测西伯利亚鲟HSP70的二级结构Fig.4 Predicted HSP70 secondary structure in Siberian sturgeon by DNAStar
2.6 西伯利亚鲟HSP70 mRNA的组织分布
实时定量时取1尾鱼8种组织的cDNA等量混合,梯度稀释30~36倍为模板,假定稀释36倍的模板的copy值为1,作相对标准曲线(图5),仪器根据标准曲线和各组织Ct值自动给出copy值。所得标准曲线斜率为-3.3611,R2=0.9956,扩增效率为98.4%,说明该标准曲线可靠。熔解曲线分析没有杂峰,说明产物单一。NTC(无模板阴性对照)、无反转录酶(NRT)和无RNA(NOT)对照均无信号,表明试验过程没有外源污染和基因组污染。本试验结果表明,水温为17.5 ℃时,西伯利亚鲟肝脏、鳃、脾脏、心脏、肌肉、中肠、性腺、脑8种组织中均有HSP70表达(图6),其中脾脏中的表达量最高,鳃中的次之,肝脏中的最低。除心脏与性腺的表达量、中肠与脑的表达量之间差异不显著外(P>0.05),其余各组织中的表达量之间差异均显著(P<0.05)。
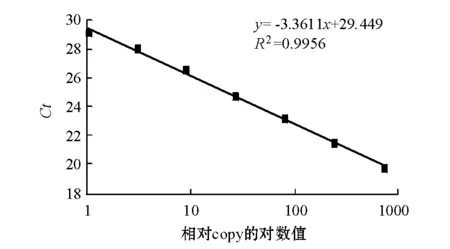
图5 实时定量标准曲线Fig.5 The Standard curve by Real-time PCR
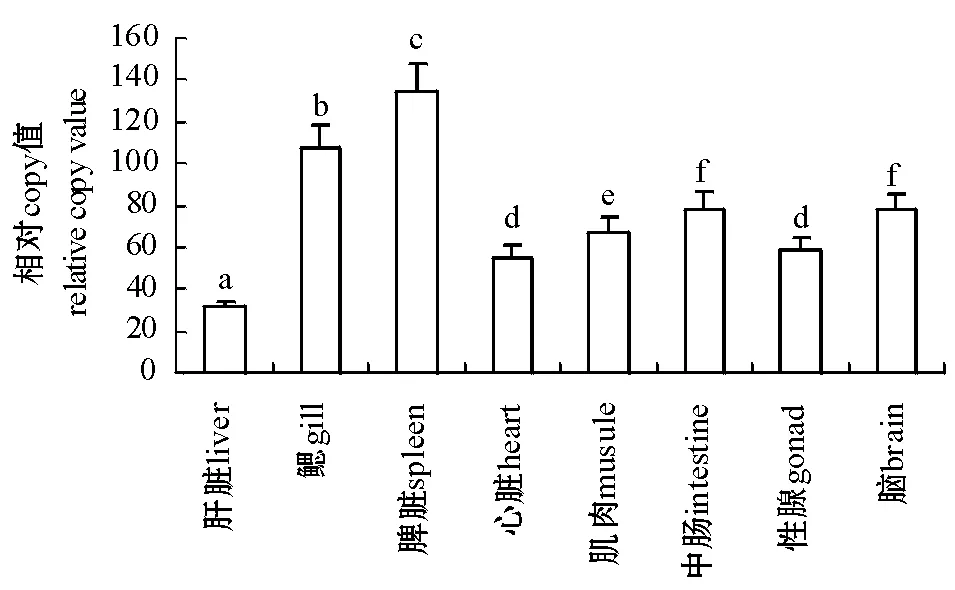
注:标有不同小写字母者表示组间差异显著(P<0.05),标有相同小写字母者表示组间差异不显著(P>0.05)。Note:The means with the different letters are significant differences at the 0.05 probability level,and the means with the same letters are not significant differences.图6 西伯利亚鲟HSP70 mRNA的组织分布Fig.6 Relative expression of HSP70 mRNA in 8 tissues in Siberian sturgeon Acipenser baerii
3 讨论
本研究中得到了西伯利亚鲟HSP70 cDNA的全序列,但由于热休克蛋白家族成员众多,保守性强,成员间有一定的碱基同源性,存在不同基因被错误拼接的可能,所以又进行了OFR克隆与设计片段的比对,发现除几处突变外完全一致,说明拼接的为同一序列。经NCBI Blast测序,所得cDNA序列与其它生物的HSP70 cDNA序列具有较高的同源性,与牙鲆Paralichthysolivaceus的相似度为82%,与草鱼Ctenopharyngodonidella的相似度为84%。GenBank中高首鲟Acipensertransmontanus(AY880255)、大西洋鲟Acipensersturio(EU816599)只有HSP70 cDNA片段,通过DNAssist比对与所得序列中间片段基本一致。所得序列全长为2 343 bp,可读编码框(ORF)为1 947 bp,编码为648个氨基酸。氨基酸序列分析结果表明,该序列中含有HSP70家族的3个特征序列——IDLGTTYS、IFDLGGGTFDVSIL和IVLVGGSTRIPKIQK,所以得到的序列是HSP70 cDNA序列,序列C末端具有热休克蛋白高度保守的细胞质特异调控序列EEVD[7,9],说明该蛋白是细胞质热休克蛋白。蛋白质的二级结构预测结果表明,该蛋白是亲水性的,以α-螺旋为主,具有丰富的抗原位点,无信号肽位点。
热休克蛋白家族中HSP70家族成员最多,包括两种蛋白质:结构型HSC70(Heat shock cognate protein 70)和诱导型HSP70(Heat shock protein 70),这两种蛋白质的氨基酸序列90%相同,大部分特性也相同[13]。鱼类与哺乳动物、两栖类非洲爪蛙蟾HSP70氨基酸序列相似度平均值分别为86.2%和86.8 %[14],进化较为保守。本研究中,系统发育树显示,牙鲆、斑马鱼和人热休克蛋白各自的结构型和诱导型聚成一支,西伯利亚鲟与非洲爪蛙蟾Xenopuslaevis、密西西比短吻鳄Alligatormississippiensis和美西螈Ambystomamexicanum聚为一支,与美西螈亲缘关系最近,这与上述观点相符。高宇等[14]认为,杂交鲟Husohuso♀×Acipenserschrenck♂、匙吻鲟与团头鲂、鲫、鲤、斑马鱼聚为一个类群,和鲫的亲缘关系最近,可能因为他们使用的是部分核苷酸序列构建的MP系统发育树。
鱼类结构型HSC70基因具有内含子,而诱导型HSP70基因没有内含子[2, 15];诱导型HSP70在正常细胞中不表达或表达量很少,在应激条件下表达量迅速增高,而结构型热休克蛋白在所有细胞中均有表达[16]。本研究中,克隆的HSP70在8种组织中都有一定的基础表达,符合结构型的表达特征。试验中用丁香酚迅速将鱼麻醉后取样,避免了因操作而引起的应激。丁香酚对西伯利亚鲟稚鱼耗氧率和幼鱼生化指标能产生一定的影响[17],但能否诱导西伯利亚鲟热休克蛋白的表达尚未见报道。
Deane等[18]对热休克蛋白的结构进行了分析,认为银鲷Sparussarba结构型HSP70有多个GGXP模体和九肽模体SGPTIEEVD,诱导型HSP70多数情况只出现一次GGXP,九肽结构也不同于结构型的SGPTIEEVD。这与明建华等[7]对团头鲂Megalobramaamblycephala和Cui等[19]对三疣梭子蟹Portunustrituberculatus的热休克蛋白基因克隆结果相同。本研究中,克隆的HSP70氨基酸序列中GGMP重复两次,并含有九肽模体SGPTIEEVD,按照Deane等[18]的观点应为结构型HSC70。但是人的诱导型热休克蛋白HSP701A/1B中具有SGPTIEEVD九肽结构模体(NP_005336.3),而结构型HSP701-Like(NP_005518.3)中却无此九肽结构[20];非洲爪蛙蟾Xenopuslaevis诱导型HSP70 1B(NP_001091238.1)中也含有九肽模体SGPTIEEVD和重复的GGMP[21]。这与Deane等[18]的结论不一致。
目前对鲟热休克蛋白的研究较少,仅有部分学者使用免疫印迹法对其它鱼类的热休克蛋白进行了研究[22-24]。中吻鲟Acipensermedirostris仔鱼受到热应激后,会导致畸形个体和正常个体中HSP72 和HSP78的表达量持续升高,HSP60表达水平可能与仔鱼热应激易损性相关[23]。投饲率和温度均能引起高首鲟Acipensertransmontanus热休克蛋白的变化,且HSP70的上升幅度较HSP60更为明显,因而HSP70是更灵敏的生物学指标[24]。鲟属于亚冷性鱼类,水温是非常重要的胁迫因子[22,24]。研究表明,鱼类经过长期的高温驯化可以改变其机体内HSP70的表达机制[25]。所以研究西伯利亚鲟在不同生态环境中HSP70 cDNA的表达,有助于在分子水平上研究西伯利亚鲟的抗逆机理。本研究中克隆了西伯利亚鲟HSP70的cDNA序列,通过实时定量,得到了其在各组织中的分布情况,其中脾脏中的表达量最高,鳃中的表达量次之,肝脏中的表达量最低(P<0.05),因此,HSP70表达量较高的组织可用于监测西伯利亚鲟的应激反应状态。
参考文献:
[1] 孙旭彤,张士璀,刘振辉,等.热激蛋白研究概况及其在海洋生物中的研究进展[J].海洋科学,2002,26(6):24-27.
[2] Basu N,Todgham A E,Ackerman P A,et al.Heat shock protein genes and their functional significance in fish[J].Gene,2002,295:173-183.
[3] Feige U,Polla B S.Heat shock proteins: the hsp70 family[J].Cellular and Molecular Life Sciences,1994,50:979-986.
[4] 张晓鹏.HSP70的生物学功能新进展[J].国外医学卫生学分册,2002,29(6):337-343.
[5] 祝璟琳,王国良.鱼类HSP70 研究进展[J] .宁波大学学报:理工版,2007,20(4):446-450.
[6] 董云伟,董双林,纪婷婷.水生动物热休克蛋白研究进展[J].中国海洋大学学报,2008,38(1):39-44.
[7] 明建华,谢骏,刘波,等.团头鲂HSP70 cDNA的克隆、序列分析以及热应激对其mRNA表达的影响[J].中国水产科学,2009,16(5):635-647.
[8] 万文菊,王纪亭,石存斌,等.溶藻弧菌感染对剑尾鱼HSP70基因表达的影响[J].大连水产学院学报,2007,22(5):330-334.
[9] 张娟,张其中,张占会,等.黄颡鱼HSC70基因及其组织表达分析[J].水生生物学报,2009,33(3):426-434.
[10] 沈骅,王晓蓉,张景飞.应用应激蛋白HSP70作为生物标志物研究锌、铜及其联合毒性对鲫鱼肝脏影响[J].环境科学学报,2004,24 (5):895-899.
[11] 张驰宇,徐顺高,黄新祥.一种新颖简便的荧光实时RT-PCR相对定量方法的建立[J].生物化学与生物物理进展,2005,32 (9):883-888.
[12] Duarte-Guterman P,Trudeau V L.Transcript profiles and triiodothyronine regulation of sex steroid- and thyroid hormone-related genes in the gonad-mesonephros complex ofSiluranatropicalis[J].Molecular and Cellular Endocrinology,2011,331(1):143-149.
[13] 王薇,韩岚岚,赵奎军.昆虫热休克蛋白HSP70的研究进展[J].东北农业大学学报,2009,40(11):129-132.
[14] 高宇,袁改玲,李大鹏,等.杂交鲟和匙吻鲟克隆与序列分析[J].华中农业大学学报,2010,29(1):85-89.
[15] Graser R T,Malnar-Dragojevic D,Vincek V.Cloning and characterization of a 70 kD heat shock cognate (hsc70) from the zebrafish (Daniorerio)[J].Genetica,1996,98:273-276.
[16] 王宇萍,蒋建东.热休克蛋白HSP70的结构和功能[J].中国细胞生物学学报,2010,32(2):305-313.
[17] 冯广朋,庄平,章龙珍,等.麻醉剂丁香酚对西伯利亚鲟稚鱼耗氧率与幼鱼生化指标的影响[J].大连水产学院学报,2010,25(2):118-126.
[18] Deane E E,Woo N Y S.Cloning and characterization of the HSP70 multigene family from silver sea bream:Modulated gene expression between warm and cold temperature acclimation[J].Biochemical and Biophysical Research Communications,2005,330:776-783.
[19] Cui Zhaoxia,Liu Yuan,Luan Weisha,et al.Molecular cloning and characterization of a heat shock protein 70 gene in swimming crab (Portunustrituberculatus)[J].Fish & Shellfish Immunology,2010,28:56-64.
[20] 陈兰英,朱泮民,李冰冰,等.HSP70的结构、特性和功能研究进展[J].平顶山学院学报,2004,13(2):41-43.
[21] Klein S,Starausberg R,Wagner L,et al.Genetic and genomic tools for Xenopus research:The NIH Xenopus initiative[J].Developmental Dynamics,2002,225:384-391.
[22] Werner I,Linares-Casenave J,Van Eenennaam J P,et al.The effect of temperature stress on development and heat-shock protein expression in larval green sturgeon(Acipensermedirostris)[J].Environmental Biology of Fishes,2007,79:191-200.
[23] Silvestre F,Linares-Casenavea J,Doroshov S I,et al.A proteomic analysis of green and white sturgeon larvae exposed to heat stress and selenium[J].Science of the Total Environment,2010,408:3176-3188.
[24] Deng Dongfang,Wang Chunfang,Lee Seunghyung,et al.Feeding rates affect heat shock protein levels in liver of larval white sturgeon (Acipensertransmontanus)[J].Aquaculture,2009,287:223-226.
[25] 许友卿,曹占旺,丁兆坤,等.高温对鱼类的影响及其预防研究[J].水产科学,2010,29(4):235-242.
