大麦TILLING体系在抗病基因研究中的应用
2012-07-16齐新丽吴佳洁付道林
胡 鑫,齐新丽,吕 波,吴佳洁,付道林
(山东农业大学,山东省作物生物学重点实验室,作物生物学国家重点实验室,山东泰安 271018)
栽培大麦(Hordeum vulgare L.)是世界上重要的禾谷类作物,其面积和产量分别为55.90百万公顷和155.34百万吨,仅次于小麦、玉米和水稻[1]。大麦主要用作饲料,其次(大约15%)用于啤酒酿造及人类食用。大麦和小麦同属禾本科小麦族(Triticeae),基本染色体组均包含7个染色体,在基因组成和排列方面高度保守。由于大麦为二倍体,是研究六倍体小麦的重要的模式植物。
TILLING(Targeting Induced Local Lesions In Genomes)是开展反向遗传学研究的有效技术,具有高通量、低成本、不依赖于基因型等特点[2]。TILLING技术涉及突变群体的构建和随机突变的筛选,通常采用甲基磺酸乙酯(Ethyl methane sulfonate,EMS)诱导突变,利用芹菜内切酶CEL I处理鉴定突变位点。Till等人[3]详细介绍了TILLING技术的有关步骤。CEL I内切酶的制备相对简单[4],多数实验室可以独立纯化CEL I。目前,基于CEL I酶切的TILLING技术被广泛应用于多种生物突变群体的筛选。随着深度测序技术的发展,人们开始尝试运用现代测序技术实现高通量TILLING筛选[5]。TILLING技术最先应用于模式生物拟南芥(Arabidopsis thaliana)和果蝇(Drosophila melanogaster)等[6][7]。目前,国际上该技术已经成功应用于水稻[8]、小麦[9][10]和大麦[11]等多种粮食作物。然而,我国在此领域,尤其是在麦类作物方面,还未建成实用的TILLING筛选平台。
在拟南芥中,水杨酸(salicylic acid,SA)和茉莉酸(Jasmonic acid,JA)等植物激素参与调控防御反应[12][13][14]。水杨酸和茉莉酸之间存在协同和拮抗并存的复杂调控关系,通过与植物免疫信号网络的微妙配合,决定植物细胞的防御效果[15]。水杨酸信号途径主要调控对活体寄生的病原菌和病毒的反应,而茉莉酸信号途径主要调节对腐生病原菌和昆虫的抗性[16][17]。植物促分裂原活化蛋白激酶(mitogen-activated protein kinases,MAPKs)则对各个信号通路行使重要的调控作用[18][19]。拟南芥 EDR1(Enhanced Disease Resistance 1)基因属于MAPK激酶的一员,位于MAPK信号转导途径的顶端,对SA抗病途径具有负调控功能[20]。相反,病程相关基因非表达子 1(NONEXPRESSOR OF PATHOGENESIS-RELATED GENES 1,NPR1)是水杨酸调控途径下游的重要调控因子,同时协调不同植物激素信号通路间的交互效果[13]。研究表明,拟南芥EDR1和NPR1基因在麦类作物中存在同源基因[21],暗示它们可能参与抗病反应,同时也暗示水杨酸是启动麦类作物防御响应的信号分子。
本研究通过构建大麦TILLING筛选体系、鉴定大麦EDR1和NPR1基因突变体,探索开展麦类作物功能基因组学的新思路。
2 材料和方法
2.1 植物材料
本实验采用六棱春性裸粒大麦(H.vulgare L.cv.‘Tamalpais’)为原始材料创建突变群体。CEL I内切酶的提取使用山东省泰安市当地种植的新鲜旱芹材料。
2.2 实验方法
2.2.1 突变群体的创建 大麦种子经0.3%(v/v)EMS(Sigma-Aldrich,St.Louis,MO,USA)水溶液震荡处理10 h(150 rpm,25℃),随后使用自来水持续冲洗4 h,然后置于通风橱吹干备用。M1种子在温室播种,分单株收获M2种子。M2种子按株系种植,分别播种4~8粒、从中保留1个M2健壮植株,用以创建TILLING群体。
2.2.2 CELⅠ酶切筛选体系的建立 实验利用小麦开花基因FT的两个质粒(单碱基差异)作为PCR扩增模板,PCR产物经过CELⅠ酶切和凝胶电泳(琼脂糖或聚丙烯酰胺)分离,建立筛选体系。模板质粒包括FT(12-1)和FT(12-6),扩增产物长 1427 bp,酶切产生370 bp和1056 bp的片段。扩增引物、差异碱基和省略片段大小如下:
FT(12-1):CACCTCGTACCCTAGCTAGC|350bp|G|1033bp|GTGATAAGAAGCCAGCCACGAAT
FT(12-6):CACCTCGTACCCTAGCTAGC|350bp|T|1033bp|GTGATAAGAAGCCAGCCACGAAT
PCR采用25 μL反应体系,包括1×Reaction Buffer(Promega公司Colorless GoTaqReaction Buffer,catalog#M7921),dNTPs(TaKaRa公司)200 μM,扩增引物各25 pmol,模板 5 ~300 ng,Taq 酶 5U。PCR 反应步骤包括:预变性94℃ 5 min,40个扩增循环(94℃ 30 s,52.4℃ 30 s,72℃ 1.5 min),终延伸72℃8 min,退火预变性99℃ 10 min,80℃ 20 s,95个退火循环(80℃ 7 s,每循环温度递减0.3℃),4℃保存。
CELⅠ内切酶的提取参照Till等人[4]的方法,所有操作在4℃进行。CELⅠ酶切和酶切产物分离参照Uauy 等人[10]的方法。其中,CELⅠ酶切体系20 μL,含有切割缓冲液(20 mM HEPES pH 7.5,10 mM KCl和3 mM MgCl2)2 μL ,CELⅠ酶1 μL ,PCR 产物14 μL ,双蒸水3 μL 。在45 ℃条件下,酶切30 min,然后加入反应终止液(75 mM EDTA pH 8.0)5 μL,在45℃条件下放置2 min。实验比较分析CELⅠ浓度、酶切温度和酶切时间等因素对酶切效果的影响,用以优化酶切体系。
2.2.3 大麦突变体的筛选 实验围绕拟南芥水杨酸抗病信号通路,选择了上下游2个关键途径基因EDR1(GenBank:AF305913)和 NPR1(GenBank:ATU76707)开展工作。以上2基因在大麦上保守存在(EDR1:AF305912;NPR1:AM050559)。利用公共数据库,比如 NCBI(http://www.ncbi.nlm.nih.gov/)和HarvEST(http://www.harvest- web.org/),获得了大麦基因的完整信息。利用 Primer3(http://frodo.wi.mit.edu/primer3/)进行PCR引物设计。通过PCR扩增、CELⅠ酶切和非变性聚丙烯酰胺凝胶电泳鉴定携带目的基因突变的DNA混合池(本实验2154M2株系,每4个 M2株系混合成池,共形成534个DNA混合池),进而检测M2个体和野生亲本DNA混合池扩增产物的酶切效果、确定突变株系。对来自突变个体的PCR扩增产物测序鉴定突变位点,分析其可能的突变效应。有关操作步骤参照Uauy等人[10]的方法。大麦EDR1和NPR1基因的扩增反应的退火温度分别为63.1℃ 和64.7℃,扩增引物和中间片段大小如下:
EDR1(AF305912):CGAAGTGGCTACCTTTGGAC|1606bp|CACCTATCGAGCCGAACCTA
NPR1(AM050559):GAACTAGATCCCCACCACCA|1352bp|GTGGTGTTTTGCATGTGGAGT
3 结果与分析
3.1 大麦TILLING群体的创建
根据我们使用EMS处理二倍体小麦的经验,选用了0.3%(v/v)EMS处理大麦品种‘Tamalpais’。人工气候室发芽一周显示,经EMS处理的种子平均发芽率63.9%,未处理对照的发芽率为73.0%;然而幼苗生长状态差异明显,其中EMS处理对应的幼苗,生长正常、高于3公分的幼苗占15.3%,而未处理对照正常苗占98.6%。温室条件下,大约14.3% 的M1个体表现叶片色变(图1A)。在M2世代,可以看到株高(图1B)、株型(图1C)等方面的表型突变(图1BC)。实验共收获2154个M2株系,每4个 M2株系合并成单个DNA池,共形成534个DNA混合池。

图1 大麦突变体:Fig.1 Barley mutants
3.2 TILLING筛选体系的优化
本实验中CEL I内切酶自山东省泰安市当地的旱芹中提取,有必要建立相应的CELⅠ酶切体系。为此,实验首先利用携带单碱基差异的FT基因质粒(1:1混合)测试了酶切温度和酶切时间对酶切的影响。采用30 min的酶切时间,温度梯度实验(图2A)显示,温度低于31.8℃时,酶切特征条带(370 bp和1056 bp)不明显,说明在此温度下CELⅠ内切酶活性低,不能有效切割错配(mismatch)杂合双链。温度从33.7℃上升到49.7℃,CELⅠ表现活性增加,可以明显观察到酶切特征条带;随着酶切活性增加,泳道背景信号放大(smear现象)。实验证明,酶切温度介于43.9℃到46.8℃范围内较为合适。在酶切时间方面,我们设定了30到180 min的范围(图2B)。采用45℃的酶切温度,结果证明,30 min的酶切就能获得清晰的酶切特征条带,随着时间延长,酶切特征条带增强,但泳道背景信号也逐渐放大。因此,酶切时间在30到75 min较为合适。另外,我们还发现CELⅠ粗提产物的用量、酶切缓冲液(Nicking Buffer)组分等对CELⅠ酶切效果同样有影响。对于不同批次提取的CELⅠ内切酶,需要对最佳使用浓度进行优化。在此基础上,建立了有效的酶切体系(酶切温度45℃,酶切时间30 min,酶切缓冲液(20 mM HEPES pH 7.5,10 mM KCl和 3 mM MgCl2)2 μL。
为验证本实验TILLING检测体系的灵敏度,我们利用携带单碱基差异的FT基因质粒作为模板,按1∶1到1∶15比例混合,经PCR扩增和CELⅠ酶切发现,1∶1到1∶9的混合模板均可以检测到清晰的酶切特征条带;而1∶10到1∶15混合范围内,酶切特征条带相对模糊(图2C)。因为大麦为二倍体,1∶3、1∶5、1∶7和1∶9的质粒比例相当于单个大麦杂合突变个体分别和1、2、3和4个野生型大麦个体等量混合。由此可见,4株 M2个体的DNA混合池可以保证检测到突变基因。
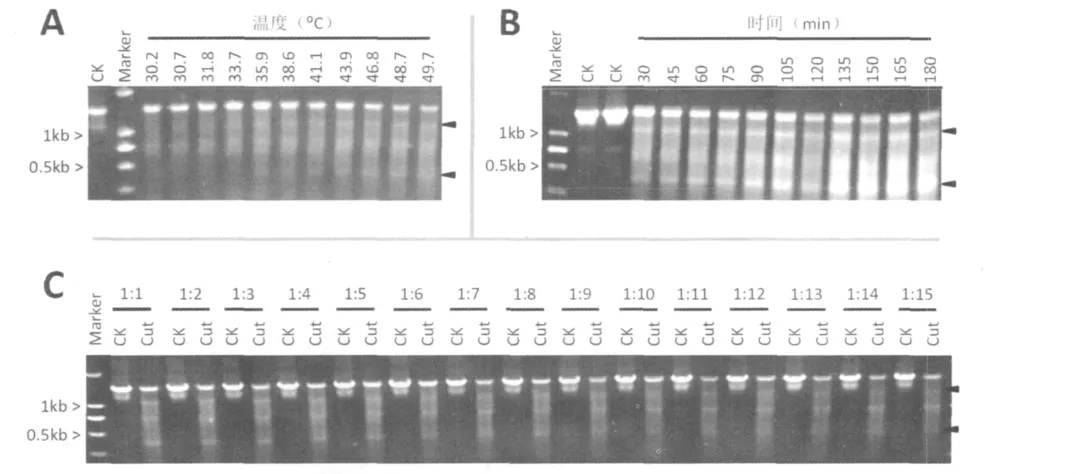
图2 TILLING筛选体系的优化Fig.2 Optimization of TILLING screening system
3.3 大麦EDR1和NPR1基因突变体筛选
本实验利用优化的CELⅠ酶切体系,对建成的大麦EMS诱变群体开展了突变体筛选。实验针对水杨酸信号通路相关的2个重要基因EDR1和NPR1。CELⅠ酶切显示(图3AB),现有的2154个M2大麦株系中,鉴定出5个目标基因的突变体,其中EDR1突变体3个(210-2、255-3、261-4)、NPR1突变体2个(340-4、360-1)。序列分析证明,EDR1和NPR1基因各有1个突变发生在内含子区域(255-3、340-4),分别涉及T→C和G→A转换(Transition)。另外3个突变发生在外显子编码区,EDR1基因的2个转换C1051T和C1666T分别导致His351Tyr和Pro556Ser突变(图3CD),而NPR1基因的G792A转换则不改变Arg编码(图3E)。
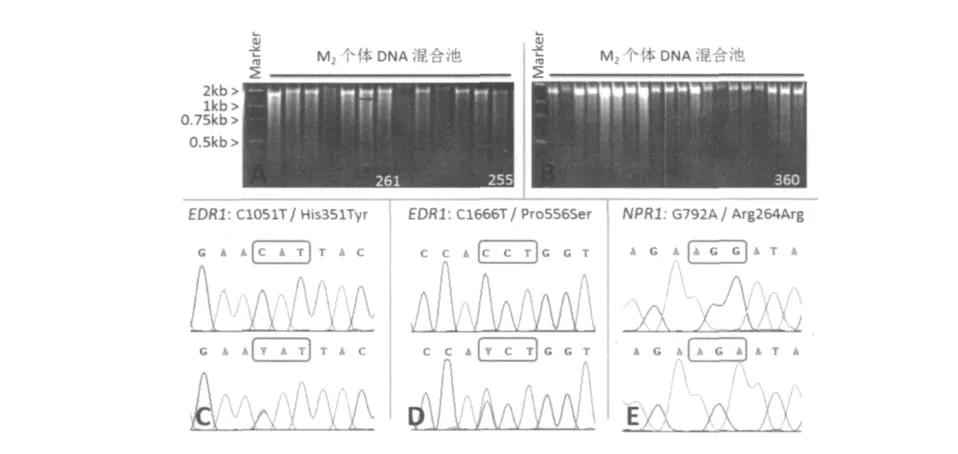
图3 大麦EDR1和NPR1基因突变体筛选Fig.3 Screening of EDR1 and NPR1 mutants in Barley population
4 讨论
4.1 TILLING技术在麦类作物功能基因组研究中的应用
麦类作物是世界上重要的粮食作物,其功能基因组研究对全球粮食安全具有重要意义。然而,麦类作物对遗传转化的排斥(recalcitration)严重阻碍着相关功能基因组研究。TILLING是近年发展起来的重要的反向遗传学研究工具,在动植物研究中都有应用[4]。麦类作物方面,TILLING技术已成功应用于小麦[9][10]和大麦[11]。Slade等人[9]从1920 个小麦突变个体中筛选到 246 个影响直链淀粉合成的 WAXY 基因突变,提供了丰富的等位基因资源。Uauy等人[10]也创建了多倍体小麦的TILLING群体,已成功用于研究小麦条锈病抗性基因 WKS1[22]和淀粉分支酶基因 SBEIIa和SBEIIb。在大麦上,德国学者Gottwald等[11]创建了二棱大麦‘Barke’的TILLING群体,被成功用来研究决定大麦穗形的homeobox基因HvHox1。该大麦突变群体在M2和M3世代呈现丰富的形态变异,同时为开展正向遗传学研究提供了宝贵资源。在我国,尚没有大麦TILLING群体的报道。本研究构建了六棱大麦‘Tamalpais’的EMS突变群体。本研究为我国麦类作物反向遗传学研究奠定了基础。
4.2 水杨酸信号通路相关基因突变体的获得
在模式植物拟南芥上,水杨酸介导的防卫信号途径在抵御活体寄生的病原菌和病毒抗性方面起着重要作用[14][16]。尽管水杨酸被证明是双子叶植物中的重要防御信号,他们在单子叶植物特别是麦类作物中的作用尚不明确。水杨酸或其类似物INA或BTH可以诱导小麦[23][24]和大麦[25][26]中病程相关蛋白基因(pathogenesis-related,PR)的表达或提高抗性。拟南芥AtNPR1基因是水杨酸信号通路中的重要调控元件[27]。AtNPR1基因在小麦中的过量表达增强小麦对赤霉病菌的抗性[28]。因此,水杨酸信号通路在麦类作物抗病方面或许行使重要功能。
EDR1和NPR1是拟南芥水杨酸信号通路中的两个重要基因[13][20]。在麦类作物上,EDR1和NPR1基因保守存在,但二者的具体功能还没有深入的研究。本研究获得了EDR1和NPR1基因的5个突变体,其中2个EDR1突变导致氨基酸转换(His351Tyr,Pro556Ser),其他3个突变发生在内含子或不影响氨基酸编码。鉴于His351Tyr和Pro556Ser突变位于EDR1蛋白的非保守区段,很难预测此突变的功效,我们将研究突变体对大麦锈病和白粉病等生物胁迫的反应,进而推断大麦EDR1基因的功能。
4.3 群体突变频率
植物倍性不同,对突变的承载能力各异。在麦类作物上,小麦具有较强的耐突变能力,现有的六倍体小麦TILLING群体平均每24或38 kb一个点突变;相应的四倍体小麦 TILLING群体平均每40或51 kb一个点突变[9][10]。按照六倍体和四倍体小麦平均每38和51 kb一个点突变,对应1.3 kb的目标片段,从1536个突变个体中可获得52个六倍体或39个四倍体目标基因突变体,含有敲除(knockout)突变的可能性在80%以上[10]。大麦是二倍体,TILLING群体的突变频率相对较低,大约每500 kb一个点突变[11]。
本项研究中,EDR1和NPR1基因的筛选目标片段分别为1.6 kb和1.4 kb,因为本实验采用常规聚丙烯酰氨凝胶分离CELⅠ酶切产物,片段两端0.25 kb内的错配将超出检测范围,因此两基因筛选的有效目标片段累计2 kb。现有群体包括2154个M2株系,根据获得的5个突变体推断现有群体平均862 kb一个点突变。同Gottwald等[11]报道的每500kb一个点突变相比,本研究中获得的EDR1和NPR1基因的突变体数目偏少,这可能主要是由于引物的扩增效率有关,导致部分突变体漏选。
5 结论
本研究创建了大麦品种‘Tamalpais’的TILLING筛选群体,并成功用于水杨酸信号通路中重要基因EDR1和NPR1突变体的筛选。本研究在国内领先搭建了大麦反向遗传学研究的平台,为麦类作物功能基因组研究奠定了基础。
[1]WAP World Agricultural Production.United States Department of Agriculture - Foreign Agricultural Service[R].Circular Series WAP 12-10,December 2010
[2]Gilchrist EJ,Haughn G W.TILLING without a plough:a new method with applications for reverse genetics[J].Current Opinion in Plant Biology,2005.8:1 -5
[3]Till BJ.Comai L,Henikoff,S.TILLING and ECOTILLING for Crop Improvement[C].In:Varshney RK and Tuberosa R.Genomics - Assisted Crop Improvement.Dordrecht.Springer.2007,333 -349
[4]Till BJ,Zerr T,Comai L,et al.A protocol for TILLING and Ecotilling in plants and animals[J].Nature Protocols,2006,1:2465 - 2477
[5]Tadele Z,MBAC,Till BJ.TILLING for mutations in model plants and crops[C].In:Jain SM and Brar DS.Molecular Techniques in Crop Improvement,Dordrecht.Springr.2010,307 - 332
[6]Bentley A,MacLennan B,Calvo J,et al.Targeted recovery of mutations in Drosophila[J].Genetics,2000,156:1169 -1173
[7]McCallum CM,Comai L,Greene EA,et al.Targeted screening for induced mutations[J].Nature Biotechnology,2000,18:455 - 457
[8]Till BJ.,Cooper J,Tai TH,et al.Discovery of chemically induced mutations in rice by TILLING[J].BMC Plant Biology,2007b,7:19
[9]Slade A J,Fuerstenberg SI,Loeffler D,et al.A reverse genetic,nontransgenic approach to wheat crop improvement by TILLING[J].Nature Biotechnology,2004.23:75 - 81
[10]Uauy C,Paraiso F,Colasuonno P,et al.A modified TILLING approach to detect induced mutations in tetraploid and hexaploid wheat[J].BMC Plant Biology,2009,9:115
[11]Gottwald S,Bauer P,Komatsuda T.TILLING in the two - rowed barley cultivar'Barke'reveals preferred sites of functional diversity in the gene HvHox1 [J].BMC Research Notes,2009,2:258
[12]Bari R,Jones JDG.Role of plant hormones in plant defence responses[J].Plant Molecular Biology,2009,69:473-488
[13]Pieterse CMJ,Leon - Reyes A,Ent SVd,et al.Networking by small-molecule hormones in plant immunity[J].Nature Chemical Biology,2009,5:308 - 316
[14]Vlot AC,Dempsey DMA,Klessig DF.Salicylic acid,a multifaceted hormone to combat disease[J].Annual Review of Phytopathology,2009.47:177-206
[15]Verhage A,Wees SCMV,Pieterse CMJ,et al.Plant immunity:It's the hormones talking,but what do they say[J].Plant Physiology,2010,154:536-540
[16]Glazebrook J.Contrasting mechanisms of defense against biotrophic and necrotrophic pathogens[J].Annu.Rev.Phytopathol,2005,43:205-227
[17]Howe GA,Jander G.Plant immunity to insect herbivores[J].Annual Review of Plant Biology,2008,59:41 -66
[18]Asai T,Tena G,Plotnikova J,et al.MAP kinase signalling cascade in Arabidopsis innate immunity[J].Nature,2002,415:977 -983
[19]Brodersen P,Petersen M,Nielsen HB.Arabidopsis MAP kinase 4 regulates salicylic acid- and jasmonic acid/ethylene-dependent responses via EDS1 and PAD4 [J].Plant Journal,2006,47:532 -546
[20]Frye CA,Tang D,Innes RW.Negative regulation of defense responses in plants by a conserved MAPKK kinase[J].Proc Natl Acad Sci USA,2001,98:373 -378
[21]Niu J,Zhang L,Hong D,et al.Cloning,characterization and expression of wheat EDR1(Enhanced Disease Resistance)gene[J].Journal of Plant Physiology and Molecular Biology,2005,31:477 -484
[22]Fu D,Uauy C,Distelfeld A,et al.A Kinase -START Gene Confers Temperature-Dependent Resistance to Wheat Stripe Rust[J].Science,2009,323,1357
[23]Anand A,Schmelz EA,Muthukrishnan S.Development of a lesion-mimic phenotype in a transgenic wheat line overexpressing genes for pathogenesis-related(PR)proteins is dependent on salicylic acid concentration[J].Mol Plant Microbe Interact,2003,16:916 -925
[24]G rlach J,Volrath S,Knauf-Beiter G.A novel class of inducers of systemic acquired resistance activates gene expression and disease resistance in wheat[J].Plant Cell,1996.8:629 - 643
[25]Kogel KH,Beckhove U,Dreschers J,et al.Acquired resistance in barley(The resistance mechanism induced by 2,6 -Dichloroisonicotinic acid is a phenocopy of a genetically based mechanism governing race-specific powdery mildew resistance) [J].Plant Physiology,1994,106:1269-1277
[26]Muradov A,Petrasovits L,Davidson A,et al.A cDNA clone for a pathogenesis-related protein 1 from barley[J].Plant Mol Biol,1993,23:439-442
[27]Dong X.NPR1,all things considered[J].Current Opinion in Plant Biology,2004,7:547-552
[28]Makandar R,Essig JS,Schapaugh MA,et al.Genetically engineered resistance to Fusarium Head Blight in wheat by expression of Arabidopsis NPR1 [J].Mol Plant Microbe Interact,2006,19:123 -129
