福建及贵州等地烟草青枯菌系统发育分析
2012-02-28潘哲超许景升陈顺辉
潘哲超, 徐 进, 顾 钢, 吴 畏,许景升, 陈顺辉, 冯 洁*
(1.中国农业科学院植物保护研究所,植物病虫害生物学国家重点实验室,北京 100193;2.云南省农业科学院经济作物研究所,昆明 650205; 3.福建省烟草农业科学研究所,福州 350003;4.西南大学植物保护学院,重庆 400715)
烟草青枯病是由茄科雷尔氏菌[Ralstonia sol anacear u m (Smit h)Yabuuchi et al.]引起的烟草生产上最具毁灭性的病害之一。该病1880年发现于美国的北卡罗来纳州,1940年前后,该病于美国和印度尼西亚猖獗危害,此后,又逐渐成为日本、澳大利亚、韩国等许多产烟国家烟草上重要的细菌病害[1]。烟草青枯病在我国长江流域及其以南烟区普遍发生,其中以广东、福建、湖南、四川及贵州烟区危害最为严重,个别年份的暴发流行可造成毁灭性损失[2]。近些年来,该病逐渐有扩展蔓延至北方烟区的趋势,山东、河南、陕西及辽宁等省均有该病发生的报道,且局部地区危害较重[3]。
传统的分类框架根据青枯菌的寄主范围或对3种双糖和3种己醇的氧化利用情况,将其划分为5个不同的生理小种(race)或5个生化变种(biovar)[4-7]。烟草青枯菌包括青枯菌1号生理小种的生化变种Ⅰ、Ⅲ和Ⅳ(即R1/Bv1、R1/Bv3和R1/Bv4菌株)。
2005年,Fegan和Prior[8]提出了青枯菌演化型分类框架(phylotype classification shemes),青枯菌被划分为与地理起源密切相关的4个演化型—即演化型Ⅰ、Ⅱ、Ⅲ和Ⅳ型,在地理起源上依次对应于亚洲分支、美洲分支、非洲分支以及印尼分支;每个演化型又由诸多序列变种(sequevar)构成。序列变种定义为:以内切葡聚糖酶基因(endogl ucanase,egl)进行系统发育分析时,一组序列高度保守的菌株集合体。演化型分类框架是建立在青枯菌基因组长期缓慢累积而形成的遗传变异的基础上,与传统的小种及生化变种分类方法相比,演化型的分类框架可以更精确地反映青枯菌这一复合种的地理起源及种内的遗传多样性。国际上的研究表明无毒基因avr A是控制青枯菌对烟草致病能力的决定因素之一,其于青枯菌群体中的分布也存在较大的差异性[9],亚洲分支青枯菌无毒基因avr A存在RS1000类型和GMI1000类型的两种类型[10]。
国内目前对烟草青枯病菌系的研究大都迟滞于基于传统方法的分类框架上[11-17],仅郑向华等[18]采用RAPD对广东省烟草青枯菌的遗传多样性进行了研究。本研究采用国际上最新的演化型分类框架,对福建及贵州部分地区采集的烟草青枯病菌株进行系统发育分析并进行序列变种鉴定,并对各序列变种的无毒基因avr A进行了序列类型的分析,为我国烟草青枯病的防控工作及烟草的抗病育种提供一定的基础。
1 材料与方法
1.1 供试菌株
烟草青枯菌株由本实验室采集分离。供试62个烟草青枯菌株和15个参考菌株见表1。

表1 供试烟草青枯菌株和参考菌株1)
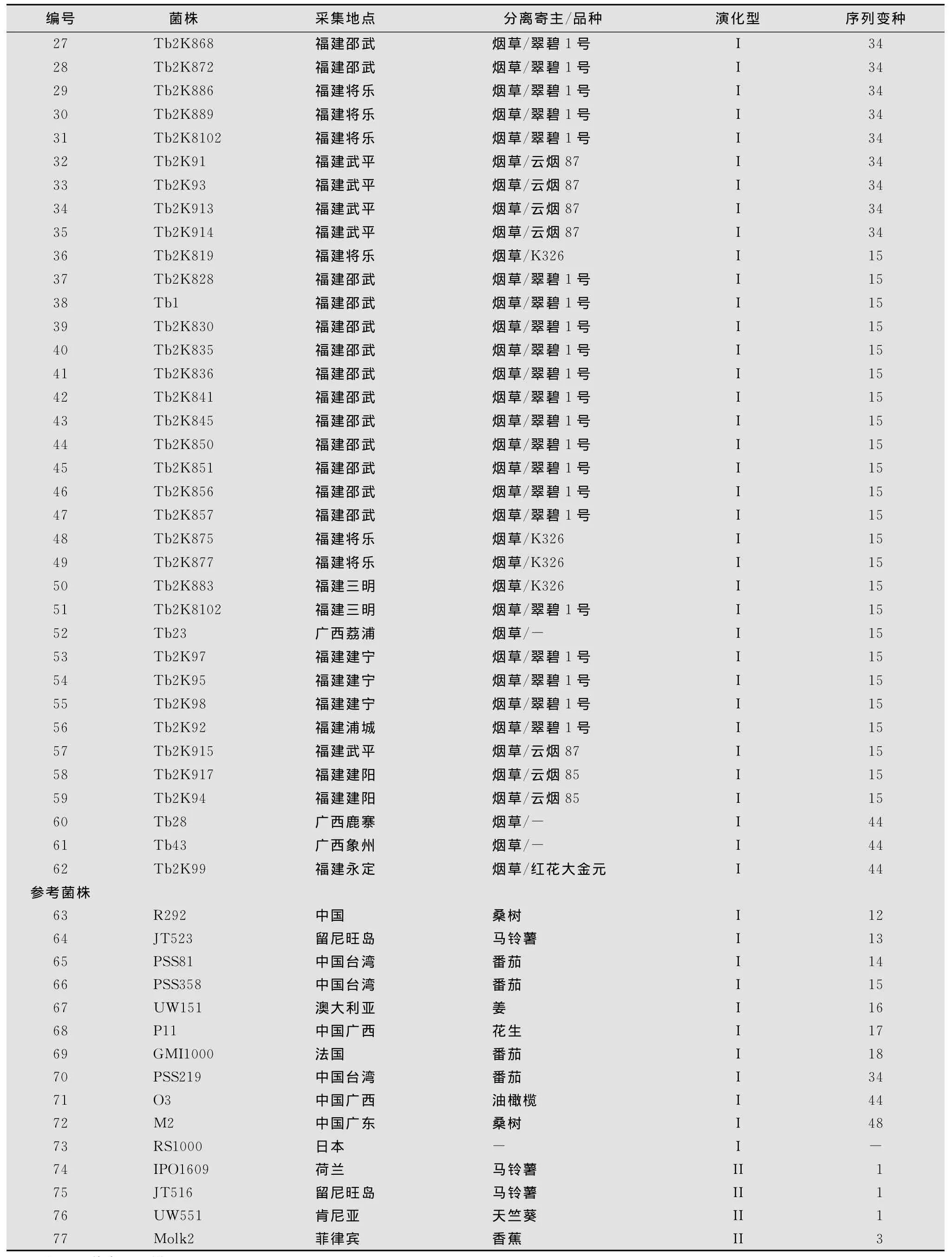
续表1
1.2 供试菌株egl基因的扩增
用 于 扩 增egl 的 引 物 为:Endo-F (5′-ATGCATGCCGCTGGTCGCCGC-3′) 和 Endo-R (5′-GCGTTGCCCGGCACGAACACC-3′)[19]。 所 用 引物由上海生工生物公司合成。
PCR反应体系组分(25μL)包含1.5 mmol/L MgCl2,250μmol/L d NTPs,50 mmol/L KCl,10 mmol/L Tris-HCl及1.25 U Taq DNA聚合酶,引物各4 p mol,50 ng模板DNA。
PCR反应程序:96℃预变性9 min;95℃变性1 min,64℃复性1 min,72℃延伸2 min,30个循环;最后72℃延伸10 min,4℃保存[19]。PCR扩增产物回收后交由上海生工公司测序。
1.3 avr A基因的扩增
用于扩增avr A的引物寡核苷酸序列为:avr AA1(5′-TGTAAAACGACGGCCAGTATGAGAAGAATYG GMAA-3′)和 avr AB1(5′-CAGGAAACAGCTATGACCTCGCTRTCGCTATCGYT-3′)[20]。
PCR反应程序:94℃预变性3 min,94℃变性1 min,58℃复性1.5 min,72℃延伸1 min,30个循环;最后72℃延伸10 min,4℃保存[20]。
1.4 数据分析方法
测序结果经Bio Edit[21]软件编辑后应用Clustal W 软件[22]对egl序列比对,用 MEGA4.1软件[23]进行系统发育分析,采用模型邻接法(NJ)构建系统发育树[24],自举分析为1 000次重复抽样检验得到的自举置信度。从Gen Bank选取12个已确定序列变种归属的参考菌株egl序列作为参考,以确定参试菌株在系统发育树中的位置;同时选取各序列变种代表性菌株及5个已确定avr A序列的参考菌株进行avr A基因氨基酸序列比对。
2 结果与分析
2.1 egl基因系统发育学的分析结果
参试烟草青枯病菌株的egl基因序列分布于亚洲分支以下的4个亚组内(图1)。采用已知序列变种的标准菌株序列作为参照,4个亚组分别被鉴定为序列变种15、17、34和44。在该4个序列变种中,序列变种15所包含的菌株数在全部供试菌株中所占比例最高,为38.7%。序列变种17略次之,为37.1%。序列变种34再次之,占19.4%。序列变种44最低,仅占4.9%。
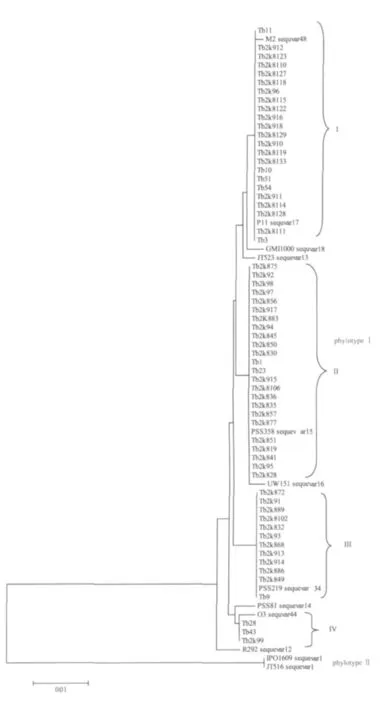
图1 基于egl基因的系统发育分析
序列变种15的标准菌株为我国台湾的番茄青枯病菌株PSS358,该序列变种包括了24个来源于福建和广西的分离菌株,寄主品种分别为‘K326’、‘翠碧1号’、‘云烟85’以及‘云烟87’;以来源于广西花生青枯病菌株P11作为标准菌株的序列变种17,包括23个分离自福建、广西和贵州的菌株,分离寄主品种分别为‘K326’、‘翠碧1号’和‘红花大金元’。其中分离自贵州的烟草青枯病菌株全部集中于该序列变种内。
序列变种34的标准菌株为我国台湾番茄青枯病菌株PSS219,该序列变种包括的参试菌株数目较少,且12个菌株全部分离自福建,分离品种为‘翠碧1号’和‘云烟87’;序列变种44仅包括3个分离菌株,其标准菌株为广东油橄榄青枯病菌株O3。
2.2 avr A基因氨基酸序列比对结果
对各序列变种代表性菌株Tb2K91(序列变种34)、Tb2 K94(序列变种15)、Tb2 K99(序列变种44)和Tb2K918(序列变种17)及5个已确定avr A序列的参考菌株GMI1000、IPO1609、UW551、MOLK2和RS1000进行了氨基酸比对,结果表明4个序列变种代表菌株与亚洲菌株RS1000的序列一致,且4个序列变种代表菌株的avr A氨基酸序列并不存在差异(图2)。

图2 基于avr A基因的氨基酸序列比对
3 讨论
青枯菌寄主范围极为广泛,可侵染54个科的450余种植物[25];不同地理起源的青枯菌在与其寄主长期协同进化的过程中,演化出明显的生理分化或菌系多样性。以致美国科学家Buddenhagen在文章中如是写道:“有如此之多的青枯病,和如此之多的青枯菌”[26]。
Xu等基于演化型分类框架,对国内13个省份、17种不同寄主植物上的286个青枯菌株材料的研究结果表明:中国青枯菌种内具有丰富的遗传多样性,存在亚洲分支的10个序列变种及1个美洲分支的序列变种[27]。在此基础上,本研究采用该框架对福建、贵州等地区62个烟草青枯病菌株进行了系统发育分析,结果表明:62个参试烟草青枯病菌株全部归属于亚洲分支的4个已知序列变种,即序列变种15、17、34和44;未发现归属于美洲或非洲分支的烟草青枯病菌株(即传统上的R1/Bv1菌株)。其中序列变种15和17为优势菌系,占全部参试菌株数的75.8%。值得注意的是,作为新鉴定出的序列变种,以我国台湾番茄青枯病菌株PSS219为标准菌株的序列变种34虽包含的参试菌株数目较少,但无一例外均系分离自福建省的菌株;同时,分离于贵州的菌株全部归属于序列变种17。但由于本研究采集的菌株数目有限,这种序列变种与国内省份或地区之间的对应关系尚需进一步研究验证。
某些狭窄寄主范围的菌株归属于特定的序列变种。如来源于香蕉的2号小种菌株属于序列变种3、4、6、24;来源于马铃薯的3号小种菌株属于序列变种1、2,来源于姜的4号小种菌株属于序列变种16,来源于桑的5号小种的菌株属于序列变种12、44、48。烟草青枯菌虽属于广泛寄主的生理小种1,但其在致病性上具有一定的特殊性。如1975年,烟草青枯病成为美国南、北卡罗来纳州烟草生产上最主要的病害;但毗邻的佐治亚和佛罗里达州却鲜有该病发生危害的报道,尽管在这些地区番茄青枯病极为普遍[20]。何礼远对中国青枯菌致病性测定的研究结果表明:绝大多数非烟草寄主的青枯菌株不能对烟草致病;但烟草菌株Tb2不仅对烟草致病,还对茄科、花生和辣椒等其他寄主植物具不同程度的致病能力[28]。本研究表明福建、贵州等地区烟草青枯菌存在于序列变种15、17、34和44,但是否仅存于这4个序列变种尚需今后更多的烟草菌株来验证。
本研究进一步对采集的烟草青枯病菌4个序列变种的代表性菌株进行了氨基酸比对分析。结果表明,4个序列变种代表菌株的avr A氨基酸序列并不存在差异,都属于日本菌株RS1000类型而非GMI1000类型。但因为这两种类型的无毒基因avr A读码框是一致的,所以无毒基因avr A类型与青枯菌株对烟草致病力之间是否存在相关性尚需明确。
作为青枯病防控策略的首选措施之一,抗病育种一直以来备受各国研究者的青睐。但由于青枯菌群体所特有的高度适应性及变异性,又成为长期困扰抗病育种研究人员的难题。故而,成功培育抗性品种的先决条件是揭示出青枯菌这一复杂群体内部丰富的遗传多样性[29]。国际上以美国为代表的烟草抗青枯病育种工作,主要是针对演化型Ⅱ型(美洲分支)烟草青枯病菌株开展的。本研究的结果揭示出国内对烟草致病的菌株均系演化型Ⅰ型菌株(亚洲分支),因此亟需评价国内烟草青枯病菌4个序列变种对现有烟草抗病品种的毒性水平差异,藉以指导现有品种的合理布局;同时,根据我国烟草青枯菌的遗传背景选育新的抗病品种。
[1] 朱贤朝,王彦亭,王智发.中国烟草病害[M].北京:中国农业出版社,2002.
[2] 陈瑞泰,朱贤朝,王智发,等.全国16个主产烟省(区)烟草侵染性病害调研报告[J].中国烟草科学,1997(4):1-7.
[3] 孔凡玉.烟草青枯病的综合防治[J].烟草科技,2003(4):42-48.
[4] Buddenhagen I,Sequeira L,Kel man A.Designation of races in Pseudomonas sol anacear u m [J]. Phytopat hology,1962,52:726.
[5] He L Y,Sequeira L,Kel man A.Characteristics of strains of Pseudomonas sol anacear u m [J].Plant Disease,1983,67:1357-1361.
[6] Pegg K G,Moffett M.Host range of the ginger strain of Pseudomonas sol anacear u m in Queensland[J].Australian Jour nal of Experi mental Agriculture and Ani mal Husbandry,1971,11:696-698.
[7] Hay ward A C.Characteristics of Pseudomonas sol anacear u m[J].Jour nal of Applied Bacteriology,1964,27:265-277.
[8] Fegan M,Prior P.How complex is the“Ralstonia solanacear um species complex”[M]∥Allen C,Prior P,Hay ward A C.Bacterial wilt disease and the Ralstonia solanacear um species complex.St.Paul:APS,2005:449-462.
[9] Pouey miro M,Cunnac S,Barberis P,et al.Two typeⅢsecretion syste m effectors fro m Ralstonia sol anacear u m GMI1000 deter mine host-range specificity on tobacco[J]. Molecular Plant-Microbe Interact,2009,22(5):538-550.
[10]Liu Y,Kanda A,Kiba A,et al.Distribution of avir ulence genes avr A and pop P1 in 22 Japanese phylotype I strains of Ralstonia sol anacear um[J].Journal of General Plant Pathology,2009,75(5):362-368.
[11]邓正平,匡传富,周志成,等.湖南烟草青枯病菌生理小种测定[J].湖南农业大学学报,2004,30(1):47-49.
[12]顾江涛,许大凤,李英,等.安徽皖南烟区青枯病病原菌生化型研究[J].中国烟草科学,2008,29(3):60-61.
[13]黄福新,陈永惠,华静月,等.广西草青枯菌系及其主要生理特性研究[J].植物保护学报,1998,25(3):240-244.
[14]刘琼光,陈泽鹏,董春,等.广东烟草青枯病菌菌系研究[J].中国烟草学报,1999,5(4):25-27.
[15]张建华,郑继法,陈瑞泰,等.我国南方主要烟区烟草青枯病菌的生物型及产细菌特性的研究[J].烟草学刊,1990(2):85-93.
[16]邹阳,肖崇刚,易龙.重庆地区烟草青枯病菌的生物型测定[J].烟草科技,2007(6):62-64.
[17]邹阳,肖崇刚.重庆地区烟草青枯菌生理小种的初步鉴定[J].烟草科技,2008(5):60-64.
[18]郑向华,邓海滨,刘琼光,等.广东省烟草青枯菌的菌系和遗传多样性[J].华中农业大学学报,2007,26(4):463-468.
[19]Poussier S,Prior P,Luisetti J,et al.Partial sequencing of the hrp B and endoglucanase genes confir ms and expands the known diversity within the Ralstonia solanacearum species complex[J].Systematic and Applied Microbiology,2000,23:479-486.
[20]Robertson A E,Wechter W P,Denny T P,et al.Relationship bet ween avir ulence gene (avr A)diversity in Ralstonia sol anacearu m and bacterial wilt incidence[J].Molecular Plant-Micr obe Interact,2004,17:1376-1384.
[21]Hall T A.Bio Edit:a user-friendly biological sequence alignment editor and analysis pr ogra m for Windows 95/98/NT[C].Nucleic Acids Sy mposiu m Series 41,1999.
[22]Tho mpson J D,Higgins D G,Gibson T J.CL USTAL W:i mpr oving the sensitivity of progressive multiple sequence alignment t hr ough sequence weighting,positions-specific gap penalties and weight matrix choice[J].Nucleic Acids Research,1994,22:4673-4680.
[23]Ku mar S,Ta mura K,Nei M.MEGA3:integrated soft ware for molecular evol utionary genetics analysis and sequence alignment[J].Brief Bioinfor m,2004,5:150-163.
[24]Jukes T H,Cantor C R.Evol ution of pr otein molecules[M]∥Munro H N.Mammalian protein metabolism.New Yor k,USA:Academic Press,1969:121-132.
[25]Wicker E,Grassart L,Coranson-Beaudu R,et al.Ralstonia sol anacear u m Strains fro m Martinique(French West Indies)Exhibiting a New Pathogenic Potential[J].Applied and Envir on mental Microbiology,2007,71:6790-6801.
[26]Buddenhagen I W.Bacterial wilt revisited[R]∥Persley G.Bacterial wilt disease in Asia and the South Pacific:proceedings of an international workshop held at PCARRD,Los Banos,Philippines,8-10 October 1985,Canberra:ACIAR,1986.
[27]Xu J,Pan Z C,Prior P,et al.Genetic diversity of Ralstonia sol anacear u m strains from China[J].Eur opean Jour nal of Plant Pathology,2009,186:6186-6197.
[28]He L Y,Sequeira L,Kel man A.Characteristics of strains of Pseudomonas solanacear u m fro m China[J].Plant Disease,1983,67:1357-1361.
[29]Gillings M R,Fahy P.Genomic fingerprinting:towards a unified view of the Pseudomonas sol anacear u m species co mplex[M]∥Hay ward A C,Hart man G L.Bacterial wilt:the disease and its causative agent,Pseudomonas solanacear u m,Wallingford,United Kingdom:CAB International,Wallingfor d,1994:95-112.
