海湾扇贝组织蛋白酶L基因编码区的克隆和分析
2011-12-28李娟李莉张国范
李娟,李莉,张国范
(1.中国科学院海洋研究所,山东 青岛 266071;2.中国科学院研究生院,北京 100049)
海湾扇贝组织蛋白酶L基因编码区的克隆和分析
李娟1,2,李莉1,张国范1
(1.中国科学院海洋研究所,山东 青岛 266071;2.中国科学院研究生院,北京 100049)
通过cDNA末端快速扩增技术(RACE), 从海湾扇贝(Argopecten irradians)中克隆得到了组织蛋白酶L基因(AiCL)的编码区全
长,为1 095 bp, 推测编码364个氨基酸。经比对与分析发现蛋白序列中存在4个组织蛋白酶L活性位点保守氨基酸:Q164, C170, H309, N329;6个极为保守的半胱氨酸残基:C167,C201,C210,C243,C302,C351。预测其N端17个氨基酸为信号肽序列,C端ASYPTV可能也是分泌信号。AiCL成熟蛋白分子由219个氨基酸构成,前体肽的切割位点预计在A145和M146之间。序列同源性分析中,AiCL蛋白序列与软体动物同源蛋白最为相似,序列一致性在50%以上,在系统进化树中与其它无脊椎动物组织蛋白酶L聚合到一起。通过SWISS-MODEL构建的AiCL成熟蛋白三维模型表明该蛋白空间结构高度保守。根据其序列特征,推测AiCL可能具有水解多种肌蛋白的活性。
海湾扇贝;组织蛋白酶L基因;克隆;序列分析
组织蛋白酶(cathepsin)是一类主要存在于溶酶体的蛋白水解酶,与核糖核酸酶、β-葡萄糖醛酸酶、乙酰基转移酶以及其他的蛋白酶一起参与溶酶体介导的蛋白质的消化及转化过程[1]。“组织蛋白酶”一词1920年被首次使用,它们几乎存在于所有生物,包括病毒、细菌、植物、动物。这一酶家族包括半胱氨酸蛋白酶(L型等)、丝氨酸蛋白酶(A型、G型等)以及天冬氨酸蛋白酶(D型、E型等)[2]。研究发现组织蛋白酶L (cathepsin L)能够水解多种蛋白质,包括肌球蛋白、肌动蛋白、细胞溶质蛋白、胶原及弹力蛋白[3]。高等动物中,组织蛋白酶L在胞内和胞外很多重要的生理、病理过程中起作用,被认为参与抗原呈递、组织再生、肿瘤的侵染转移、骨质的吸收溶解、寄生虫感染等过程[4-7]。水生动物中关于组织蛋白酶L的研究还相对较少。在甲壳动物中,该酶在各组织普遍表达,而消化器官中表达水平最高,可能主要消化食物为机体提供营养。在基围虾(Metapenaeus ensis)中还发现其卵母细胞核中存在未活化的组织蛋白酶L酶原,但对其功能并不很清楚[8]。组织蛋白酶在对虾的蜕皮过程中可能起重要的作用,组织蛋白酶L基因也被认为是对虾生长相关基因[9]。而在水产品加工方面,组织蛋白酶L强大的蛋白水解能力是影响鱼糜品质的罪魁祸首[10,11]。目前,贝类中组织蛋白酶L的分子水平研究还鲜见报道。
海湾扇贝(Argopecten irradians)自然分布于美国的东海岸和墨西哥湾沿岸,于1982年首次成功引入我国,之后又多次引种,现已成为我国主要海水养殖贝类之一[12,13]。其扇贝柱的品质与口感可能会受到闭壳肌内残留组织蛋白酶L的影响。本研究通过cDNA末端快速扩增技术(RACE)从海湾扇贝中克隆得到了组织蛋白酶L基因(AiCL)编码区全长,并通过序列分析发现其与许多物种同源序列有相同特征,在进化上十分保守,可能具有水解多种肌蛋白的活性。
1 材料和方法
1.1 海湾扇贝总RNA提取及cDNA合成
取稚贝软体部加液氮冷冻并研磨,使用Trizol(Invitrogen)提取总RNA ,并经1.0%琼脂糖凝胶电泳检测。以2 μg总RNA为模板,加入Oligo dT(10uM) 1 μl,70 °C热变性5 min,冰浴2 min,迅速加入MLV Reaction Buffer 5μl, dNTP(2.5mM) 5μl, RNase inhibitor(TaKaRa)1μl, M-MLV 反转录酶(Promega) 1 μl, 于42 °C反转录1 h, 95 °C 5 min灭活反转录酶。
1.2 AiCL编码区全长的获得
对本实验室所构建的海湾扇贝均一化cDNA文库进行随机大规模测序并比对分析,获得一条注释为组织蛋白酶L的EST,长为663 bp。
1.2.1 AiCL编码区3’端确认 通过DNAssist2.2将已有的AiCL的EST进行编码区分析,发现其具有与Blastx预测为组织蛋白酶L编码框吻合的终止密码子TGA,初步确认该EST具有完整的编码区3’端序列。
1.2.2 AiCL 5’RACE模板制备 用PCR产物纯化试剂盒(TaKaRa)纯化cDNA。以末端转移酶(TdT,TaKaRa)为cDNA加尾:cDNA 10 μl, 5×TdT buffer 5 μl, dCTP(10mM)0.5 μl, BSA buffer(0.1%) 6.25 μl, 于94°C 保温3 min,然后置于冰上,再加入:TdT 酶 1 μl, 37°C保温10 min, 65°C 10 min失活TdT。
1.2.3 AiCL 5’RACE PCR扩增 使用Primer Premier 5.0根据AiCL的EST序列设计两条反向引物:AiCL gsp R1(5’-TGTAAGGGTACTCAGATTC CGACTCAA-3’)和AiCL gsp R2 (5’-CAAGAG CACCAG TTGTACTAAATGACC-3’),用于AiCL 5’RACE PCR扩增。以加尾cDNA为模板,AiCL gsp R1和Oligo(dG) -adaptor (5’-GGCCACGCGTCGAC TAGTACG10-3’)为引物进行第1次PCR,PCR反应程序为:94°C 2min预变性,94 °C 40s, 60°C(-1°C/循环) 50s, 72 °C 2min, 10个循环; 94 °C 40s, 50 °C 50s, 72°C 2min, 25个循环, 最后72°C延伸10 min。将第一次PCR产物稀释20倍,取1 μl作为模板进行巢式PCR,引物为AiCL gsp R2和AP(5’-GGCCACGCGTCGACTAGTAC-3’),PCR反应条件同样采用前面所使用的touchdown PCR程序。
1.2.4 PCR产物的克隆与测序 (1)PCR产物纯化:PCR 产物在1.2%的琼脂糖凝胶中进行电泳,经过凝胶成像系统(VDS)观察照相后切下特异目的条带,用PCR产物凝胶回收试剂盒(Axygen)回收,方法参考试剂盒说明手册,最后将产物溶解于TE中,-20°C保存备用。
(2)连接T载体与转化及阳性克隆筛选:使用 pMD18-T vector(TaKaRa) 16℃过夜连接PCR产物。取10 μl的连接产物转化大肠杆菌DH5α感受态细胞。感受态细胞的制备和转化参照分子克隆实验指南(第三版)的方法进行。在含有Amp、IPTG和X-gal的LB平板上培养过夜后挑出白色菌落,筛选阳性克隆。将含阳性质粒的单菌落扩大培养,进行测序。
1.3 AiCL的结构和特性分析
所测得的序列去掉载体序列后进行拼接,进行BLAST(http://www.ncbi.nlm.nih.gov)比对,并通过NCBI网站工具ORF finder进行编码区的预测,确认获得了完整AiCL编码区序列。根据所测得的cDNA序列推导其氨基酸序列,利用Expasy网站(http://www.expasy.ch)提供的蛋白质组和序列分析工具ProtParam软件进行蛋白质基本物理化学参数分析,通过SWISS-MODEL对其进行结构预测,使用SMART软件预测功能域,利用SingalP程序(http://genome.cbs.dtu.dk/services/SignalP-2.0/)分析是否存在信号肽。将不同物种的组织蛋白酶通过ClustalX进行比对,进行相似性同源性分析(http://www.bioinformatics.org/sms/), 使用Mega3.1软件进行系统进化树的构建。

图1 AiCL基因编码区及其所推测的氨基酸序列。起始密码子ATG、终止密码子TGA以方框标注,N端信号肽和4个酶活性位点保守氨基酸以下划线标注,前体肽的切割位点以“|”表示Fig.1 Nucleotide sequence and the deduced amino acid sequence of AiCL.The translation initiation codon ATG and terminal codon TCA are boxed, and the signal peptide of N terminal and four conserved amino acid residues for enzyme activity are underlined, and the potential cleavage site is indicted by “|”

图 2 AiCL与其它物种的组织蛋白酶L多序列比对结果。一致性和相似度高的部分以阴影表示。物种来源及GenBank序列号见表1Fig.2 Alignment of AiCL with cathepsin L sequences from other species.The conserved parts are shadowed.The species and GenBank accession numbers are given in Tab.1
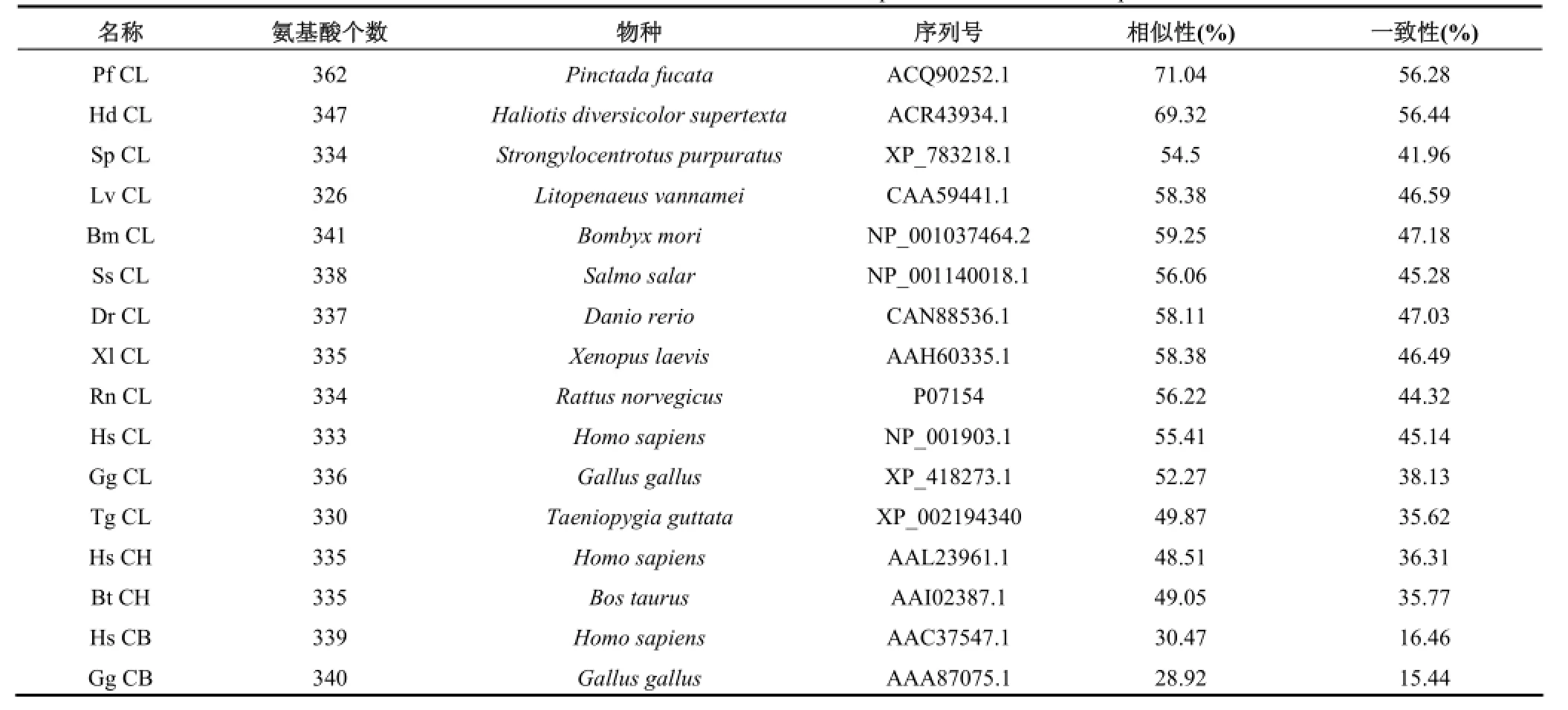
表 1 AiCL 与其它物种组织蛋白酶L的相似性和一致性Tab.1 Identities and similarities of AiCl compared with different cathepsins
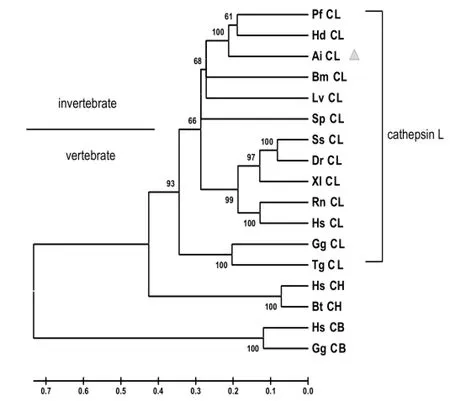
图 3 组织蛋白酶系统进化树。AiCL的位置以灰色三角表示Fig.3 Phylogenetic tree of cathepsins.AiCL is emphasized by a grey triangle
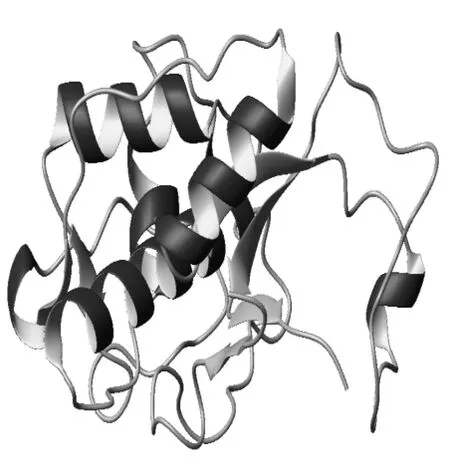
图 4 SWISS-MODEL预测的AiCL三维模型Fig.4 3D-model of AiCL predicted by SWISS-MODEL
2 结果与分析
所获得AiCL的cDNA序列长1 203 bp, 其中5’非编码区为76 bp,3’非编码区为32 bp。cDNA序列中编码区长1 095 bp, 编码364个氨基酸,通过ProtParam软件预测其蛋白分子量为41.2kD,等电点为4.98。通过NCBI的Conserved Domain Database比对,发现根据cDNA序列所翻译的蛋白序列中存在组织蛋白酶L活性位点的4个保守氨基酸:Q164, C170, H309, N329,它们对成熟蛋白空间结构和功能的稳定起着重要的作用[2]。通过SingalP预测在第17和18个氨基酸之间有一个信号肽的切割位点,而根据序列比对结果推测形成成熟蛋白时前体肽的切割位点在A145和M146之间(图1)。这与其它物种中成熟的有活性组织蛋白酶L的N端所具有高度保守的脯氨酸残基的特征相符,位于切割位点周围的氨基酸P147可以起到预防N端蛋白水解的作用[14]。AiCL的成熟蛋白分子由219个氨基酸构成,预测等电点较前体肽略有下降,为4.52。从序列比对结果中还发现6个极为保守的半胱氨酸残基,分别为:C167,C201,C210,C243,C302,C351,根据同源序列中相应位置的结构,它们能够形成3对二硫键:C167-C210,C201-C243,C302 -C351[15](图2)。在AiCL蛋白序列的C端存在ASYPTV,而在哺乳动物中其同源的ASYPLV被认为是一个细胞分泌的信号序列[16]。序列同源性分析中,AiCL蛋白序列与另外两种软体动物的组织蛋白酶L的同源性最高,序列一致性在50%以上;与脊椎动物的组织蛋白酶L序列一致性大多在40%至50%,而与鸟类的序列同源性略低;与其它两种组织蛋白酶H和B型的序列一致性最低,分别在35%左右和15%左右(详见表1)。此外,根据序列比对结果,通过Mega3.1软件利用邻接法(Neighbor-joining)构建了系统进化树(图3)。从图中可以看出,H型和B型组织蛋白酶都各自形成独立分枝,AiCL与其它物种的组织蛋白酶L聚合到一起,组成了一个分枝。在L型组织蛋白酶这一分支中,无脊椎动物和脊椎动物的组织蛋白酶L清晰的分为两个区域。通过SWISS-MODEL构建了AiCL成熟蛋白的三位立体结构(图4),虽然与其它组织蛋白酶L在序列上有所不同,但在空间结构却高度相似[17-20]。
综上所述,本研究通过RACE技术克隆得到了海湾扇贝组织蛋白酶L基因编码区全长,经序列比对与分析发现该基因的蛋白序列与许多物种同源序列十分相似,具有多个组织蛋白酶L的特征结构。这表明该基因在进化上比较保守。已有研究表明木瓜蛋白酶的半胱氨酸蛋白酶家族可能早在真核生物和原核生物分离之前就存在了,并且L、H、B型组织蛋白酶跨物种的高度保守性提示它们通过基因复制演变而来[21]。海湾扇贝组织蛋白酶L很可能在功能上与其同源蛋白相似,具有水解多种肌肉相关蛋白的能力。近年来越来越多的证据表明组织蛋白酶可能参与非caspase介导的细胞凋亡过程[22]。细胞凋亡又是免疫调节的中心环节,各种免疫细胞的代谢过程大多通过细胞凋亡实现[23]。而随着对无脊椎动物组织蛋白酶L日益增多的研究,人们发现这种蛋白水解酶可能在无脊椎动物的免疫反应中具有重要的作用[24]。在受鳗弧菌(Vibrio anguillarum)刺激后,中华绒鳌蟹(Eriocheir sinensis)组织蛋白酶L的表达量发生显著变化[25];相似的,溶藻弧菌(Vibrio alginolyticus)会引起合浦珠母贝(Pinctada fucata)组织蛋白酶L的急剧变化[15]。贝类组织蛋白酶L的多种功能有待我们进一步通过实验证实。
[1]Mellman I.Endocytosis and molecular sorting [J].Annual Review of Cell and Developmental Biology, 1996, 12: 575-625.
[2]Lecaille F, Kaleta J, Bromme D.Human and parasitic papain-like cysteine proteases: Their role in physiology and pathology and recent developments in inhibitor design [J].Chemical Reviews, 2002, 102(12): 4459-4488.
[3]Chauhan S S, Goldstein L J, Gottesman M M.Expression of cathepsin L in human tumors [J].Cancer Research, 1991, 51(5): 1478-1481.
[4]Kos J, Werle B, Lah T, et al.Cysteine proteinases and their inhibitors in extracellular fluids: markers for diagnosis and prognosis in cancer [J].The International Journal of Biol ogical Markers, 2000, 15(1): 84-89.
[5]Lindeman J H, Hanemaaijer R, Mulder A, et al.Cathepsin K is the principal protease in giant cell tumor of bone [J].The American Journal of Pathology, 2004, 165(2): 593-600.
[6]Sloane B F, Honn K V.Cysteine proteinases and metastasis [J].Cancer Metastasis Reviews, 1984, 3(3): 249-263.
[7]Sloane B F, Rozhin J, Hatfield J S, et al.Plasma membraneassociat--ed cysteine proteinases in human and animal tumors [J].Experimental Cell Biology, 1987, 55(4): 209-224.
[8]Hu K J, Leung P C.Shrimp cathepsin L encoded by an intronless gene has predominant expression in hepatopancreas, and occurs in the nucleus of oocyte [J].Comparative Biochemistry and Physiology.Part B, Biochemistry & Molecular Biology, 2004, 137(1): 21-33.
[9]Glenn K L, Grapes L, Suwanasopee T, et al.SNP analysis of AMY2 and CTSL genes in Litopenaeus vannamei and Penaeus monodon shrimp [J].Animal Genetics, 2005, 36(3): 235-236.
[10]Akpinar O, An H.Purification and determination of inhibitory activity of recombinant soyacystatin for surimi application [J].Molecular Nutrition & Food Research, 2005, 49(3): 247-255.
[11]Jiang S T, Lee B L, Tsao C Y, et al.Mackerel Cathepsins B and L Effects on Thermal Degradation of Surimi [J].Journal of Food Science, 1997, 62(2): 310-315.
[12]张福绥,何义朝,刘祥生, 等.海湾扇贝的引种、育苗与试养 [J].海洋与湖沼, 1986, 17(5): 367-374.
[13]张福绥,何义朝,杨红生.海湾扇贝引种工程及其综合效应 [J].中国工程科学, 2002, 2(2): 30-35.
[14]Wang B, Shi G P, Yao P M, et al.Human cathepsin F.Molecular cloning, functional expression, tissue localization, and enzymatic characterization [J].The Journal of Biological Chemistry, 1998, 273(48): 32000-32008.
[15]Ma J, Zhang D, Jiang J, et al.Molecular characterization and expression analysis of cathepsin L1 cysteine protease from pearl oyster Pinctada fucata [J].Fish & Shellfish Immunology, 2010, 29(3): 501-507.
[16]Chauhan S S, Ray D, Kane S E, et al.Involvement of carboxyterminal amino acids in secretion of human lysosomal protease cathepsin L [J].Biochemistry, 1998, 37(23): 8584-8594.
[17]Lewis S D, Johnson F A, Shafer J A.Effect of cysteine-25 on the ionization of histidine-159 in papain as determined by proton nuclear magnetic resonance spectroscopy.Evidence for a his-159-Cys-25 ion pair and its possible role in catalysis [J].Biochemistry, 1981, 20(1): 48-51.
[18]Rawlings N D, Barrett A J.Evolutionary families of peptidases [J].The Biochemical Journal, 1993, 290 (1): 205-218.
[19]Schröder E, Phillips C, Garman E, et al.X-ray crystallographic structure of a papain-leupeptin complex [J].FEBS Letters, 1993, 315(1): 38-42.
[20]Gillmor S A, Craik C S, Fletterick R J.Structural determinants of specificity in the cysteine protease cruzain [J].Protein Science : A Publication of the Protein Society, 1997, 6(8): 1603-1611.
[21]Berti P J, Storer A C.Alignment/phylogeny of the papain superfamily of cysteine proteases [J].Journal of Molecular Biology, 1995, 246(2): 273-283.
[22]Chwieralski C E, Welte T, Buhling F.Cathepsin-regulated apoptosis [J].Apoptosis, 2006, 11(2): 143-149.
[23]Krammer P H.CD95's deadly mission in the immune system [J].Nature, 2000, 407(6805): 789-795.
[24]Söderhäll K, Cerenius L.Role of the prophenoloxidase-activating system in invertebrate immunity [J].Current Opinion in Immunology, 1998, 10(1): 23-28.
[25]Li W W, Jin X K, He L, et al.Molecular cloning, characterization, expression and activity analysis of cathepsin L in Chinese mitten crab, Eriocheir sinensis [J].Fish & Shellfish Immunology, 2010, 29(6):1010-1018.
Cloning and sequence analysis of cathepsin L gene from bay scallopArgopecten irradians
LI Juan1,2, LI Li1, ZHANG Guo-fan1
(1.Institute of Oceanology, Chinese Academy of Sciences, Qingdao 266071, China; 2.Graduate University of Chinese Academy of Sciences, Beijing 100049, China)
The complete open reading frame (ORF) of cathepsin L was obtained from bay scallop (Argopecten irradians) by RACE technique.The ORF was of 1 095 bp, encoding 364 amino acids.Four conserved residues for enzyme activity (Q164, C170, H309, N329) and six preserved cysteines were found in the protein sequence.17 amino acids of N terminal were predicted to be the signal peptide and the sequence ASYPTV at C terminal was found to be a possible signal sequence.The mature protein of AICL contained 219 amino acids and the potential cleavage site was between A145 and M146.Analysis indicated AiCL shared the highest identities (>50%) with molluscan cathepsin Ls.It clustered with other invertebrate cathepsin Ls in the phylogenetic tree.The 3D-model predicted by SWISS-MODEL demonstrated that cathepsin L was conserved in tertiary structure.Collectively, data showed that AiCL was likely to catalyze the hydrolysis of multiple muscle related proteins.
Bay scallop (Argopecten irradians); Cathepsin L gene; cloning; sequence analysis
S917
A
1001-6932(2011)03-0338-06
2010-11-08;收修改稿日期:2011-01-10
国家高技术研究发展计划(863计划,2010AA10A401);国家自然基金(30800842);国家公益性行业(农业)科研专项(3-53)。
李娟(1981-),博士研究生,研究方向:海洋生物学。电子邮箱:lijuan@qdio.ac.cn。
张国范,研究员。电子邮箱:gfzhang@qdio.ac.cn。
