基于分类器串联融合的生物医学命名实体识别
2011-12-14马瑞民马民艳王浩畅
马瑞民,马民艳,王浩畅
(东北石油大学计算机与信息技术学院,黑龙江大庆 163318)
基于分类器串联融合的生物医学命名实体识别
马瑞民,马民艳,王浩畅
(东北石油大学计算机与信息技术学院,黑龙江大庆 163318)
鉴于生物医学命名实体识别的多数模型使用单机器学习算法时识别效果不好,提出一种基于条件随机域(CRFs)与最大熵(Maxent)分类器融合的方法,利用基分类器之间的相关性和互补性,结合有效的特征集合,进行再学习,得到融合模型.实验表明,该模型的识别性能与单一分类器和JNLPBA专题会议相关的系统比较,取得很好成绩,F测度达到70.7%,证明该融合方法有效.
条件随机域;最大熵;分类器融合;特征提取;生物医学命名实体识别
0 引言
生物医学的快速发展产生大量的生物医学数据,从生物医学文献中发掘出隐含的生物医学知识是生物医学信息抽取的重要意义所在.生物医学命名实体识别是生物医学信息抽取的一项重要任务,它主要是从医学文献中发现基因、蛋白质、DNA、RNA等生物医学命名实体.目前,生物医学命名实体识别的方法主要有基于字典、基于规则和基于机器学习3种方法[1].
在生物医学命名实体识别的研究初期,常使用基于字典的方法,如 Krauthammer M等[2]利用DNA和蛋白质序列比较工具BLAST识别生物医学命名实体.此方法简单实用,对于字典中存在的生物医学命名实体有极高的识别准确率;但由于新的生物医学命名实体不断出现,所以基于字典的方法对于自由文本的生物医学命名实体识别效果不佳.
Olsson F等[3]利用基于规则的方法识别生物医学命名实体,F达到67%.与基于词典的方法比较,基于规则方法的识别性能有所增强,但它具有花费大量人工劳动、可移植性差等缺点.
近年来,基于机器学习的方法成为生物医学命名实体识别的重要方法.文献[4-7]分别提出基于隐马尔可夫模型、感知器、支持向量机、最大熵的方法,这些方法把词性、词形等特征融入到机器学习模型中,利用训练得到的学习模型从生物医学文本集合中识别指定类型的名称.Zhou Guodong等[4]的研究 F达到74%.
由于基于机器学习的方法能够判别生物医学命名实体数据库中未包含的实体,方法灵活.该方法已成为生物医学命名实体识别研究的主流方法.目前,生物医学命名实体识别的多数识别模型是使用单机器学习算法,单机器学习算法不能够取得非常好的识别效果.这说明单一算法结合丰富的特征并不能完全表达生物医学命名实体的特点.通过对各种统计学习方法的分析可以发现,不同的学习模型之间存在着互补性和相关性,所以分类器融合是一种改进的新思路.
为此,笔者采用条件随机域算法和最大熵算法二者融合的方法,结合丰富的特征集合,包括局部特征、全文特征和外部资源特征,以提高生物医学命名实体识别的识别性能.
1 算法
1.1 条件随机域算法
条件随机域(Conditional Random Fields,CRFs)在自然语言处理领域当中适合解决标注序列化数据任务,模型的特性表明它非常适用于生物医学领域的命名实体识别研究,该算法详见文献[8].
1.2 最大熵算法
最大熵(Maximum Entropy,Maxent)算法在自然语言处理方面也是一种主要的处理方法.该方法最大的优点是,实验者只需要考虑提取特征,而不用考虑如何使用这些特征,每个特征的贡献由相应的权值来决定,而这些权值可由 GIS学习算法自动得到,该算法详见文献[9].

图1 分类器融合图示
1.3 融合方法
如果把单个分类器比作一个决策者,分类器融合的方法就相当于多个决策者共同进行一项决策.
分类器融合的基本思想见图1.选用丰富的特征集合,首先利用条件随机域算法训练得到一个基本分类器M1,输出结果为C1,然后把 C1添加到特征集合中,再利用最大熵算法进行训练和测试,即将这2种基本分类器进行融合,得到分类器M2,最终的测试训练结果为C2.
2 特征选取
生物医学命名实体识别中常用的特征主要有:局部特征,包括文本符号本身的特征和文本符号局部的上下文特征及其周围的词或符号的特征;全文特征,即文本符号在整个篇章中的上下文特征;外部资源特征,如使用一些外部资源词典等.
使用7种特征[10]:
(1)单词本身.把单词本身作为一个识别特征.
(2)词形特征.由于生物医学命名实体一般含有数字、大写字母和特殊符号等,将这些简单的表面特征定义为词形特征.本实验将大写字母用‘A’替换,数字用‘0’替换,非英语字符用‘-’替换,小写字母用‘a’替换.
(3)标准化拼写特征.某些同类的生物医学命名实体拼写方式很类似,如NP-1and NP-5.对这些类似词采取方法处理,如Jcllc-B,将它规范化为‘Aaaaa—A’,将连续相同的字符再缩短,即‘Aa—A’.此方法能够将拼写相似的命名实体提取的特征保持一致.
(4)词性特征.生物医学命名实体的大写字母特征对其识别性能贡献不大,并且生物医学命名实体多是描述性的名称而且名称很长,所以,词性特征对识别生物医学命名实体边界很有帮助.本系统使用 GEN IA tagger2.0.2[11]词性标注器,该词性标注器是生物医学领域文本专用的词性标注器.
(5)关键词特征.利用统计方法在训练集中统计出高频的生物医学命名实体关键词,将这些词是否出现作为特征.
(6)别名特征.把已经识别出来的生物医学命名实体保存在一个表中,当系统开始识别某个词时,生物医学命名实体识别算法会对该词是否是表中词的别名做出决定.
(7)字典特征.使用一些字典资源作为特征加入特征向量空间,有Common Word词典、Species词典、Tissue词典和 Endings of Chem icals词典[12]等.
如句子:Number of GLucocorticoid(p rotein)recep to rs in lymphocytes(cell-type)and their sensitivity to hormone action特征提取见表1.

表1 特征提取实例
3 实验与结果分析
实验采用的语料是JNLPBA 2004,用它进行训练和测试.JNLPBA的训练语料由 GEN IA 3.02语料中的2 000篇摘要组成,测试语料由当时未出版的404篇M EDL INE摘要组成.
实验使用2 000篇训练语料,语料中的命名实体分为5类:DNA、RNA、Protein、Cell—line和 Cell—type.利用.net平台、采用c#编程语言完成文中实验系统,实验结果由精确率(P)、召回率(R)和 F测度(F)评价,且使用全部匹配模式.
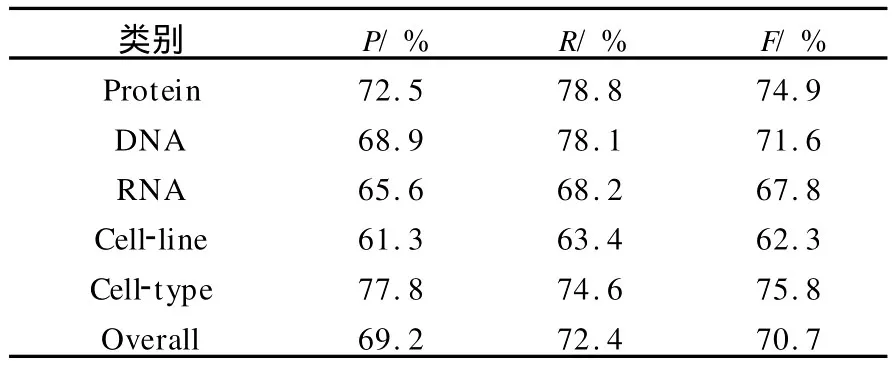
表2 CRFs与Maxent融合模型各类别实体实验结果
利用CRFs算法与Maxent算法进行融合,融合模型对各类别实体实验结果见表2,其中O-verall为5类生物医学命名实体的平均值.
为验证融合方法的有效性,本实验测试CRFs和Maxent单一分类器的识别性能,F测度分别为69.2%和67.5%,采用 CRFs与 M axent融合方法后,F测度达到70.7%,结果见表3.
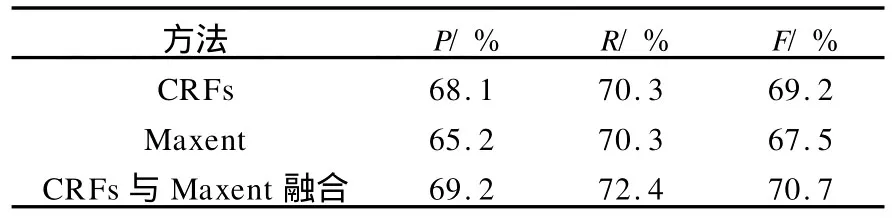
表3 CRFs与Maxent单一分类器与融合后结果
由表3可以看出,CRFs的性能要高于M axent,而融合后的分类器比单一分类器的识别性能提高1.5%左右,证明该融合方法有效.
基于CRFs与Maxent融合的系统和JNLPBA专题会议相关系统比较的结果见表4.由表4可以看出,该方法取得较好的效果.
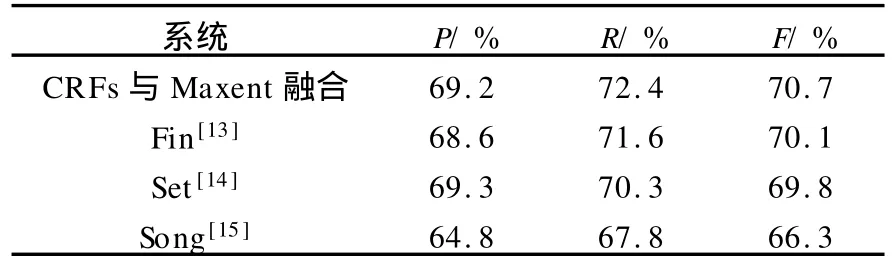
表4 CRFs与Maxent融合与JNLPBA相关系统结果
通过分析实验数据,可以得到:
(1)最大熵模型获得的是所有满足约束条件的模型中信息熵极大的模型.此模型中二值化特征只是记录特征的出现与否,而文本分类需要知道特征的强度,因此它在分类方法中不是最优的;但是它能解决统计模型中参数平滑的问题.
(2)CRFs模型是在M axent模型的基础上改进的,它能更好地利用待识别文本中所提供的上下文信息,并且避免严格的独立性假设和数据归纳偏置问题.
(3)CRFs模型和最大熵模型融合后,系统性能明显提高,F测度达到70.7%.这是因为CRFs模型能够赋予M axent模型适当的权重,有效利用基分类器结果之间的互补性和相关性,从而提高识别性能.
4 结束语
提出了基于CRFs分类器和Maxent分类器融合的方法.该方法利用2种分类器之间的互补性和相关性,有效地弥补单一分类器的不足.实验表明,结合有效特征集合,将CRFs分类器和M axent分类器融合是一种非常有效的融合方法,融合后识别性能明显优于基于单分类器的识别系统,F测度达到70.7%,对于一定领域内的生物医学命名实体识别任务有很好的效果.
[1]王浩畅,赵铁军.生物医学文本挖掘技术的研究与进展[J].中文信息学报,2008,22(3):89-98.
[2]Krauthammer M,Rzhetsky A,Morozov P,et al.Using BLAST for identifying gene and p rotein names in journal articles[J].GENE,2000,259(1):245-252.
[3]Olsson F,Er iksson G,Franzen K,et al.Notions of co rrectness w hen evaluating p rotein name taggers[C/OL]//Proceedings of the 19 th international conference on computational linguistics.2002:765-771[2007-05-10].http://www.sics.se/~fredriko/papers/coling02.pdf.
[4]Zhou Guodong,Zhang Jie,Su Jian,et al.Recognizing names in biomedical texts:a machine learning app roach[J].Bioinformatics,2004,20(7):1178-1190.
[5]胡俊锋,陈浩,陈蓉,等.基于感知器的生物医学命名实体边界识别算法[J].计算机应用;2007,27(12):3026-3031.
[6]王浩畅,赵铁军.基于SVM的生物医学命名实体识别[J].哈尔滨工程大学学报,2006,27(增):570-574.
[7]L N Y F,TSA IT H,Chou W C,et al.A maximum entropy app roach to biomedical named entity recognition[C/OL]//4th workshop on datamining in bioinfo rmatics.2004:56-61[2007-05-01].http://iasl.iis.sinica.edu.tw/w ebpdf/paper-2004-A—Maximum—Entropy—App roach—to—Biomedical—Named_Entity—Recognition.pdf.
[8]Lafferty J,M ccallum A,Pereira F.Conditional random fields:p robabilistic models for segmenting and labeling sequence data.p roc.of the 18th international conference on machine learning[C].San Francisco:2001:282-289.
[9]Tom M.机器学习[M].北京:机械工业出版社,2000:166-170.
[10]马瑞民,马民艳.基于CRFs的多策略生物医学命名实体识别[J].齐齐哈尔大学学报,2011,27(1):39-42.
[11]Yoshimasa T,Yuka T,Kim Jin-Dong,et al.Developing a robust part-of-speech tagger fo r biomedical text[A].Advances in Info rmatics-10th panhellenic conference on info rmatics[C].Japen,[s.l.]2005.
[12]M ika S R.Protein names peeled p recisely off free text[J].Bioinfo rmatics,2004,20:241-247.
[13]Finkel J,Dingare S,Nguyen H,et al.Exp loiting context fo r biomedical entity recognition:from syntax to the web[A].Proceedings of the joint wo rkshop on natural language p rocessing in biomedicine and its app lications(JNLPBA-2004)[C].Geneva:Sw itzerland,2004.
[14]Settles B.Biomedical named entity recognition using conditional random fields and novel feature sets[A].Proceedings of the joint wo rkshop on natural language p rocessing in biomedicine and its app lications(JNLPBA-2004)[C].Geneva,Sw itzerland,2004.
[15]Song Y,Km E,Lee G G,et al.POSB DTM-NER in the shared task of BioNLP/NLPBA 2004[C]//Proceedings of the joint wo rkshop on natural language p rocessing in biomedicine and its app lications,2004:100-103[2007-05-01].http://isoft.postech.ac.kr/publication/iconf/bionlp04—song.pdf.
Bio-entity recogn ition based on cascade generalization/2011,35(2):91-94
M A Rui-m in,M A M in-yan,WANG Hao-chang
(College of Com puter and Inform ation Technology,N ortheast Petroleum University,Daqing,Heilongjiang 163318,China)
Currently,most of methods for bio-entity recognition are based on a single machine learning algo rithm and it can not achieve better perfo rmance.Therefo re,in this paper,w e p ropose a cascade generalization method based on the CRFs and Maxentw hich makes use of the compensation and relativity among different classifiers.Experimental results show that the cascade generalization method isobviously superior to the individual classifier based method and the most state of the art system s in JNLPBA conferences.F value reached 70.7%,show ing that the fusion method is effective.
conditional random fields;maximum entropy;cascade generalization;feature extraction;bio-entity recognition
TP311.135
A
1000-1891(2011)02-0091-04
2010-11-03;审稿人:刘贤梅;编辑:陆雅玲
黑龙江省自然科学基金项目(F200603)
马瑞民(1958-),男,教授,主要从事数据库及相关技术方面的研究.
