兔防御素基因在毕赤酵母中的串连表达及抑菌机理研究
2011-11-10刘霭珊徐来祥赵亚华
李 霞,刘霭珊,徐来祥,赵亚华
(1 华南农业大学生命科学学院,广东广州 510642;2 曲阜师范大学生命科学学院,山东曲阜 273165)
哺乳动物防御素具有广泛的抗性谱,可以抗细菌、真菌、被膜病毒,甚至对支原体、衣原体、螺旋体及肿瘤细胞和爱滋病病毒也有杀伤作用[1-2].此外,防御素还可启动获得性免疫系统,因此越来越受到人们的关注.哺乳动物的免疫系统包括特异性和非特异性免疫.非特异性免疫对各种入侵机体内的微生物在最初反应中起关键作用,哺乳动物防御素(Defensins)是其非特异性免疫的主要成分,存在于多形核粒细胞和上皮组织内,如人、大鼠、豚鼠、兔的嗜中性粒细胞及人和小鼠的小肠潘氏细胞内[3].防御素富含Cys,在分子内形成二硫键,根据其分子内二硫键的连接位置不同可分为α-防御素和β-防御素[4].α-防御素由 29 ~36 个氨基酸(aa)组成,分子内含6个保守半胱氨酸形成的3对二硫键,连接位点分别为 Cys1-Cys6、Cys2-Cys4 和 Cys3-Cys5[5].二硫键可使小分子防御素分子致密以防蛋白酶水解,致使防御素在吞噬溶酶体环境中能保持活性.在兔中性粒细胞(Rabbit neutrophil peptide,NP)中分离到6种 α-防御素的阳离子抗菌肽:NP-1、NP-2、NP-3a、NP-3b、NP-4、NP-5,长度均约 32-34 个 aa,富含 Cys和Ary,每种防御素都含3个分子内二硫键.比对它们的aa组成,其序列高度保守,对其生物学活性及结构有重要的作用.2003年徐文生等[6]对NP-1进行过原核表达,表达量占细菌总蛋白的34.7%.本研究采用毕赤酵母Pichia pastoris表达系统对pNP-1基因及其串联基因进行表达,表达出的多价串联体的重组蛋白经溴化氰切割,将得到的产物进行抑菌试验,初步研究兔防御素的抑菌机理.
1 材料与方法
1.1 菌株与质粒
Pichia pastoris GS115菌株、pPICZα-A质粒购于Invitrogen公司;常规克隆宿主菌大肠埃希菌Escherichia coli DH5α,抗菌活性检测菌株大肠埃希菌K12D31、白色念珠菌Candida albicans、灵菌Serratia macrescens、枯草杆菌Bacillus subtilis、金黄葡萄球菌Staphylococcus aureus及苏云金杆菌Bacillus thuringiensis均由华南农业大学生命科学学院生化实验室保存.
1.2 酶与试剂
Taq DNA聚合酶,CIAP碱性磷酸酶,T4DNA连接酶,限制性内切酶 EcoRⅠ、SalⅠ、XhoⅠ、SacⅠ 购自大连宝生物工程公司;肠激酶购自NOVAGEN公司;质粒小提试剂盒、DNA凝胶回收试剂盒购自天根生化科技有限公司;Zeocin、PVDF超滤膜和ProBond蛋白纯化试剂盒购自Invitrogen公司;引物合成及测序由上海英骏生物公司完成;其余试剂均为进口或国产分析纯.
1.3 pNP-1基因及其串联基因的设计
根据毕赤酵母密码子偏爱性重编兔防御素基因后的pNP-1基因序列:5'-CGCGAATTCGTCGTCTGTGCTTGTAGAAGAGCTTTGTGTTTGCCAAGAGAAAGAAGAGCTGGTTTCTGTAGAATCAGAGGTAGAATCCACCCATTGTGCTGCAGAAGAGTCGACCGC-3'(下划线为EcoRⅠ和SalⅠ酶切位点).根据已调整的pNP-1序列,使用 Primer Priemir 5.0设计4条搭桥引物pNP-1(1)、pNP-1(2)、pNP-1(3)和 pNP-1(4),用于合成pNP-1序列.
利用PCR的方法在pNP-1基因5'端加上XhoⅠ酶切位点及蛋氨酸密码子ATG,在3'端加上SalⅠ酶切位点,合成pNP-1的串联基因[pNP-1(2×)]和[pNP-1(3×)].引物序列见表1.

表1 引物序列Tab.1 List of primers
1.4 pNP-1基因与其串联基因的合成与扩增
1.4.1 pNP-1基因的合成及重组质粒 pPICZaA-pNP-1的构建 4条搭桥引物作为模板进行PCR,PCR循环参数:94℃ 2 min,94℃ 30 s、52℃ 30 s、72℃ 40进行25个循环,72℃ 2 min.取上步产物作为模板,用pNP-1(1)和pNP-1(4)为引物进行第2次PCR扩增,程序为:94℃ 2 min,94℃ 30 s、52℃30 s、72℃ 40 s进行25个循环,72℃ 10 min.琼脂糖凝胶回收试剂盒纯化后得到pNP-1基因.
将pNP-1基因与载体pPICZα-A均经EcoRⅠ和SalⅠ酶切后回收,连接,转化E.coli DH5α.将此克隆的重组质粒命名为 pPICZaA-pNP-1.用 AOXⅠ和pNP-1(1)(表1)作为引物进行菌落PCR检测.
1.4.2 pNP-1串联基因的合成 以pPICZα-A-pNP-1重组质粒为模板,将pNP-1基因串联的正向和反向引物(表1)进行PCR反应合成串联基因pNP-1(2×),以此类推得到pNP-1(3×).PCR反应程序:94℃ 5 min,94 ℃ 30 s、52 ℃ 30 s、72 ℃ 20 s进行25 个循环,72℃ 10 min.琼脂糖凝胶回收试剂盒纯化后得到pNP-1(2×)和pNP-1(3×).
1.4.3 pNP-1多价串联重组质粒的构建 pPICZaA-pNP-1重组质粒经SacⅠ单酶切消化后CIAP去磷酸化;pNP-1串联基因经XhoⅠ和SalⅠ双酶切,连接转化E.coli DH5α.将此双拷贝重组质粒命名为pPICZaA-pNP-1(2×).同样在 pPICZaA-pNP-1(2×)基础上构建pNP-1三拷贝基因重组质粒pPICZaA-pNP-1(3×).
1.5 多倍体酵母重组质粒的构建
3种重组质粒分别经SacⅠ酶切线性化.电转毕赤酵母 GS115 感受态细胞(2.0 kV,25 μF ,200 Ω).30℃培养72 h.重组质粒使用AOXⅠ和pNP-1(4)通过菌落PCR鉴定.PCR反应程序:94℃ 10 min,(94℃ 30 s,50℃ 30 s,72 ℃ 50 s)30个循环,72 ℃10 min.产物进行10 g·L-1琼脂糖凝胶电泳观察鉴定.
1.6 重组酵母诱导表达后重组蛋白检测及纯化
分别选取生长最旺盛的3种重组酵母菌进行甲醇诱导表达.挑取单菌落接种于BMGY培养基中,诱导培养72 h后停止,离心收集上清,使用三氯乙酸(TCA)沉淀法初步浓缩上清,Tricine-SDS-PAGE检测.大量表达重组蛋白使用硫酸铵沉淀法沉淀,取沉淀溶解.将溶解后的样品使用Sephadex-G25凝胶过滤脱盐.对于多倍体重组蛋白而言,上述操作后还需进行镍柱纯化.纯化样品经Tricine-SDS-PAGE凝胶电泳检测.
1.7 单体与多倍体裂解后的蛋白抑菌效价测定
将1.6得到的多倍体重组蛋白使用CNBr进行特异水解,冷冻干燥备用抑菌试验.
1.7.1 琼脂扩散法和稀释法测定抑菌效价 取纯化后的单体蛋白和多倍体裂解后样品,加ddH2O溶解备用.制备指示菌(金黄葡萄球菌,大肠埃希菌K12D31,枯草杆菌,苏云金杆菌、白色念珠菌、灵菌)菌悬液.配制LB及PDA半固体培养基平板.分别取菌悬液200 μL涂布于相应的半固体平板上.待平板完全吸收菌液后,取灭菌的滤纸片放置在平板上,分别浸润5、10、20和40 μL的样品,待样品溶液被平板完全吸收后,放置于37℃培养12 h.观察抑菌情况.
取琼脂糖扩散法中菌悬液,10倍倍比稀释109倍,取5、10、15、20 和25 μL 涂布固体培养基,记录各板生长的菌落数,计算出菌液的每毫升单位菌落总数(CFU/mL).此外,将菌悬液稀释至105CFU/mL,分别取 50 μL 于 96 孔板,加入样品液 0、2、4、6、8 和10 μL,再加入无菌水补至10 μL总体积,37℃培养2 h全部取出涂布半固体平板,37℃过夜.计算平板上生长的菌落,算出细菌存活率.
1.7.2 多种抗生素对pNP-1抗金黄葡萄球菌抑菌效价的影响 取氨苄青霉素、链霉素、氯霉素、卡那霉素、利福平、新生霉素和四环素,用上述各抗生素的5倍最低抑菌浓度[6],对处于对数生长期(107CFU/mL)的金黄葡萄球菌处理1 h,离心沉淀菌体,用PBS洗净,并用LB培养基稀释菌浓度至105CFU/mL,分别取50 μL 稀释后菌液于96 孔板,加入在菌悬液0、2、4、6、8和10 μL,补水至10 μL总体积,37 ℃培养2 h后测定细菌存活率.以未用抗生素处理的菌体作对照.
2 结果与分析
2.1 pNP-1单基因的合成
pNP-1单基因的PCR扩增,电泳如图1所示,第1、2、3泳道均有一条约100 bp的电泳谱带,与预期的pNP-1基因大小117 bp相符.
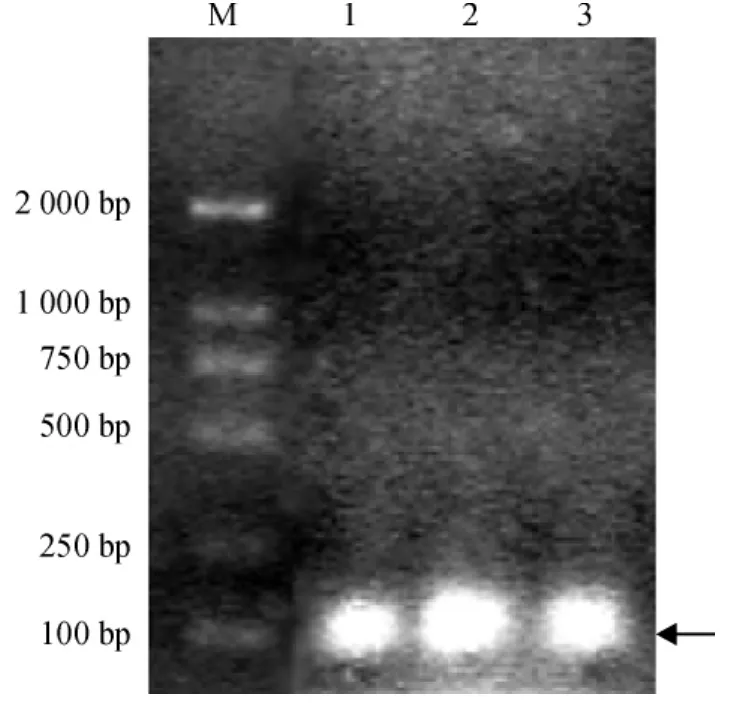
图1 pNP-1基因的PCR合成Fig.1 PCR amplification of pNP-1 gene
2.2 3种重组质粒的PCR鉴定
3种重组质粒转化DH5a的单菌落进行菌落PCR,电泳结果见图 2.泳道 1、2、3 分别与 pNP-1、pNP-1(2×)和pNP-1(3×)的预期大小456、556和656 bp相符,说明重组质粒中存在有不同拷贝数的目的片段.
2.3 pNP-1及其重组子重组酵母的菌落PCR鉴定
3个基因转化GS115后长出的酵母单菌落PCR后电泳结果如图3.3个基因重组酵母与其重组DH5a的菌落PCR结果基本一致,说明重组质粒已经成功整合进入酵母基因组中.
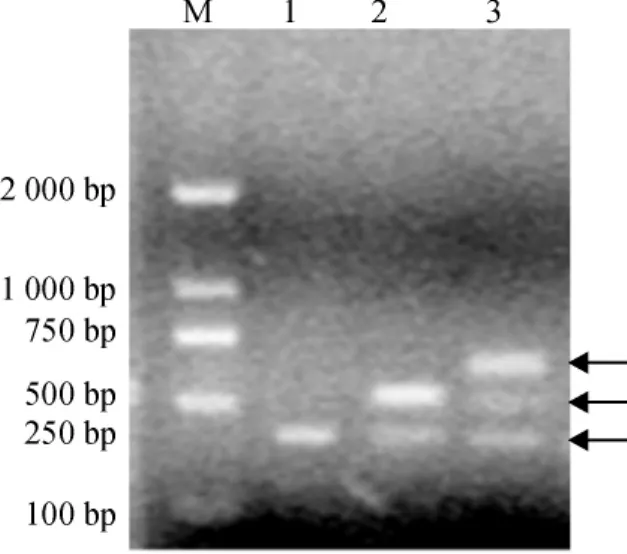
图2 pNP-1及其多串联体重组子转化大肠埃希菌后的菌落PCR鉴定Fig.2 Identification of pNP-1,pNP-1(2 × )and pNP-1(3 × )recombination plasmids by PCR in Escherichia coli
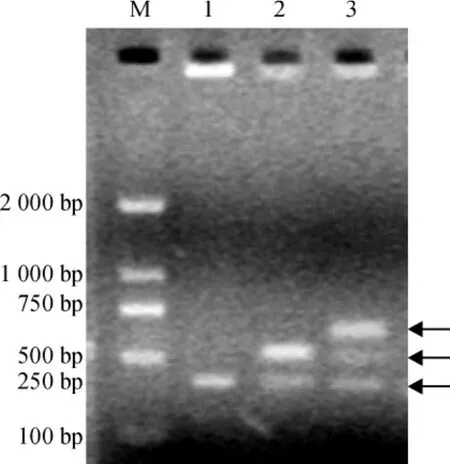
图3 pNP-1及其多串联体重组子转化GS115后的菌落PCR鉴定Fig.3 Identification of pNP-1,pNP-1(2×)and pNP-1(3×)by colony PCR in GS115
2.4 pNP-1重组酵母的甲醇诱导表达
对诱导表达的重组蛋白进行Tricine-SDS-PAGE,结果见图4,pNP-1、pNP-1(2×)和 pNP-1(3×)重组蛋白相对分子质量(Mr)约与Marker上5000、9000和13000相对应,与其重组蛋白的理论Mr5490、9380、13270大小基本相符.
用 Bandscan 5.0软件计算 pNP-1、pNP-1(2×)和pNP-1(3×)重组蛋白的表达量分别为21.6%、30.7%和41.8%.表达上清液经考马斯亮蓝G250法测定总蛋白质量浓度分别为15.7、20.8和31.2 mg/L,总体积为100mL.由此得知重组蛋白在发酵液中的质量浓度分别为3.39、6.39和13.05 μg/mL.
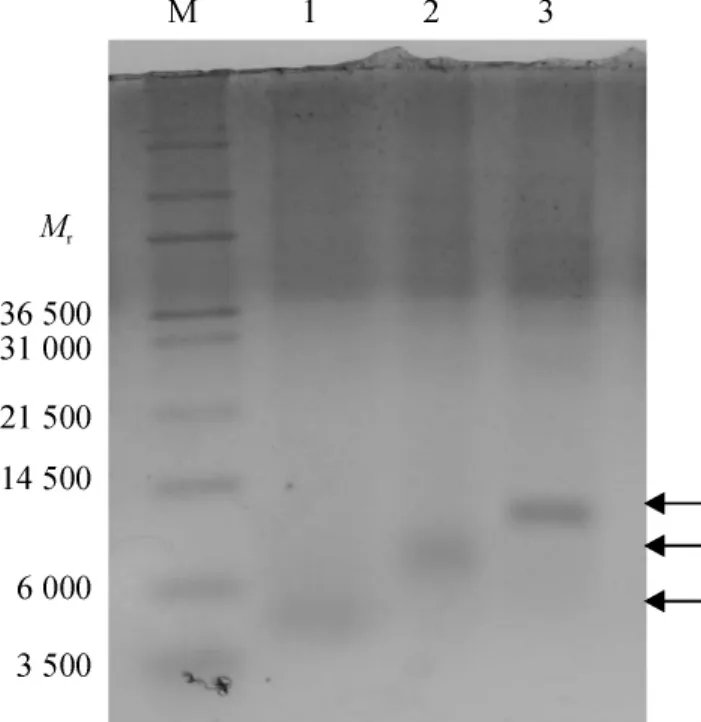
图4 重组酵母的甲醇诱导表达Fig.4 Induced expression of pNP-1,pNP-1(2 × )and pNP-1(3×)by methanol in recombinant GS115
2.5 pNP-1重组蛋白的浓缩与纯化
2.5.1 硫酸铵沉淀浓缩重组蛋白 硫酸铵沉淀浓缩后的 pNP-1、pNP-1(2×)和 pNP-1(3×)进行Tricine-SDS-PAGE后结果如图5所示.重组蛋白条带单一,与Marker上Mr约5000、9000和13000的条带相对应,与其重组蛋白的理论值相符.
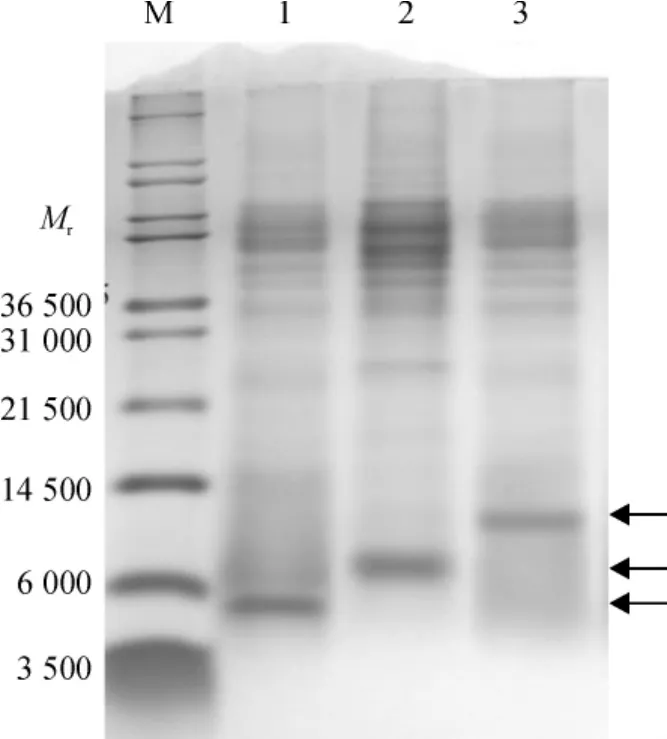
图5 硫酸铵沉淀浓缩后的重组蛋白电泳图Fig.5 Tricine-SDS-PAGE of recombined protein concentrated by ammonium sulfate
2.5.2 pNP-1多倍体重组蛋白的Ni柱纯化 pNP-1(2×)和pNP-1(3×)重组蛋白经Ni柱纯化后电泳结果如图6所示,纯化后的pNP-1(2×),pNP-1(3×)重组蛋白基本只剩下Mr为9000和13000的组分,与预期大小相符.
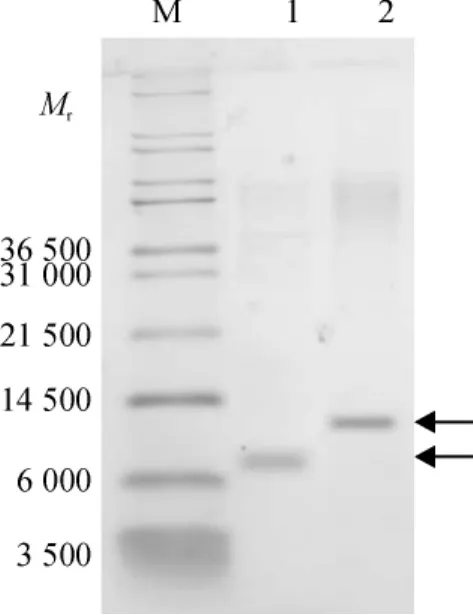
图6 纯化的pNP-1(2×)和pNP-1(3×)重组蛋白电泳图Fig.6 Tricine-SDS-PAGE of purified recombinant pNP-1(2 × )and pNP-1(3×)
2.5.3 多倍体重组蛋白的CNBr特异水解 经CNBr水解后冻干的pNP-1(2×)和pNP-1(3×)重组蛋白溶于ddH2O,电泳结果如图7所示.由图7可见,出现了Mr约为3800和5000的2条带.其中Mr约为5000的带较浅,为带有表达载体上1600 bp的pNP-1重组蛋白,Mr约为3800的带为CNBr水解下来的pNP-1蛋白.

图7 水解[pNP-1(2×)]、[pNP-1(3×)]重组蛋白纯化电泳图Fig.7 Tricine-SDS-PAGE of hydrolyzation of recombined pNP-1(2×)and pNP-1(3×)by cyanogen bromide
2.6 重组pNP-1的抑菌试验结果
琼脂糖扩散法抑菌试验结果显示,pNP-1对苏云金杆菌、金黄葡萄球菌、枯草杆菌等G+和大肠埃希菌K12D31等G-细菌均有明显的抑菌作用,而对白色念珠菌没有抑菌作用,对灵菌有轻微的抑菌作用.取琼脂糖扩散法试验中的孢子悬液10倍倍比稀释后得到的菌液做指示菌存活率试验,得到的pNP-1质量浓度与指示菌存活率关系如图8所示,该图中显示的结果与琼脂糖扩散法抑菌试验结果相符合.
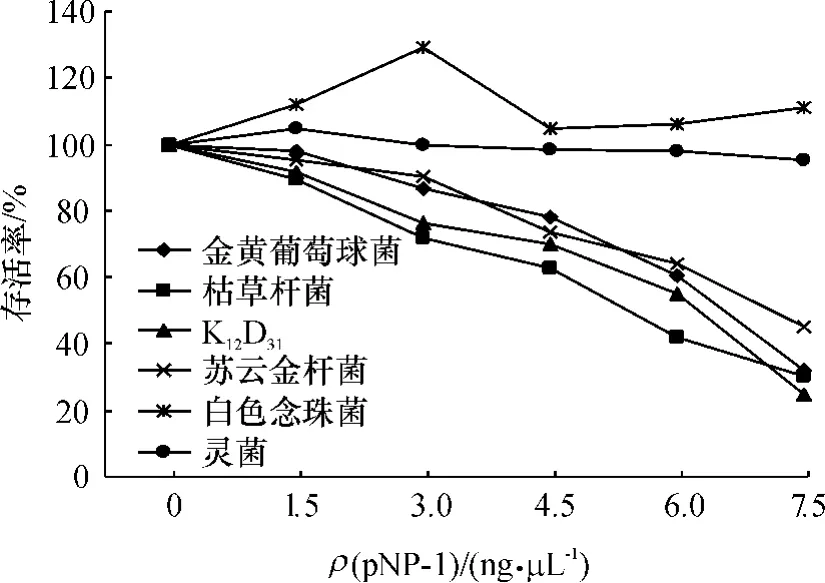
图8 pNP-1质量浓度与指示菌存活率的关系Fig.8 The relationship between pNP-1 mass concentration and survival rate of indicator bacteria
经过抑菌数据的线性回归分析,pNP-1对各菌的最小抑菌质量浓度分别为金黄葡萄球菌8.59 μg/mL,枯草杆菌 8.13 μg/mL,K12D318.46 μg/mL,苏云金杆菌 10.63 μg/mL.
2.7 不同抗生素处理对重组pNP-1抑菌活性的影响
将经氨苄青霉素、氯霉素、链霉素、卡那霉素、新生霉素、四环素和利福平处理过的金黄葡萄球菌与不同浓度的pNP-1作用,得到7种抗生素对pNP-1抑菌活性影响的数据,如表2所示.细胞壁合成抑制剂氨苄青霉素处理后再培养2 h的金黄葡萄球菌的存活率比对照组下降了48.7%,添加了重组pNP-1之后,存活率比对照组平均下降了45.9%,说明pNP-1与氨苄青霉素都对金黄葡萄球菌有抑制作用,两者之间的抑菌表现出同促效应.蛋白质合成抑制剂卡那霉素、氯霉素、链霉素和四环素处理2 h后的金黄葡萄球菌的存活率分别下降了59%、13%、28%和40.8%,再加入pNP-1处理后存活率均没有出现显著的下降,说明多种蛋白质合成抑制剂与pNP-1在抑菌方面没有明显的关系.RNA合成抑制剂利福平处理组的数据显示,重组pNP-1对利福平产生同促效应.DNA合成抑制剂新生霉素处理组,经过不同浓度的重组pNP-1处理后,细菌存活率最大下降值为6.5%,与对照组相比其下降幅度较小.2种核酸合成抑制剂对重组pNP-1表现不同的效应.
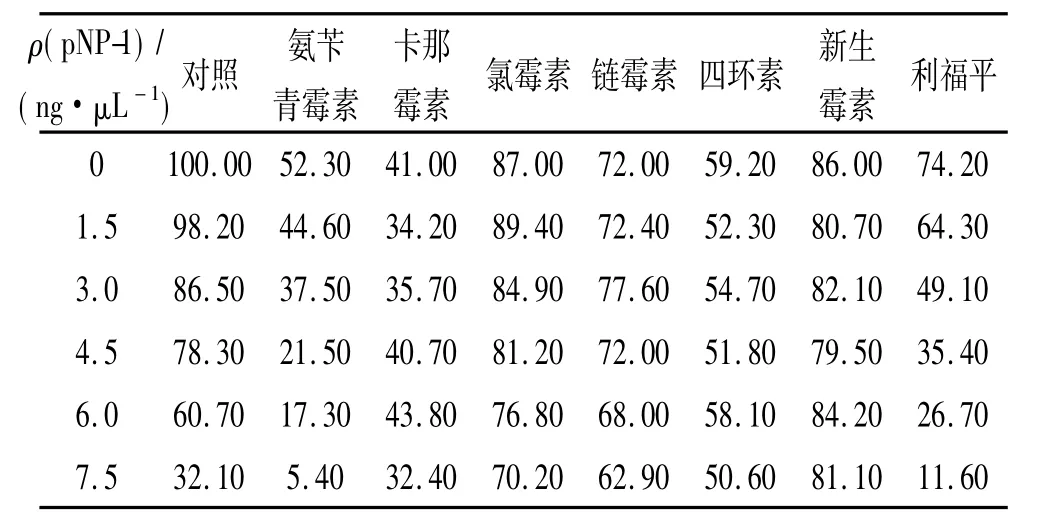
表2 不同抗生素处理过的金黄葡萄球菌在不同质量浓度重组pNP-1蛋白下的存活率Tab.2 Survival rates of different antibiotic-treated Staphylococcus aureus under different mass concentrations of recombinant pNP-1 protein %
3 讨论
围绕兔防御素基因的表达,本研究实施了一系列有利于该基因表达的策略.目的基因中含过多的稀有密码子被认为是低表达水平和产生不完全产物的一个原因[7-8].尽管少量稀有密码子的出现通常不会给目的蛋白的合成造成太大影响,但如果一个基因中含有成串或多个稀有密码子,外源蛋白的表达水平将非常低.当在氨基端附近出现多个稀有密码子时,情况更为严重[9].根据毕赤酵母表达偏爱性优化最适合表达的pNP-1基因,设计引物人工搭桥合成,使用同尾酶构建多串联体在毕赤酵母中表达,明显提高了重组蛋白的表达量.1996年Lee等[10]为扩大NP-1基因的表达量,尝试使用E.coli表达各种的融合串联多肽,这一方法使多肽的表达有显著提高,但表达量的增加并未与串联数的增长成正相关,特别是当串联数较大时,串联体的表达量反而降低.
限于试验条件,本研究对于pNP-1在毕赤酵母系统中的表达的所有数据都由摇瓶发酵获得,而培养温度、通气量、培养基酸碱度、培养密度等培养条件对表达量均有重要的影响,这些因素只有在发酵罐发酵的条件下才能得到严格控制,因此pNP-1基因的毕赤酵母发酵表达的过程仍有待进一步的优化.
很多研究都表明其作用位点不局限于只对细菌的膜结构,而更多是参与抑制细菌中核酸和蛋白合成的过程[11-14].同时,也有研究[15-16]表明,防御素对细菌细胞膜的作用只是其整个抑菌过程的第1步,通过在细菌膜上形成穿孔或增加膜的通透性使其更容易进入细胞内部,并与细胞内的其他作用位点相互作用而进一步抑制细菌的生长.不同的抗生素有其特异的作用位点,对于防御素的作用机制,这也在本试验中有所体现,但其具体作用机制还有待进一步研究.
[1]韩玉萍,翟朝阳.抗菌肽的抗肿瘤作用[J].生命的化学,2006,26(2):12-16.
[2]朱元军.防御素抗HIV作用[J].国际病毒学杂志,2006,13(1):12-20.
[3]王伯瑶,吴琦.内源性抗生素肽—天然免疫的介质[J].生命科学,1999,11(64):41-52,
[4]GANZ T.Defensins:Antimicrobial peptides of vertebrates[J].C R Biol,2004,327(6):539-549,
[5]ENNO K,KNUT A,AXEL S.Synthesis and structure-activity relationship of β-defensins,multi-functional peptides of the immune system[J].J Pept Sci,2006,12(4):243-257.
[6]徐文生,许杨.兔防御素NP1在大肠杆菌中的表达[J].南昌大学学报:理科版,2003,27(3):295-298.
[7]SORENSON M A,KURLAND C G,PEDERSON S.Codon usage determines translation rate E.coli[J].Mol Biol,1989,207(2):365-377.
[8]ZHANG Chun-ting,ZHANG Ren.Analysis of distribution of bases in the coding sequences by a diagrammatic technique[J].Nucleic Acids Res,1991,19(22):6313-6317.
[9]CHEN Gia-fen,INOUYE M.Suppression of the negative effect of minor arginine codons on gene expression;preferential usage of minor codons within the first 25 codons of the Escherichia coli genes[J].Nucleic Acids Res,1990,18(6):1465-1473.
[10]LEE J H,SKOWRON P M,RUTKOWSKA S M,et al.Sequential amplification of cloned DNA as tandem multimers using class-IIS restriction enzymes[J].Genet Anal,1996,13(6):139-145.
[11]XIONG Yan-qiong,YEAMAN M R,ARNOLD S.In vitro antibacterial activities of platelet microbicidal protein and neutrophil defensin against S.aureus are influenced by antibiotics differing in mechanism of action[J].Antimicrob Agents Chemother,1999,43(5):1111-1117.
[12]FRIEDRICH C L,DIANNE M,BEVERIDGE T J,et al.Antibacterial action of structurally diverse cationic peptides on gram-positive bacteria[J].Antimicrob Agents Chemother,2000,44(8):2086-2092.
[13]BROGDEN K A.Antimicrobial peptides:Pore formers or metabolic inhibitors in bacteria? [J]Nat Rev Microbiol,2005,3(3):3238-3250.
[14]DHOPLE V,KRUKEMEYER A,RAMAMOORTHY A.The human beta-defensin-3,an antibacterial peptide with multiple biological functions[J].Biochimi Biophys Acta,2006,1758(9):1499-1512.
[15]SAHL H G,PAG U,BONNESS S.Mammalian defensins:structures and mechanism of antibiotic activity[J].J Leukoc Biol,2005,77(4):466-475.
[16]LEHRER R I.Multispecific myeloid defensins[J].Curr Opin Hematol,2007,14(1):16-21.
