生物序列比对算法的研究现状
2011-10-26杨洁刘海湖南信息职业技术学院410200
杨洁 刘海 湖南信息职业技术学院 410200
生物序列比对算法的研究现状
杨洁 刘海 湖南信息职业技术学院 410200
本文综述了生物序列比对的基本思想和主要方法。通过序列比较可以发现生物序列中的功能、结构和进化的信息。进行序列比对的目的是让人们能够判断两个序列之间是否具有足够的相似性,从而判定二者之间是否具有同源性。
生物序列;比对;算法
生物序列的比较是计算分子生物学或生物信息学中最基本的操作。其作用有:同源性的判断、相似性的搜索、功能区的预测、基因突变的判断、复制区域的判断等。在分子生物学中,序列之间的相似性是多方面的,可能是序列之间的相似,可能是结构的相似,也可能是功能的相似。一个普遍的规律是序列决定结构,结构决定功能。研究序列相似性的目的之一是通过相似的序列得到相似的结构或相似的功能,另一个目的是通过序列的相似性,判别序列之间的同源性,推测序列之间的进化关系。
序列比对有双序列比对和多序列比对之分,常见的双序列比对算法点阵图方法和动态规划算法,而多序列比对算法主要有渐进比对和迭代比对两大类。
1、点阵图法
点阵图法[1]的基本思想是通过将一条序列排在上端,另一条序列纵列在左端,两个序列在任何位置上若出现相同残基,就在两个序列对应位置上标注一个点,做成一个图。排列成对角线的点列体现出两条序列间具有相同的字符串,从而形象地表明序列间的相似性,双序列点阵图示如图1所示。
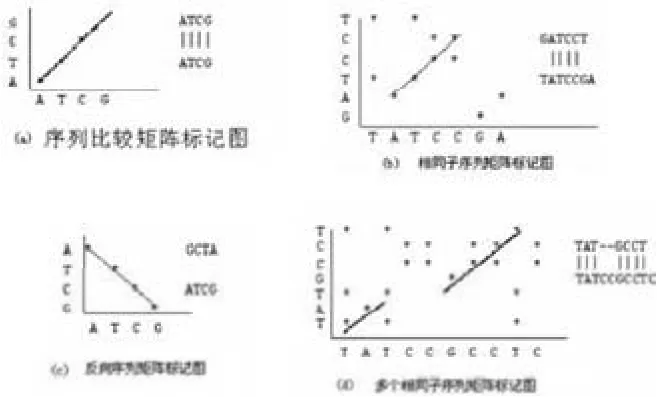
图1 双序列点阵图示
点阵图法的主要优点在于可以找到序列间的所有可能的残基匹配,但主要的局限是点阵计算机程序并不能显示真实的比对排列。
2、动态规划算法
动态规划算法主要有全局排列和局部排列两大类。
全局排列动态规划算法是由Needleman和Wunsch于1970年首先提出的[2],算法的基本思想是:用比对的两条序列构建一个相似打分矩阵S,矩阵中的元素可通过公式(1.1)获得。

例1:对序列a=GCTGATATAGCT,b=GGGTGATTAGCT,选择参数s(a,a)=1,s(a,b)=-1,插入删除单个字母的罚分为2,计算相似打分矩阵S如图2。

图2 相似打分矩阵S
根据上述矩阵可得最优比对如图3所示:

图3 例1中最优比对结果
局部排列动态规划算法是由Temple Smith 和 Michael Waterman 于1981年提出的,同样算法也是通过比对的两条序列构建一个相似打分矩阵(记为H),矩阵中的元素Hi,j可通过公式(1.2)获得。

这里,Hi,j是序列a在位置i和序列b在位置j的分值。w(ai,bj)是序列a位置i和序列b位置j上排列性状的分值。Wx是序列a中长度为x的间隔罚分,Wy是序列b中长度为y 的间隔罚分。
3、渐进比对算法
渐进比对算法属于启发式的多序列比对算法。最常见的渐进比对算法就是由Feng和Doolittle提出的Clustal算法,Clustal的基本思想是基于相似序列通常具有进化相关性这一假设。比对过程中,先对所有的序列进行两两比对并计算它们的相似性分数值,然后根据相似性分数值将它们分成若干组,并在每组之间进行比对,计算相似性分数值。根据相似性分数值继续分组比对,直到得到最终比对结果。比对过程中,相似程度较高的序列先进行比对,而相似性较低的序列则添加在后面。Clustal算法的主要三个步骤如下:
①两两比对:先将比对序列进行两两比对分别构建距离矩阵。
②系统发生树构建:根据计算所获得的距离矩阵构建系统发生树。
③进化式比对:对关系密切的序列进行加权,然后从紧密的两条序列开始,逐步引入临近的序列并不断重新构建比对,直到所有序列都被加入为止。
4、迭代比对算法
迭代比对算法的基本思想是基于一个比对算法,通过迭代方式精细多序列比对,直到比对结果不再改变为止。根据迭代策略的不同,迭代比对算法大致可分为Prrp法,隐马尔科夫法,模拟退火法和遗传算法等。
小结
进行序列比对的目的之一是让人们能够判断两个序列之间是否具有足够的相似性,从而判定二者之间是否具有同源性。值得注意的是,相似性和同源性虽然在某种程度上具有一致性,但它们是完全不同的两个概念:相似性是指一种很直接的数量关系,比如部分相同或相似的百分比或其它一些合适的度量;而同源性是指从一些数据中推断出的两个基因在进化上曾具有共同祖先的结论,它是质的判断。基因之间要么同源,要么不同源,绝不像相似性那样具有多或少的数量关系。
[1] Gilbert DG. Dot plot sequence comparisons on Macintosh computers. Comput. Appl.Biosci., 1990, 6(2): 117-117;
[2] Smith TF, Waterman MS. Identification of common molecular subsequences. J Mol Biol.1981. 147(1): 195-197.
10.3969/j.issn.1001-8972.2011.09.021
