甲型流感病毒DNA序列的长记忆ARFIMA模型*
2011-10-25刘娟高洁
刘 娟 高 洁
(江南大学理学院,无锡 214122)(2010年4月16日收到;2010年8月4日收到修改稿)
甲型流感病毒DNA序列的长记忆ARFIMA模型*
刘 娟 高 洁
(江南大学理学院,无锡 214122)(2010年4月16日收到;2010年8月4日收到修改稿)
流感病毒分为三类:甲型(A型),乙型(B型),丙型(C型).在这三种类型中甲型(A型)流感病毒是最致命的流感病毒,对人类引起了严重疾病.本文对甲型流感病毒DNA序列建立了一种新的时间序列模型,即CGR(Chaos Game Representation)弧度序列.利用CGR坐标将甲流病毒DNA序列转换成CGR弧度序列,且引入长记忆ARFIMA模型去拟合此类序列,发现随机找来的10条H1N1序列,10条H3N2序列都具有长相关性且拟合很好,并且还发现这两种序列可以尝试用不同的 ARFIMA模型去识别,其中 H1N1可用 ARFIMA(0,d,5)模型去识别,H3N2可用ARFIMA(1,d,1)模型去识别.
甲型流感,时间序列模型,CGR,ARFIMA(p,d,q)模型
PACS:87.10.Vg,02.50.Fz
1.引 言
流感是一种反复出现的传染病,在全球引起了高发病率和高死亡率[]1.流感病毒分为三类:甲型(A型),乙型(B型),丙型(C型).在这三种类型中甲型(A型)流感(以下简称甲流)病毒是最致命的流感病毒,给人类带来了严重的疾病.甲流病毒根据其表面的血凝素(hemagglutinin,HA)和神经氨酸酶(neuraminidase,NA)基因的不同又可分成16个HA亚型(H1-H16)和9个NA亚型(N1—N9),不同的HA和NA形成了甲流病毒的许多亚型,如H1N1,H3N2,H5N1 等等[2—4].笔者参看了许多文献几乎没有看到用时间序列模型来挖掘甲型流感病毒的特性的,因而本文采用时间序列模型来分析甲型流感病毒.
1992年,Peng等[5]提出了 DNA一维游走模型.同年Voss等[6]提出了不同的观点,他们发现 DNA序列的谱密度显示的1/fβ噪声无处不在,意味着当0<β<1存在长相关性,认为不仅在非编码区序列中在编码序列中也存在长相关性.另一方面Buldyrev 等[7,8]设计了一个广义-Lévy 游走模型去生成一个模型序列,使用所有可用的DNA序列发现主要在非编码序列中呈现长相关性.基于该模型,Tai等[9]提出了一个二维修正-Lévy游走模型.为区分 C和T,A和 G,Lou等建立了二维和三维游走模型[10],Yu 等则建立了图谱[11,12],来研究 DNA 序列的相关性.2006 年,Lopes和 Nunes[13]引入长记忆ARFIMA(0,d,0)模型去拟合 DNA序列的一维游走序列.2009年,Gao等[14]基于 CGR(chaos game representation)坐标提出了一种DNA序列转换成一个时间序列(CGR-游走序列)的方法,并引入长记忆ARFIMA(p,d,q)模型来分析.
本文对甲流病毒DNA序列提供了一种新的时间序列模型,即CGR弧度序列.利用CGR坐标将甲流病毒DNA序列转换成CGR弧度序列,且引入长记忆ARFIMA模型去拟合此类序列,发现随机找来的10条H1N1序列,10条H3N2序列都具有长相关性且拟合很好,并且还发现这两种序列可以尝试用不同的 ARFIMA模型去识别,其中 H1N1可用ARFIMA(0,d,5)模型去识别,H3N2可用 ARFIMA(1,d,1)模型去识别.
2.ARFIMA模型
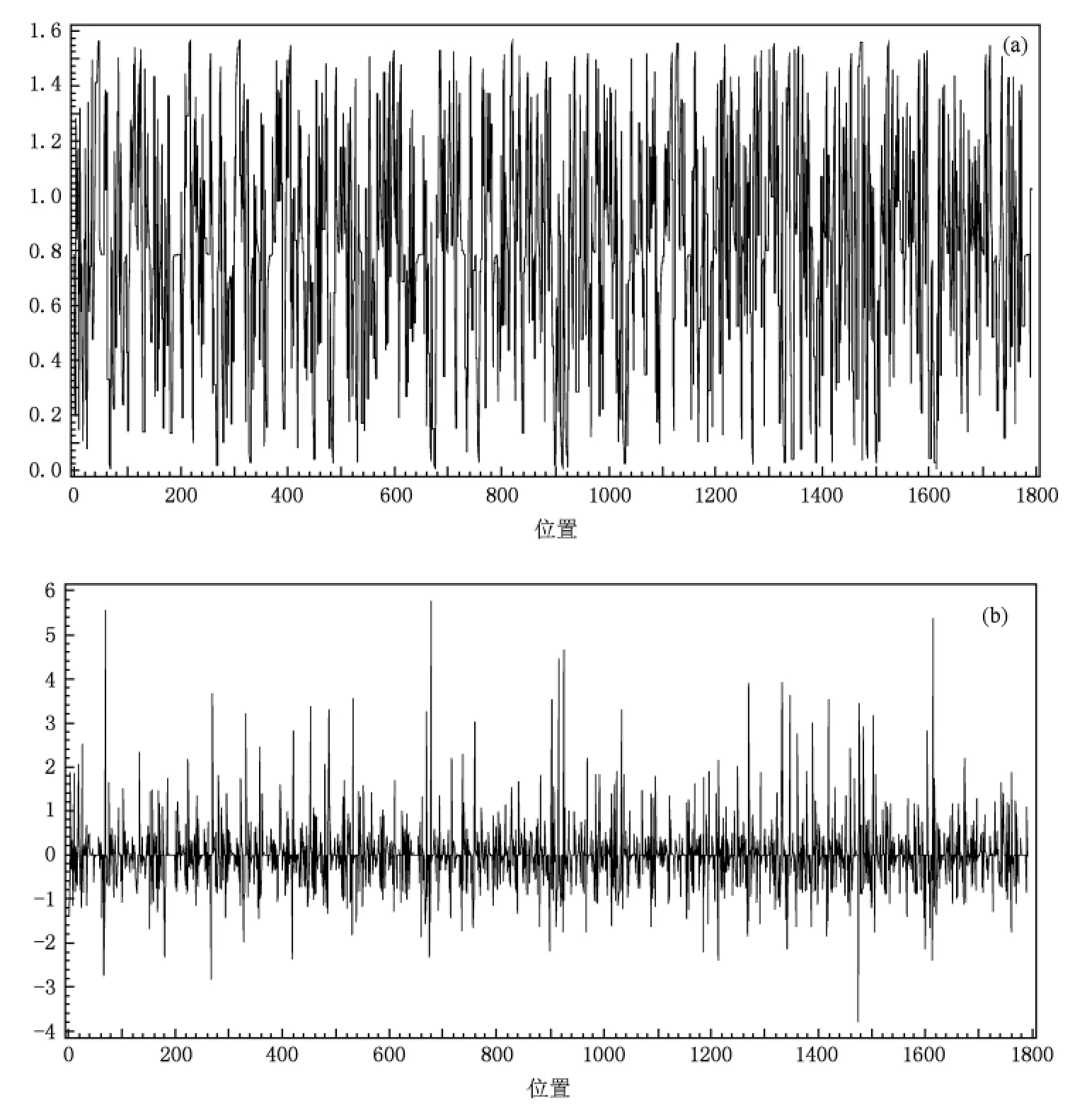
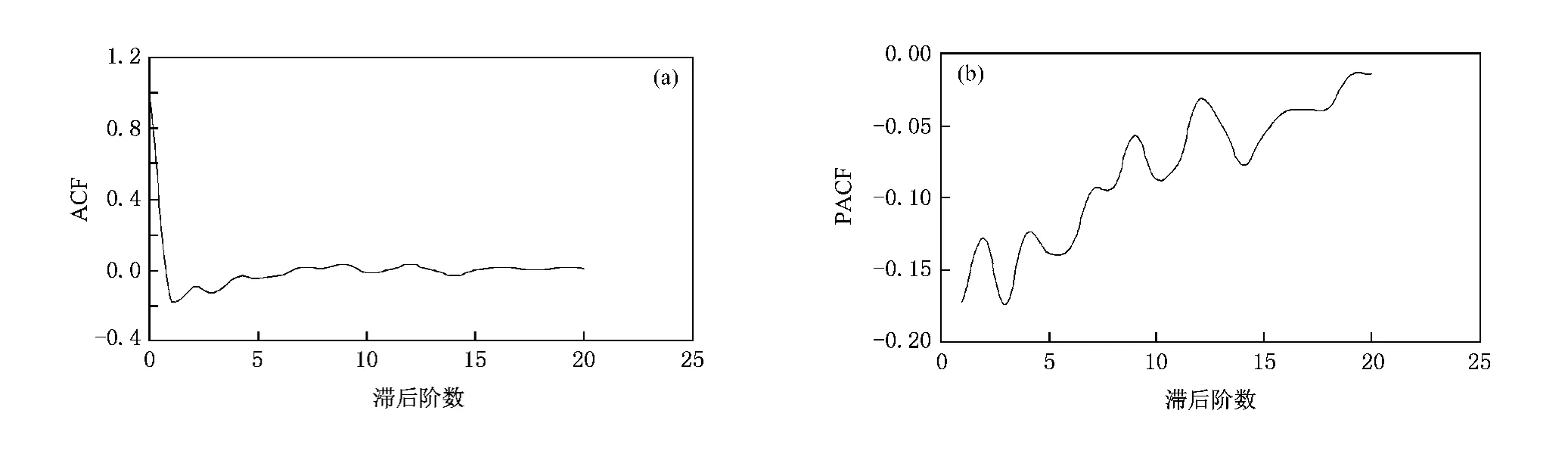
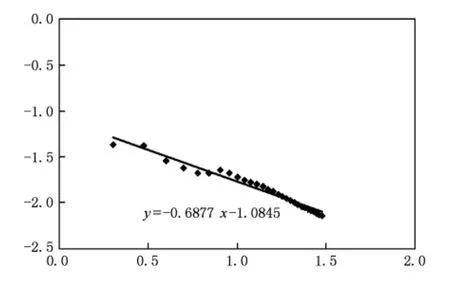
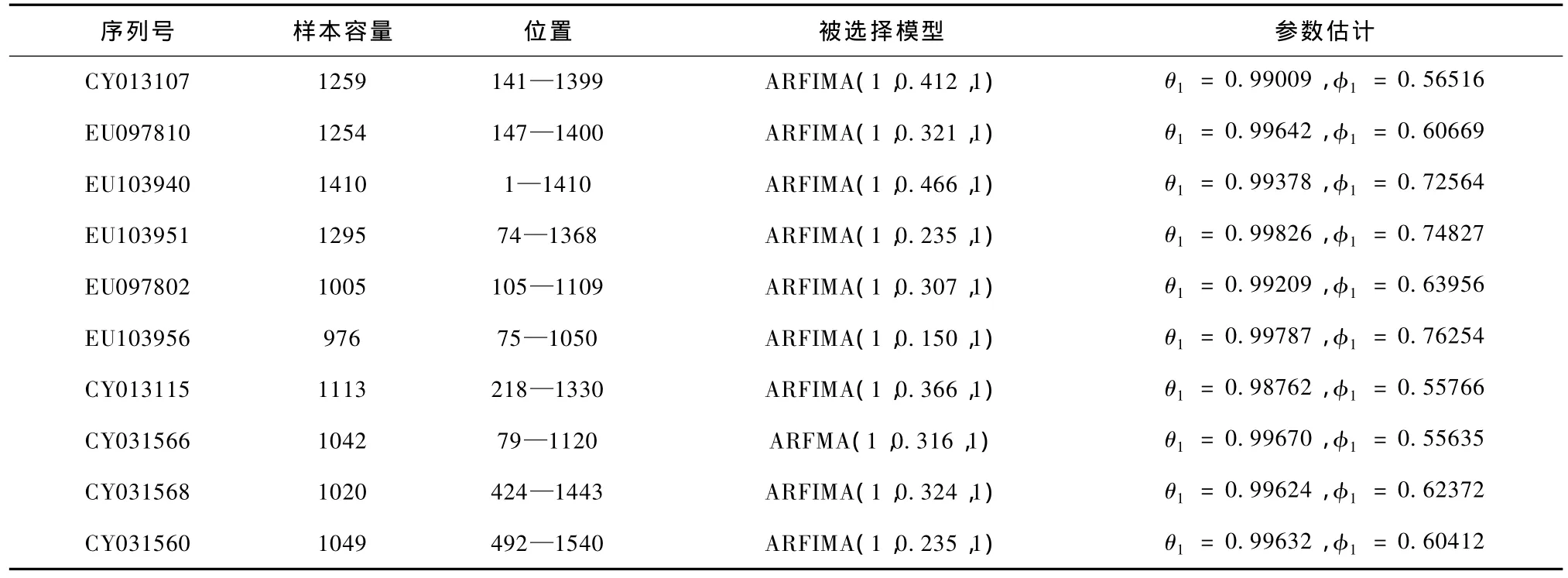
如果随机过程 {xt}是平稳的,且满足方程Φ(B)Δdxt= Θ(B)εt,其中,- 0.5 则称{xt}服从 -0.5 因此,{xt}可看作是分数差分噪声导出的 ARMA(p,q)过程.当2d-1=-1时,d=0即为短记忆过程;所以当2d-1>-1时,d∈(0,0.5)具有长记忆的特征. 1990年Jeffrey提出了一种 DNA序列可视化的方法即 CGR方法[15].CGR是一种迭代映射技术,它把序列中的每个单元,如蛋白质序列中氨基酸,DNA中的核苷酸,映射到一个连续的坐标空间中去. 正方形的四个顶点对应四种核苷酸.在这里,用DNA序列代替随机数,每一个碱基的坐标都可以来确定下一个碱基的位置.我们取 A(0,0),T(1,0),G(1,1),C(0,1),并且取点(0.5,0.5)为起始点. 下面给出DNA迭代函数,也可以认为是 CGR算法 的 公 式 化 形 式[15,16]. 对 于 一 个 序 列 S = 其中 gi={(0,0),(1,0),(1,1),(0,1)},gi和 si相对应. 对于一个DNA序列,定义 其中yn是CGRn的y坐标值,xn是 CGRn的 x坐标值.则得到一个数据序列 {Rn:n=1,2,…,N},我们把它作为一个时间序列,并称它为“CGR弧度序列”. 以甲流病毒 H1N1序列 CY056890为例,数据来自 NCBI网站,其网址:http://www.ncbi.nlm.nih.gov/. 它的CGR弧度序列“游走图”如下表1. 表1 CY056890序列所选部分前8个ACATGGTA游走结果 图1(a)是CY056890序列CGR弧度序列图(位置380—2170),样本容量1791.这些数据变动较大,呈现非平稳特征.考虑对此过程作d阶差分.先对原序列作对数变换然后再做一阶差分结果如图1(b)所示,可见除少数地方呈现异方差外,基本呈现平稳性. 图2(a)(ACF)和图2(b)(PACF)为样本取对数再一阶差分后的自相关函数图形和偏自相关函数图形.可见ACF衰减迅速,而PACF衰减缓慢,这意味着原序列具有长记忆特征. 图1 (a)甲流H1N1型病毒CY056890的弧度序列图;(b)取对数再一阶差分图 图2 (a)取对数再一阶差分的样本自相关图;(b)取对数再一阶差分的样本偏自相关图 图3给出了方差图[17]是一个估计长记忆参数d的有用工具.对于一个长记忆时间序列{Rn},它的均值珔Rk的方差满足作log[Var(珔Rk)]关于log(k)的散点图,对散点图线性拟合,可估计得到线性方程的斜率为 -0.6877,令该斜率为2d-1= -0.6877,即可得 d的估计值0.156. 根据上述理由我们可选择CGR弧度序列显示长记忆特征.目的是利用上述特点为序列建立一个合适的模型.因此,可以考虑长记忆 ARFIMA(p,d,q)模型(d∈ (0,0.5)),p,q定阶时为考虑实用性,仅考虑 p,q均小于等于5的 ARFIMA(p,d,q)模型.由 Akaike 信 息 判 别 准 则[18,19],可 选 ARFIMA(0,0.156,5)模型来拟合. 为检验该模型的合理性,选择了一个合适的检验统计量 LB 检验统计量[20,21] 其中rk是滞后k的样本自相关函数,n是样本容量,M是一个取定的比n小的正整数. 表2显示了对于各滞后阶数,LB统计量的p值均显著大于0.1,意味着拟合模型的残差序列应为白噪声(纯随机),因而可以认为 ARFIMA(0,0.156,5)模型能很合理地拟合 CY056890序列的CGR-游走序列. 图3 CY056890DNA序列的CGR弧度序列方差图 表2 残差的自相关检验 表3给出了被选择的 ARFIMA(0,0.156,5)模型的参数估计,5个参数的 T检验统计量的p值均显著小于 0.005.这意味着 ARFIMA(0,0.156,5)模型能有效地拟合这个CGR弧度序列. 表3 条件最小二乘估计 表4和表5分别给出了随机选的9条H1N1序列和10条 H3N2序列的数据信息、被选择的ARFIMA(p,d,q)模型及参数估计.从计算结果可得d均位于(0,0.5);对于各滞后阶数,LB统计量的 p值除极个别外其余均显著大于0.1;且每个被选择的模型中各参数的T检验统计量的p值均显著小于0.01.所有这些结果都显示ARFIMA(p,d,q)模型能很合理很有效地拟合这些不同的CGR弧度序列且还发现所选H1N1序列均为ARFIMA(0,d,5)模型,所选 H3N2序列均为 ARFIMA(1,d,1)模型.所以我们可尝试用 ARFIMA(0,d,5)模型,ARFIMA(1,d,1)模型分别去识别H1N1序列,H3N2序列. 表4 9条H1N1序列的数据信息、被选择的ARFIMA模型和参数估计 表5 10条H3N2序列的数据信息、被选择的ARFIMA模型和参数估计 本文基于CGR坐标提出了一种将甲流病毒DNA序列转换成时间序列(CGR弧度序列)的方法,并引入长记忆模型ARFIMA模型来分析,首先分析了甲流H1N1型病毒CY056890序列,从图1到图3可知弧度序列显示长记忆特征,并选择了ARFIMA(0,0.156,5)模型去拟合它,从表 2到表 3发现拟合合理有效. 然后又分析了随机找来的19条序列的CGR弧度序列,从表4和表5可知所有 ARFIMA(p,d,q)模型都有效合理.并且从表4中还发现所选H1N1序列均为 ARFIMA(0,d,5)模型,表5所选 H3N2序列均为 ARFIMA(1,d,1)模型. 由此可见,DNA序列的CGR弧度序列能由长记忆ARFIMA(p,d,q)模型有效合理地拟合,并且还可尝试用 ARFIMA(0,d,5)模型,ARFIMA(1,d,1)模型分别去识别H1N1序列、H3N2序列.作为具有完善算法的经典时间序列模型,不仅可以帮助我们得到甲流病毒DNA序列清晰的结构,而且还可帮助我们有效识别甲流中的两种亚型. 本文仅对甲流中的两种亚型进行了研究分析,后面我们将研究分析甲流中的其他亚型以及乙型丙型流感病毒. [1] Morens D,Folkers G,Fauci A 2004 Nature 430 242 [2] Chen J M,Sun Y X,Liu S 2009 Chinese Science Bulletin 54 1657(in Chinese)[陈继明、孙映雪、刘 朔 2009科学通报54 1657] [3] Webster R G,Bean W J,Gorman O T 1992 Microbiol Rev.56 152 [4] Shi X M,Shi L,Zhang J F 2010 Chin.Phys.B 19 038701 [5] Peng C K,Buldyrev S,Goldberg A L,Havlin S,Sciortino F,Simons M,Stanley H E 1992 Nature 356 168 [6] Voss R F 1992 Phys.Rev.Lett.68 3805 [7] Buldyrev S V,Goldberger A L,Havlin S,Peng C K,Simon M,Stanley H E 1993 Phys.Rev.E 47 4514 [8] Buldyrev S V,Goldberger A L,Havlin S,Mantegna R N,Matsa ME,Peng C K,Simon M,Stanley H E 1995 Phys.Rev.E 51 5084 [9] Tai Y Y,Li P C,Tseng H C 2006 Physica A 369 688 [10] Luo L F,Lee W J,Jia L J,Ji F M,Tsai L 1998 Phys.Rev.E 58 861 [11] Yu Z G,Chen G Y 2000 Theor.Phys.33 673 [12] Yu Z G,Anh V,Gong Z M,Long S C 2002 Chin.Phys.11 1313 [13] Lopes S R C,Nunes M A 2006 Physica A 361 569 [14] Gao J,Xu Z Y 2009 Chin.Phys.B 18 370 [15] Jeffrey H J 1990 Nucleic Acid Res 18 2163 [16] AlmeidaJonas, carrico Joao A, Maretzek António 2001 Bioinformatics 17 429 [17] Beran J 1994 Statistics for long-memory Processes(New Work:Chapman Hall) [18] Hosking J R M 1984 Water Resour.Res.20 1898 [19] Crato N,Ray B K 1996 Journal of Forcasting 15 107 [20] Ljung G M,Box G E P 1978 Biometrika 65 297 [21] Li W K,Mcleod A I 1986 Biometrika 73 217 Long-memory ARFIMA model for DNA sequences of influenza A virus* Liu Juan Gao Jie Influenza viruses are divided into three types:A,B and C.Among them,type A virus is the most virulent human pathogen and causes the most severe disease.In this paper,we propose a new time series model for influenza A virus DNA sequence,i.e.chaos game representation(CGR)radians series.The CGR coordinates are converted into a time series model,and a long-memory ARFIMA(p,d,q)model is introduced to simulate the time series model.We select randomly 10 H1N1 sequences and 10 H3N2 sequences in analysis.we find in these data a remarkably long-range correlation and fit the model reasonably by ARFIMA(p,d,q)model,and also find that we can use different ARFIMA models to identify the two kinds of sequences,i.e.ARFIMA(0,d,5)model and ARFIMA(1,d,1)model that can identify H1N1 and H3N2 respectively. influenza A virus,time series model,chaos game representation(CGR),ARFIMA(p,d,q)model .E-mail:ezhun6669@sina.com *江南大学创新团队发展计划(批准号:2008CX002)中央高校基本科研业务经费专项资金(批准号:JUSRP21117)资助的课题. .E-mail:ezhun6669@sina.com *Project supported by the Innovative Research Team of Jiangnan University(Grant No.2008CX002)the Foundamental Research Founds for the Central Universities(Grant No.JUSRP21117). PACS:87.10.Vg,02.50.Fz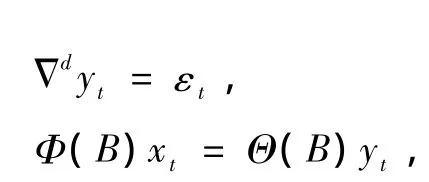
3.基于CGR的时间序列模型
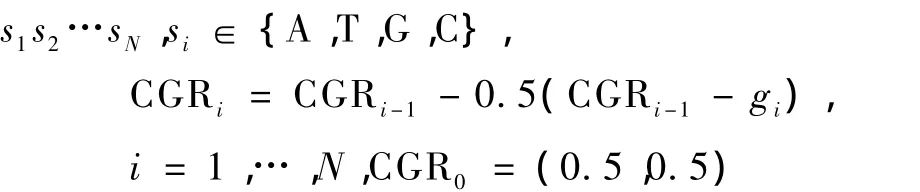


4.甲流 H1N1型病毒 CY056890的数据分析




5.其余9条H1N1序列和10条H3N2序列数据分析

6.结 论
(School of Science,Jiangnan University,Wuxi 214122,China)(Received 16 April 2010;revised manuscript received 4 August 2010)
