海南岛东部501站位沉积物古细菌多样性研究
2011-09-24刘国辉吴后波
刘国辉, 吴后波, 李 翔, 颜 文
海南岛东部501站位沉积物古细菌多样性研究
刘国辉1,4, 吴后波1, 李 翔1, 颜 文2,3
(1. 中国科学院 南海海洋研究所LMB实验室, 广东 广州 510301; 2. 中国科学院 广州天然气水合物研究中心, 广东 广州 510070; 3. 中国科学院 边缘海重点实验室, 广东 广州 510301; 4. 中国科学院 研究生院,北京 100049)
从海南岛东部501站位沉积物中扩增古菌16S rRNA基因构建文库, 并对得到的OTU(Operation taxonomic units)进行系统发育学分析。古菌文库由泉古生菌(Crenarchaeota)和广古生菌(Euryarchaeota)构成, 其中泉古生菌占总克隆文库的 96.8%, 由Ⅰ型海洋泉古生菌(Mraine crenarchaeotic groupⅠ,MGⅠ, 74.6%)、GROUP A(14.9%)和GROUP B(7.2%)三簇组成。广古生菌GROUP C簇占总克隆文库的3.2%, 序列比对结果显示它们与甲烷微菌纲(Methanomicrobial)中甲烷厌氧氧化菌(ANME)和甲烷八叠球菌目(Methanosarcinales)有较近的系统发育学关系。研究表明海南岛东南部近岸沉积物古菌多样性比较丰富, 泉古生菌是该站位古菌的优势菌。
海洋沉积物; 16S rDNA; 古菌
海洋约占地球表面积的 71%, 绝大部分海底被海洋沉积物所覆盖。随着对海洋研究的深入, 人类逐渐意识到海洋在全球物质循环和能量流动中的重要地位。海洋沉积物是海洋的最主要组成部分之一, 它是地球岩石圈和生物圈之间各元素生物地球化学循环过程的中枢。据估计, 海洋沉积物所蕴含的生物量至少占到地球总生物量的1/10~1/3[1]。自从在海洋环境中发现丰富的海洋古细菌[2], 人们在生物圈的多个生境发现古菌的踪影, 促使人们改变以往的古菌只存在于极端环境的观点, 古菌分布的广泛性得到普遍的共识。由于古菌细胞外壳较细菌细胞更难于破解, 古菌细胞内16S核糖体RNA基因拷贝数低于细菌等诸多因素影响[3], 通常导致对古菌定量分析造成较大偏差。最近, Julius[4]在全球海洋范围内的多个站位采集沉积物样品, 以细胞膜磷脂类大分子为生物分子标记物, 采用的定量 PCR和探针杂交为辅助手段, 证明古菌生物量占总沉积物原核生物量的绝大部分。
赋存于环境条件复杂的海底生境, 致使海洋古菌结构和功能表现出复杂的多样性。只有全面了解古菌的多样性, 才能理解古菌在生命进化过程的地位和各种元素生命地球化学循环过程中的作用。目前, 可以培养的古菌种类稀少, 非培养的分子生物学方法是研究古菌多样性最主要的手段。原核生物16S rRNA基因所具有的特性, 使之成为系统发育分析天然的标签[5]。16S rRNA基因克隆文库、荧光原位杂交(FISH)以及变性梯度凝胶电泳(DGGE)是目前研究微生物多样性比较普遍的方法。FISH技术操作相对比较简单高效, 其局限性是受数据库已知序列影响比较大。16S rRNA基因克隆文库和DGGE方法在 PCR的扩增的过程中出现部分偏差, 但这并不影响它们作为研究环境微生物多样性最重要的手段。
作者构建海南岛东南部近岸沉积物样品的古菌16S rRNA基因文库和系统发育树来分析该样品古菌的多样性。
1 材料和方法
1.1 材料
1.1.1 沉积物样品的采集
2007年8月南海北部海洋观测开放航次的抓斗取样, 采自海南岛东部近岸的501站位(18°59.995′N,110°41.835′E), 水深 74 m, 沉积物为黑色的砂质样品, 采集后−20°C 保存。
1.1.2 主要试剂和仪器
PCR扩增仪(BIO-RAD), DNA回收纯化试剂盒(TAKARA), 电泳仪(广州正一), 内切酶(TAKARA),凝胶成像仪(BIO-RAD), PMD18-T(TAKARA), 感受态 DH5α(TAKARA)。
1.2 DNA的提取和纯化
沉积物总DNA的提取, 在Zhou[6]的文献提供的方法基础上加以改进, 本文具体方法为: 取2 g沉积物样品, 加入 6 mL DNA提取缓冲液[100 mmol/L Tris-HCl(pH8.0), 100 mmol/L Na2-EDTA(pH8.0), 1.5 mol/L NaCl, 0.5% CTAB, 1% PVPP, 100 mmol/L Na3PO4和200 mg/L 蛋白酶K], 涡旋3 min后, 在摇床水浴振荡30 min(37℃, 225 r/min), 加入溶菌酶(终质量浓度1 g/L)后振荡30 min, 然后加入1 mL 20%SDS和该提取液体积 100 mg/L浓度的蛋白酶 K,经60℃水浴300 min, 6 000g离心10 min, 将上清液移入新的离心管。沉淀重复抽提2次, 收集3次上清液于同一离心管中, 加入等体积的酚︰氯仿︰异戊醇(25︰24︰1)混匀, 9 000g离心25 min, 将上层水相移入新的离心管, 再加入等体积氯仿︰异戊醇(24︰1), 9000g离心 25 min, 将上层水相移入新的离心管。加入0.6倍体积异丙醇, 于-20℃沉淀60 min。在4℃, 9 000g离心25 min, 弃上清, 沉淀用70%的乙醇洗两次, 吹干。溶解于100 µL TE溶液。琼脂糖电泳切胶回收, 试剂盒纯化。
1.3 PCR扩增
采用古菌通用引物 21F: 5′-TTCCGGTTGATCCYGCCGGA-3′和 958R: 5′-YCCGGCGTTGAMTCCAATT-3′。50 µL PCR 反应体系: 25 µL Extaq(Takara),顺反引物各 2 µL, 模板 1 µL, 用双蒸水补足至 50µL。PCR 条件:3 min, 94 ℃ 30 s, 55℃ 45 s,72℃ 1 min, 25个循环, 72℃ 延伸10 min。扩增完毕后采用 Reconditioning PCR[7]: 取十分之一的 16S rRNA基因PCR产物为模板, 用与上次PCR扩增相同的反应体系进行3个循环扩增。
1.4 16S rDNA文库的构建和RFLP分析
将 Reconditioning PCR得到的扩增产物克隆到T-Vector, 转化到大肠杆菌 DH5α感受态细胞里, 进行蓝白斑筛选, 通过菌落 PCR的方法筛选阳性克隆子。扩增的插入片段分别用 HhaⅠ和 HinfⅠ这两种内切酶进行完全酶切, 用3%的琼脂糖凝胶电泳检测,分析两种酶切后条带的谱型, 对不同的谱型进行分类统计并测序。
1.5 古菌16S rDNA序列分析
2 结果
2.1 16S rDNA文库分析
随机挑取280个阳性克隆子扩增产物进行酶切,其中HhaⅠ酶切得到32个带型, HinfⅠ酶切得到22个带型, 对两种酶切结果进行统计分析后共得到 58个带型。测序后经在线嵌合体检验, 剔除嵌合体序列,对剩余的序列进行Blast相似性比较, 把相似性大于97%归于同一OTU, 最后得到221个有效克隆子, 分属38个OTU, 这表明该站位沉积物中古菌多样性是相当丰富的。得到文库的覆盖率为 92.3%,该数值表明构建的 16S rRNA基因文库可以反映沉积物样品的多样性; 在线得到的稀释曲线(图 1), 图中随着系统随机取样数增多, 曲线上升趋势越来越缓慢, 显示该 16S rRNA基因文库基本能满足样品古菌多样性分析的需要。
2.2 古菌16S rDNA文库多样性
16S rRNA基因文库构建的系统发育树表明: 沉积物中古菌被分为两大类, 泉古生菌(Crenarchaeota)和广古生菌(Euryarchaeota), 其中泉古生菌含有 33个OTU, 占总克隆文库数目的96.8%; 广古菌含有5个OTU,仅占总克隆子数目的3.2%。泉古生菌主要分为 3簇, 主要为Ⅰ型海洋泉古生菌(Mraine crenar-chaeotic group Ⅰ , MG Ⅰ ), 占 74.6%; GROUP A,占14.9%; GROUP B,占 7.2%(图 2)。广古生菌簇 GROUP C与甲烷微菌纲(Methanomicrobial)中甲烷厌氧氧化菌(ANME)和甲烷八叠球菌目(Methanosarcinales)有较近的系统发育学关系(图3)。
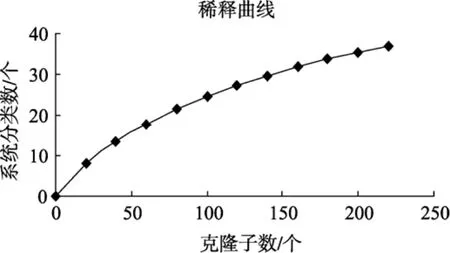
图1 海南岛东南部沉积物古菌16S rRNA基因rarefaction曲线Fig. 1 Rarefaction curve of archaeal 16S rRNA sequences from the sediment of southeastern Hainan island
Ⅰ型海洋泉古生菌共有 15个 OTU, 其中 8个OTU与来自印度的热带河口、珠江河口以及红树林沉积物的序列有很高的相似性(96%~99%), 而且有6个OTU序列的相似性高达到99%, 这8个OTU共包含 147克隆子, 占总克隆文库 66.5%, HND501-AC01序列与已完成全基因组测序海洋铵氧化古菌株(Nitrosopumilus maritimusSCM1)具有99%的相似性; 这一簇的另外7个OUT虽然仅占总克隆子文库的 8.1%但是与它们相似序列分布范围比较广, 日本海洋热泉微生物席浅层沉积物、北太平洋3 000 m深灰泥沉积物、维多利亚港西侧沉积物, 韩国沿岸海域海绵组织共生菌。GROUP B簇中序列仅有HND501-AC67序列与来自印度洋Arabian海沿岸海水中序列有 98%的相似性, 其余的序列都与来自红树林沉积物、珠江河口、印度的热带河口的序列有90%~98%的相似性。GROUP B中所包含的序列信息表明这一簇明显属于海陆交界的一个类群, 这一簇中仅有的一个序列与沿岸海水中采集到的序列相似, 这可能是来自红树林或河口的泉古菌在沿岸海水中进化适应性的结果。与GROUP A簇有93%~99%相似性的序列的生境分布范围非常广泛, 分别来自印度的热带河口、鄂霍茨克海峡甲烷冷泉区、地中海泥火山口、北冰洋沉积物等。
GROUP C中HND501-AE76与来自印度洋中脊不活跃海底热液烟囱的序列有 97%的相似性,HND501-AE60和HND501-AE14都与来自热带河口沉积物的序列有96%的相似性。HND501-AE40与来自甲烷冷泉沉积物的序列有 87%的相似性,HND501-AE39与来自海南红树林样品中序列有98%的相似性。
3 讨论
海洋沉积物, 特别是近海沉积物样品中, 含有大量腐殖酸、褐菌素和重金属离子等其他杂质, 这些杂质极大地影响最终的分析结果, 为了最大化消除它们对后续实验的影响, 采用 EDTA 有效络合重金属离子, 抑制核酸酶的活性, 并采用CTAB和PVPP有效地降低腐殖酸的浓度。在沉积物总DNA提取过程中先后两次加入蛋白酶 K, 总质量浓度为 300 mg/L, 溶菌酶和蛋白酶 K的使用大大提高了古菌的破膜效率, 提高了总DNA提取的数量。在PCR反应进入平台期后, 引物的量急剧减少, 造成序列相近的模板之间退火几率增加, 形成异源双链。本实验采用 Reconditioning PCR方法能有效地避免异源双链结构的产生。
Ⅰ型海洋泉古生菌是Delong首先发现于海洋环境中[2], 后来研究表明它广泛分布于海水和海洋沉积物环境中。Ⅰ型海洋泉古生菌在海水中含量丰富,其多样性化程度也非常高, 可能具有多样的代谢类型。隶属于Ⅰ型海洋泉古生菌海洋氨氧化古菌最近被分离纯化[8], 这一类具有硝化作用的古菌, 广泛分布于海水和沉积物上表面低氧和低氨的环境中, 在全球氮循环过程中具有关键的作用。从样品中得到的序列与硝化古菌有较高的相似性, 说明该站位也存在氨的硝化过程。该站位得到的泉古菌序列占所得总古菌序列的绝大部分, 尤其是Ⅰ型海洋泉古生菌含量最为丰富, 可以推测泉古菌是该站位的优势古菌, 泉古菌可能在该站位的碳、氮元素生物地球化学循环中占有十分重要的地位。
甲烷生成菌是一类严格厌氧, 能利用多种底物生成甲烷的古菌。甲烷氧化菌以甲烷为唯一的能源和碳源, 在减缓温室效应和全球碳循环过程中有重要作用。海洋沉积物释放的甲烷主要被海底厌氧甲烷氧化古菌吸收。以往研究表明, 厌氧甲烷氧化菌主要有三个系统发育分枝(ANME-1、2、3)都是与产甲烷古菌有较近的同源关系, 在世界范围多种沉积物环境采集到的样品表明, 厌氧甲烷氧化菌大多与硫细菌共生[9]。甲烷生成菌已经被纯培养, 它的甲烷生成分子机理已经得到很好的阐释, 海底厌氧甲烷氧化菌至今未得到纯化, 所以对它的代谢机制还不是很清楚。在厌氧甲烷氧化菌中探测到甲烷生成菌最后一步关键酶甲基辅酶M还原酶A(McrA)的基因序列[10], 为厌氧甲烷氧化是甲烷生成的逆过程的假说提供了线索。我们在 HND501站位得到的序列属于甲烷微菌纲, 从构建的系统发育树上可以看出(图3),GROUP C的序列与ANME-1有更近的亲缘关系, 暗示这些古菌可能与甲烷代谢相关。我们可以推测, 由于这个站位比较近岸, 陆地的有机物输入与上层海水较高的生产力, 沉积物表层有机物含量非常丰富,表层 O2、NO3−、金属氧化物依次作为电子受体被好氧细菌、硝化细菌等大量微生物很快消耗殆尽, 造成SO42−和CO2为电子受体的下表层具有很低的氧化还原电位, 这个环境为厌氧甲烷氧化和甲烷生成古菌生长创造有利的条件。
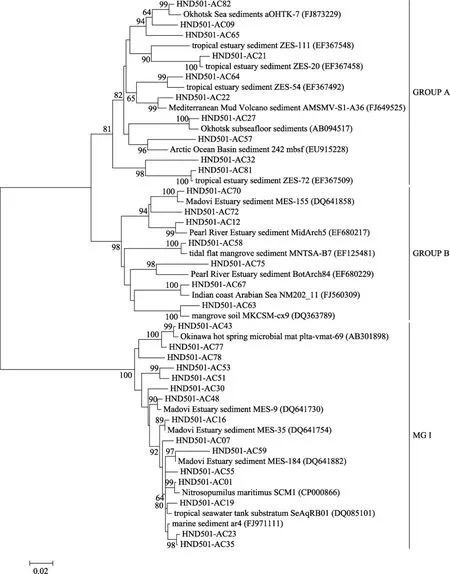
图2 海南岛东南部沉积物泉古菌16S rRNA基因系统发育树Fig. 2 Phylogenetic tree of crenarchaeota 16S rRNA clones from the sediment of southeastern Hainan island
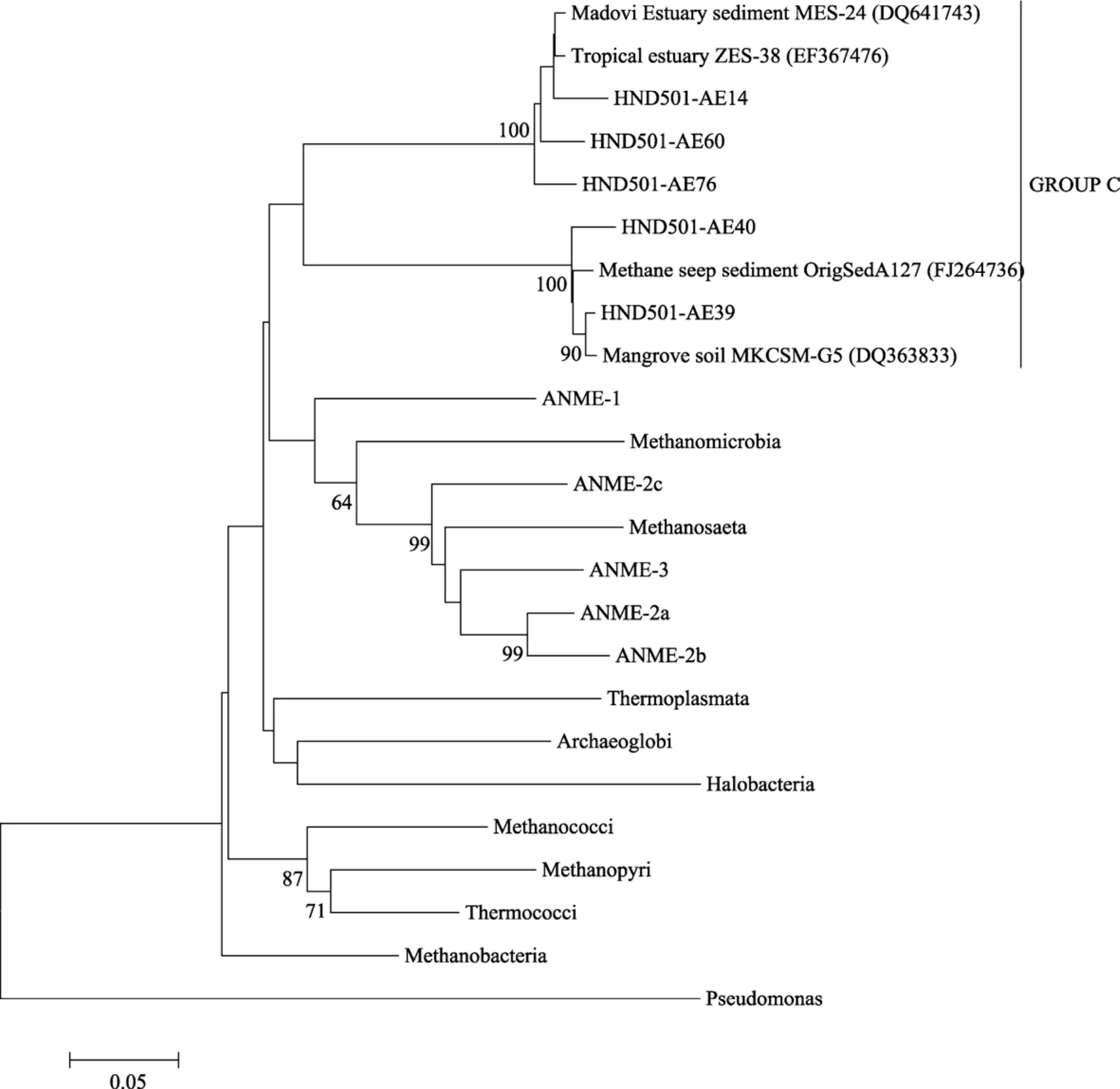
图3 海南岛东南部沉积物广古菌16S rRNA基因系统发育树Fig. 3 Phylogenetic tree of euryarchaeota 16S rRNA clones from the sediment of southeastern Hainan island
将该站位得到的古菌多样性结果分别与我国南海范围内河口、红树林和深海沉积物古菌多样性进行横向比较, 发现珠江河口淇澳岛海岸带沉积物[11]、海南岛红树林土壤[12]与本站位得到的结果十分相似:泉生古菌是这些沉积物环境古菌中的优势菌, 这些序列本身相似性很高, 易成簇, 高度同源; 序列比对结果显示, 地理相近或环境相似来源序列的相似性高, 这些序列数目也大多占优势; 广古生菌只占很少一部分, 相关序列大多与甲烷代谢相关。南沙陆坡的深海沉积物[13]所得到泉古生菌和广古生菌数量相当, 其中广古生菌多样性也比近海沉积物丰富, 泉古生菌和广古生菌大多与深海生境得到的序列有很高的相似性。由此可以看出我国南海沉积物古菌多样性和分布具有明显的地理特色。
[1] Parkes R, Cragg B, Wellsbury P. Recent studies on bacterial populations and processes in subseafloorsediments: a review[J]. Hydrogeology Journal, 2000, 8:11-28.
[2] Delong E F. Archaea in coastal marine environments[J].Proc Natl Acad Sci USA, 1992, 89: 5 685-5 689.
[3] Acinas S G, Marcelino L A, Klepac-Ceraj V, et al.Divergence and redundancy of 16s rRNA sequences in genomes with multiple rrn operons[J]. Journal of Bacteriology, 2004, 186: 2 629-2 635.
[4] Julius S L, Yuki M, Fumio I, et al. Significant contribution of archaea to extant biomass in marine subsurface sediments[J]. Nature, 2008, 454: 991-994.
[5] Susannah G, TandPhilip H. A renaissance for the pioneering 16s rRNA gene[J]. Current Opinion in Biotechnology, 2008, 11: 442-446.
[6] Zhou J, Bruns M A, Tiedje J M. DNA recovery from soils of diverse composition[J]. Applied and Environmental Microbiology, 1996, 62: 316-322.
[7] Thompson J R, Marcelino L A, Polz M F. Heteroduplexes in mixed-template amplifications: Formation,consequence and elimination by “reconditioning pcr”[J].Nucleic Acids Res, 2002, 30: 2 083-2 088.
[8] Martin K, Anne E B, Jose R, et al. Isolation of an autotrophic ammonia-oxidizing marine archaeon[J]. Nature,2005, 437: 543-546.
[9] Boetius A, Ravenschlag K, Schubert C J, et al.A marine microbial consortium apparently mediating anaerobic oxidation of methane[J]. Nature, 2000, 407:623-626.
[10] Steven J H, Peter R G, Christina M P, et al. Identification of methyl coenzyme m reductase a (mcra) genes associated with methane-oxidizing archaea[J]. Applied and Environmental Microbiology, 2003, 69: 5 483-5 491.
[11] 姜丽晶, 彭晓彤, 周怀阳. 非培养手段分析珠江口淇澳岛海岸带沉积物中的古菌多样性[J]. 海洋科学,2008, 30(4): 114-122.
[12] Yan B, Hong K, Yu Z N. Archaeal communities in mangrove soil characterized by 16s rrna gene clones[J]. The Journal of Microbiology, 2006, 44: 566-571.
[13] 李涛, 王鹏, 汪品先. 南海南部陆坡表层沉积物细菌和古菌多样性[J]. 微生物学报, 2008, 48(3): 323-329.
Received:Jul., 26, 2010
Key words:marine subseafloor sediment; archaea
Abstract:Diversity of archaea was studied in marine sediment by PCR amplification and sequence analysis of 16S rRNA. Samples were from a site at the east of Hainan island. The phylogenetic analysis revealed that archaeal population was composed of Crenarchaeota and Euryarchaeota. Mraine crenarchaeotic group Ⅰ(74.6%), GROUP A(14.9%) and GROUP B(7.2%) fall into Crenarchaeota. The GROUP C sequences were closely related to ANME and Methanosarcinales. The result indicates that archaea are diversified in surface environment of subseafloor sediment and Crenarchaeota is the predominant aechaea.
(本文编辑:梁德海)
Phylogenetic analysis of archaeal community from the sediment at location 501 at the east of Hainan island
LIU Guo-hui1,4, WU Hou-bo1, LI Xiang1, YAN Wen2,3
(1.South China Sea Institute of Oceanology, LMB, The Chinese Academy of Sciences, Guangzhou 510301,China; 2. Guangzhou Center for Gas Hydrate Research, The Chinese Academy of Sciences, Guangzhou 510070, China; 3. Key Lab. of Marginal Sea Geology, South China Sea Institute and Guangzhou Institute of Geochemistry, The Chinese Academy of Sciences, Guangzhou 510301, China; 4. Graduate University of Chinese Academy of Sciences, Beijing 100049, China)
Q938
A
1000-3096(2011)02-0086-06
2010-07-26;
2010-09-21
中国科学院知识创新工程重要方向项目(KZCX2-YW-211-03); 中国科学院南海海洋研究所创新领域前沿项目(lyqy200312)
刘国辉(1982-), 男, 山东烟台人, 在读硕士研究生, 主要从事海洋环境与分子微生物学研究, E-mail:wsw@scsio.ac.cn; 吴后波, 通信作者, 020-89023162, E-mail: wuhoubo@scsio.ac.cn
