依存信息在蛋白质关系抽取中的作用
2011-06-28钱龙华周国栋
刘 兵,钱龙华,徐 华,周国栋
(苏州大学 计算机科学与技术学院,江苏 苏州 215006;苏州大学 自然语言处理实验室,江苏 苏州 215006)
1 引言
蛋白质作为最主要的生命活动载体和功能执行者,其复杂多样的结构功能、相互作用和动态变化能在分子、细胞和生物体等多个层次上全面揭示生命现象。对生命活动过程中蛋白质相互作用关系(PPI, Protein-Protein Interaction)的研究有助于揭示生命过程的许多本质问题,因而PPI抽取成为生物医学领域信息抽取的重点研究方向,是生物医学文本挖掘的主要任务之一,具有重要的研究意义。
目前,计算机辅助的自动文本挖掘技术可以快速获取医学信息并将其构建为计算机可操作的知识库,方便数据分析和挖掘。这些方法大致可分为三类:基于共现的方法、基于规则的方法和基于机器学习的方法,其中基于机器学习的方法又可分为基于特征向量的方法和基于核函数的方法。
基于共现的方法通过简单地统计两个蛋白质的共现次数来预测两个蛋白质之间的关系,因此它的明显缺陷就是只能抽取一些频繁出现的PPI模式,并且准确率也比较有限。
基于规则的方法采用一些预先定义的词或短语模式规则来匹配可能出现的PPI关系。然而,由于PPI关系模式的多样性,预定义的规则不可能包含所有的PPI关系模式。并且在新的领域使用基于规则的方法时,这些规则都需要修正。这种方法可以获得较高的准确率,但召回率非常低。
基于机器学习的方法是目前主流的PPI抽取策略,其中基于特征向量的方法近年来得到了广泛应用。在以往的研究中,Mitsumori等[1]抽取了蛋白质实体附近的词特征,探索了词汇特征对抽取性能的影响。Sugiyama等[2]从包含PPI的句子中抽取了动词和名词信息,进一步研究了词汇信息尤其是动词对PPI抽取的作用。此外,Giuliano等[3]还探讨了词性等浅层语言学信息。然而,上述系统都没有考虑任何句法和依存信息,而研究表明这些信息在新闻领域的关系抽取中有很好的效果[4]。因此,Stre等[5]将句法信息、浅层依存关系信息和词汇特征结合起来进行PPI抽取,显著提高了PPI抽取的性能。但是目前对于依存信息的研究并不深入,所采用的特征也不能有效捕获依存树中的结构化信息,系统性能相对于新闻领域仍有很大差距。
当前机器学习领域的另一个热门课题就是核函数的研究和使用。基于核函数的方法直接以结构树为处理对象,再使用支持核函数的分类器进行关系抽取。然而受制于计算复杂度,该方法往往不能应用于实际的PPI抽取系统中,这也促使我们考虑在基于特征向量的方法中对依存信息做进一步探索,以期充分利用依存信息。
本文第2节介绍了基于特征向量的PPI抽取方法,分析了基准系统所用的各种特征。第3节详细描述了依存信息驱动的PPI抽取系统所用的特征。第4节给出了数据处理方案、实验流程和结果分析。最后一节是本文的结论和展望。
2 基于特征向量的PPI抽取
对于基于特征向量的信息抽取方法来说,PPI抽取可以看作是一个分类问题。首先,系统要将已标注或未标注的PPI实例构造成一个特征集合,并映射到一个n维的特征向量空间;然后,在特征向量空间上运用机器学习方法。这个过程可以分为两个阶段:在训练时,分类学习算法利用标注好的PPI实例学习得到一个分类器;测试时,利用该分类器判断待测试的关系实例所属的关系类别,以预测PPI是否存在。
以往的信息抽取研究表明,词汇信息和浅层句法信息在关系抽取中的作用非常明显。因此,我们先提取词汇、交叠特征、基本短语块以及简单的句法树信息构建一个基准系统,作为与加入依存信息的PPI抽取系统的对比。基准系统用到词汇、交叠、基本短语块和句法树四种类型的特征。
1) 词汇特征是最容易挖掘的语言学特征。本系统用到四种类型的词汇信息:a)蛋白质实体的名称;b)两个蛋白质实体之间的词;c)第一个蛋白质实体之前的词;d)第二个蛋白质实体之后的词。为避免引入噪音,我们只考虑第一个蛋白质前和第二个蛋白质后的两个词。
2) 实体的交叠特征属于结构化信息,可以反映实体之间的位置关系。AIMed语料库中存在少量相互嵌套的PPI实例,利用交叠特征可以反映蛋白质实体的嵌套信息。系统采用了以下交叠特征:
• #MB: 蛋白质实体之间的其他蛋白质实体数目。
• #WB: 蛋白质实体之间的单词数目。
• E-Flag: 判断蛋白质实体是否嵌套。
3) 相对于词汇特征,基本短语块包含粒度更大的局部信息。基本短语块特征是用Sabine Buchholz的perl脚本*http://ilk.kub.nl/~sabine/chunklink/从完全句法树中获得的,而句法树则是由Stanford Parser*http://nlp.stanford.edu/software/lex-parser.shtml生成。类似词汇特征,我们提取了以下基本短语块特征:
• CPHBNULL:实体之间没有短语块。
• CPHBFL:实体之间仅有一个短语块时,该短语块的核心词。
• CPHBF:实体之间至少两个短语块时,除首尾短语块,其余短语块中第一个短语块的核心词。
• CPHBL:实体之间至少两个短语块时,除首尾短语块,其余短语块中最后一个短语块的核心词。
• CPHBO:实体之间除首尾短语块外,其他短语块的核心词。
• CPP:连接两个实体所在短语块的短语块类型。
为防止以上基本短语块特征过于具体而造成数据稀疏问题,我们构造了一系列的组合特征。这些特征是将上述基本短语块特征(CPP除外)与它们对应的短语块类型结合起来得到的。
4) 句法树作为反映句子间语法关系的重要结构,可以揭示实体之间较长距离的语义关系,因此我们也抽取了句法树特征:
• PTP:句法树中两个实体之间的路径(经过去重处理)。
3 依存信息驱动的PPI抽取
依存树可以揭示句子中的长距离依存关系,并且能避免非结构化特征中出现的噪音,可以为关系抽取提供更为有效的信息。目前,利用依存信息进行PPI抽取的研究主要集中于基于核函数的方法,比如Airola等[6]采用全依存路径图核,Kim等[7]采用加权路径子串核分别进行了实验,并获得了不错的性能。除此之外,Stre等[5],Miyao等[8]和 Miwa等[9-10]采用复合核函数的方法将平面特征与结构化信息结合起来进行PPI抽取,大幅度提高了系统性能。虽然目前基于核函数的方法在性能方面要比基于特征向量的高,但是所有核函数方法都存在计算复杂性的瓶颈问题,而另一方面,依存信息在基于特征向量的PPI抽取中的作用还有待深入研究。所以在本节我们将抽取一系列依存特征,考察它们在PPI抽取中的表现。
依存树也是借助于Stanford Parser得到的。Stanford Parser依存分析的输出格式是:依存类别(word1,word2),其中word1是核心词,word2依赖于该核心词,依存类别则由Stanford Parser预先定义。根据这些依存关系对,我们可以构建一个句子的依存树,并且抽取它的如下特征,表示为DependenecySet1:
• DP1TR:依存树中蛋白质PROT1到根节点的路径。
• DP2TR:依存树中蛋白质PROT2到根节点的路径。
• DP12DT:依存树中两个蛋白质之间的依存关系类别。
• DP12:连接两个蛋白质路径上的词和依存类型的组合。
• DP12S:DP12中的每个单词及其依存类型的组合。
• DPFLAG:判断两个蛋白质是否具有直接依存关系。
以句子“PROT1 contains a sequence motif binds to PROT2.”为例,Stanford Parser生成的语法关系及构造的依存树如下:
虽然Erkan等[11]抽取了依存树中蛋白质实体之间的路径信息,并采用基于核函数的方法获得了较好的结果。然而这种长距离的路径信息并不适合基于特征向量的方法,原因在于路径太过具体,导致严重的数据稀疏问题,使得系统准确率较高而召回率则很低。为避免这种情况,我们将路径信息拆分成细粒度的特征,如DP12S特征。
除了上述依存特征,我们也探索了依存树中动词对PPI抽取的影响。不同于新闻领域,在生物医学领域文本的关系抽取中,动词扮演着更为重要的角色,这是因为主要动词的变化可以很容易导致PPI关系极性的改变。而以往的研究中并没有对这一问题给予足够的关注,因此本文抽取了以下动词及其在依存树中的位置特征,表示为DependencySet2:
• FVW:DP12特征中位于第一个蛋白质之前的动词。
• LVW:DP12特征中位于第二个蛋白质之后的动词。
• MVW:DP12特征中的其他动词。
• #FVW: FVW中动词的数目。
• #LVW: LVW中动词的数目。
• #MVW: MVW中动词的数目。
4 实验结果与分析
本节首先介绍实验中的语料预处理策略及评价标准,然后报告各种特征在PPI抽取中的表现并从语言学角度作出相应分析,最后与其他PPI抽取系统的性能进行比较,并在4个常用的PPI语料库上也进行了同样实验,以验证本系统的泛化性能。
4.1 实验设置
我们采用AIMed语料库作为主要实验数据集,AIMed是一种广泛应用于PPI抽取领域的语料库,它包含225篇从MEDLINE中提取的文章摘要。另外,我们也在其他四个经常使用的PPI语料库*http://mars.cs.utu.fi/PPICorpora/GraphKernel.html上进行了实验。

实验中,我们选择SVM作为分类器。SVM分类器本质上是二元分类器,所以它非常适合判断PPI是否存在的任务。在本系统中,我们使用了Joachims等开发的二元分类工具SVMLight*http://svmlight.joachims.org/。
实验设置方面,我们采用了与Giuliano等[3]完全相同的文档级十倍交叉验证策略,这样可以最大化地利用实验资源,也利于与前期相关研究的实验结果进行对比。我们采用的评价标准是关系抽取中普遍采用的准确率(P),召回率(R)和F测度(F1)。另外,AUC(area under the receiver operating characteristics curve)可以衡量数据类别在不同分布下的分类算法总体性能,已经广泛用于机器学习中对分类算法进行评价。
4.2 各种特征的表现
表1显示了在AIMed语料库上,采用10倍交叉验证策略时,各种特征对系统性能的影响。为了显示依存树中动词特征对性能的贡献度, 我们将依存特征分为两部分进行实验。

表1 不同特征对PPI抽取的影响
表1显示本系统在准确率,召回率和F测度上分别达到63.4%,48.8%和54.7,同时也表明了以下特点:
• 词汇特征获得的性能比较低,尤其是召回率只有41%左右。这表明由于词汇稀疏问题,单靠词汇特征本身不能准确表达PPI的关键信息,也说明PPI抽取的难度较大。
• 交叠特征略微降低了系统性能。统计显示,AIMed语料库中#MB和#WB各特征的正负例比例非常接近,从而造成这些特征不具有区分能力。所以在后面的实验中,本系统排除了交叠特征。
• 相对于词汇特征,基本短语块特征将召回率提高3.9%,F测度提高3个单位。可以看出基本短语块特征是获取局部句法信息的重要途径。
• 句法树特征的作用不很明显,它提高F测度仅0.8个单位,造成这种情况的原因之一可能是蛋白质实体之间的路径往往过长,导致了句法树特征的稀疏问题;另一方面,句法树特征PTP有时会部分包含在基本短语块特征CPP中,此时句法树特征就没有为系统提供新的可用信息。
• 依存特征DependencySet1十分有效,它将准确率和召回率分别提高了1.6%和2.3%,F测度也随之提高了2.5个单位。这表明依存特征可以有效捕获PPI实例,且能避免浅层句法信息中经常出现的噪音。统计数据显示AIMed语料库中蛋白质实体距离大于5个词的句子占总数的60%以上。所以,依存特征在PPI抽取中具有巨大潜力,因为它们可以抽取长距离的依存信息。以图1中的句子为例,虽然两个蛋白质在句子中相距较远,但它们之间的依存关系却简明而清晰地表达了其相互作用。
• 依存树中的动词特征提高F测度0.8个单位,这是因为一些动词如interact、active和inhibit等,能强烈暗示两个蛋白质实体的关系,为检测PPI提供了可靠的信息。
4.3 与其他系统的比较
表2是本系统与其他主要PPI抽取系统性能的对比, 其中仅列出了采用相同实验设置的系统。按照不同的机器学习方法,我们将所有的系统分为三类:基于特征向量的方法,基于核函数的方法和基于复合核函数的方法。表2显示了Airola 等,Miwa 等和Kim等采用基于核函数的方法获得了相对高的性能,但本系统获得的54.7的F值是所有基于特征向量的方法中最好的,即使与某些基于核函数的方法相比也处于先进水平。
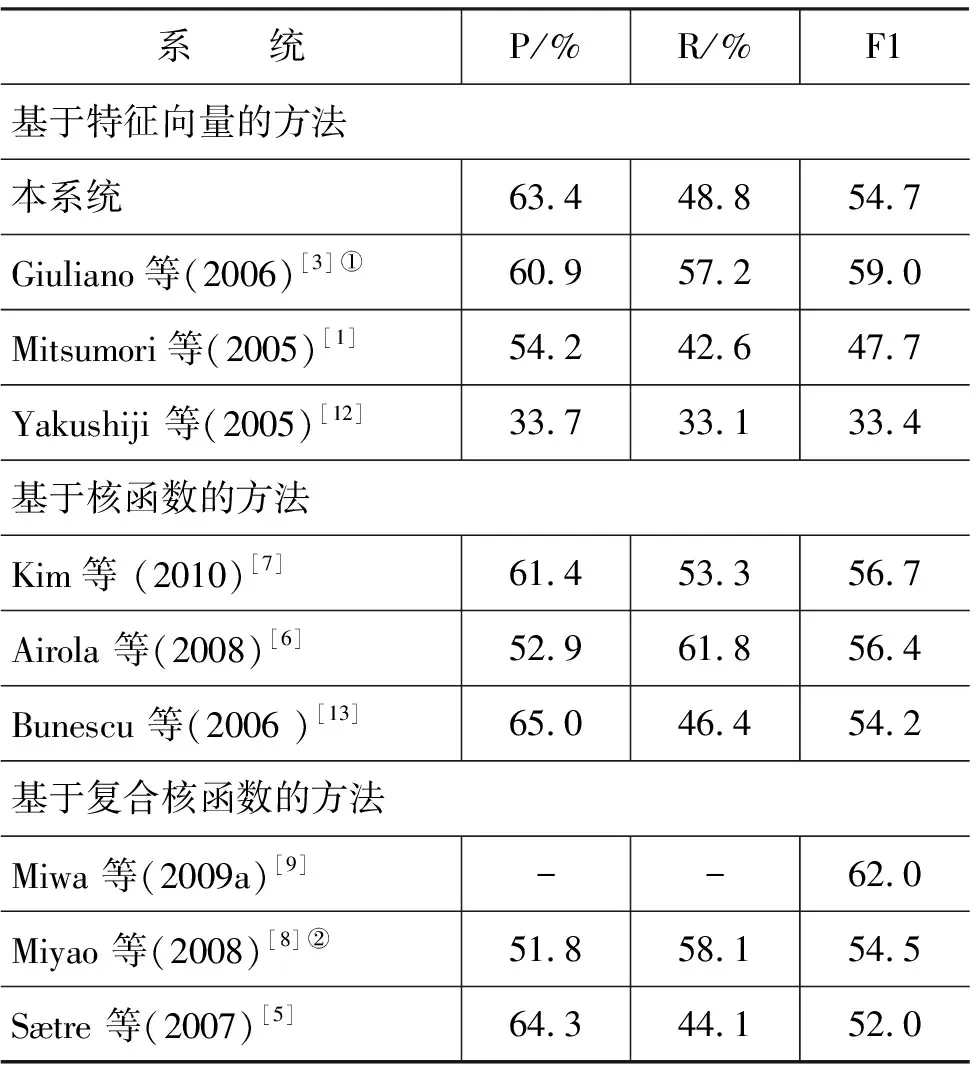
表2 与其他PPI系统的比较
为了测试本系统在生物医学语料库上的泛化性能,我们也在BioInfer、HPRD50、IEPA和LLL四个PPI语料库上用同样的方法进行了实验。表3显示了相应的F值,AUC测度及其标准差,并与Airola等[6]和Miwa等[9]的数据进行了对比。
表3显示我们的系统性能与另外两个系统的趋势基本一致,LLL语料库上均获得最好性能和最大的F值标准方差;而在AIMed的性能都是5个语料库中最差的,不过其F值标准方差并不大;BioInfer语料库的关系实例数最多,虽然F值和AUC值不高,但它们的标准方差往往较小。

表3 在其他PPI语料库上的性能
5 结论与展望
本文以SVM为分类器,用基于统计学习的方法实现了一个有指导的PPI抽取系统并且获得了F测度54.7的较好性能。本系统综合研究了各种词汇、基本短语块、句法尤其是依存特征对PPI抽取的影响。我们发现依存树特征和基本短语块特征对PPI抽取的贡献最大,并且依存树中的动词特征还能进一步提高系统性能。另外,在多个生物医学领域语料库上的实验也检验了本系统的泛化性能。
下一步工作中,我们将在基于特征向量的PPI抽取中探索更多的句法特征,同时关注结构化信息的有效表达方式及其与平面特征结合起来的途径,以期进一步提高系统性能。
[1] T. Mitsumori, M. Murata, Y. Fukuda, K. Doi, and H. Doi. 2006. Extracting protein-protein interaction information from biomedical text with SVM[J]. IEICE Transactions on Information and Systems, E89-D (8): 2464-2466.
[2] K.Sugiyama, K.Hatano, M.Yoshikawa, and S.Uemura. Extracting information on protein-protein interactions from biological literature based on machine learning approaches[J]. Journal of Genome Informatics.2003, (14): 699-700.
[3] C. Giuliano, A. Lavelli, and L. Romano. 2006. Exploiting Shallow Linguistic Information for Relation Extraction from Biomedical Literature[C]//Proceedings of EACL’06, Trento, Italy.2006:401-408.
[4] G.D. Zhou, J. Su, J. Zhang, and M. Zhang. Exploring various knowledge in relation extraction[C]//Proceedings of ACL’2005, Ann Arbor, Michgan, USA, 2005:427-434.
[6] A.Airola, S.Pyysalo, J. Björne, T.Pahikkala, F. Ginter, and T.Salakoski. All-paths graph kernel for protein-protein interaction extraction with evaluation of cross corpus learning[J]. BMC Bioinformatics.2008,9(suppl 11):s2.
[7] S. Kim, J.Yoon, J.Yang and S.Park. Walk-weighted subsequence kernels for protein-protein interaction extraction[J]. Journal of BMC Bioinformatics, 2010, 11:107-128.
[8] Y.Miyao, R.Stre, K. Sagae, T.Matsuzaki, and J.Tsujii. Task-oriented evaluation of syntactic parsers and their representations[C]//Proceedings of ACL-08: HLT, 1008: 46-54.
[9] M. Miwa, R. Stre, Y. Miyao and J. Tsujii. 2009a. Protein-Protein Interaction Extraction by Leveraging Multiple Kernels and Parsers[J]. Journal of Medical Informatics, 2009,78: e39-e46.
[10] M.Miwa, R.Stre, Y.Miyao and J.Tsujii. A Rich Feature Vector for Protein-Protein Interaction Extraction from Multiple Corpora[C]//Proceedings of EMNLP’09, August, Singapore.2009:121-130.
[11] G. Erkan, A. Ozgur, D.R. Radev. Semi-Supervised Classification for Extracting Protein Interaction Sentences using Dependency Parsing[C]//Proceedings of EMNLP-CoNLL’2007: 228-237.
[12] A.Yakushiji, M.Yusuke, T.Ohta, Y.Tateishi, J.Tsujii. Automatic construction of predicate-argument structure patterns for biomedical information extraction[C]//Proceedings of EMNLP’06, Sydney, Australia 2006: 284-292.
[13] R. Bunescu and R. Mooney. 2005. Subsequence kernels for relation extraction[C]//Proceedings of NIPS’05, December, 2005:171-178.
