基于禁忌遗传算法的RNA二级结构预测
2011-04-26刘勇国
刘勇国,朱 婵,晏 华
(1. 电子科技大学计算机科学与工程学院 成都 611731; 2. 苏州大学江苏省计算机信息处理技术重点实验室 江苏 苏州 215006;3. 四川建筑职业技术学院图书馆 四川 德阳 618000)
生物体根据脱氧核糖核酸(deoxy ribonucleic acid,DNA)的遗传信息合成蛋白质并完成各项生命活动过程,如生物信号的识别和传递,营养物质的运输等。在该过程中,蛋白质合成的直接模板是核糖核酸(ribonucleic acid,RNA)而非DNA。随着对RNA分子结构探索的不断深入,研究发现某些简单生物体如烟草花叶病毒的RNA已成为遗传信息的直接载体,负责存储遗传信息和蛋白质合成。目前,研究RNA分子结构成为认知生物遗传信息和蛋白质合成的重要途径。RNA分子由一级、二级和三级结构[1]组成。一级结构为多核苷酸链的核苷酸序列;二级结构为多核苷酸链回折形成的配对双螺旋区和不对称区域;三级结构为二级结构单元相互作用形成的三维空间结构,确定二级结构单元的空间定位取向和蛋白质合成。RNA一级结构预测起步最早,方法较完善,主要通过生物化学方法直接预测。二级结构预测主要采用物理化学和生物信息学方法。三级结构预测已被证明为NP-hard问题[2]。由于二级结构介于一级结构和三级结构之间,存储了较多高级结构信息,因此研究RNA二级结构成为预测RNA分子结构的重要途径。
RNA二级结构预测包括基于物理化学的方法和基于生物信息学的方法[3]两类。物理化学的方法通过X射线结晶衍射和核磁共振确定RNA分子结构,虽然测量精度高,但对软硬件要求较高,而且RNA分子降解较快,难以结晶,造成预测困难,所以,该类方法适用于碱基数量小于100的RNA二级结构预测问题。生物信息学方法包含系统发育比较技术和自由能最小技术[2]两类预测技术。系统发育比较技术对比待测序列与已知同源序列,通过同源序列二级结构的形成方式预测待测序列的二级结构[1]。自由能最小技术源于热动力学的能量耗散原理,通过设置RNA二级结构中茎环结构的热动力学参数,模拟实际RNA分子热运动,建立二级结构的能量模型,计算RNA序列折叠后的自由能,确定具备最小自由能的二级结构为预测结果,实现对RNA二级结构的预测[2],预测方法包括动态规划算法[4]、退火模拟算法[5]、遗传算法[6]、上下文无关文法预测算法[7]、协变信息预测算法[3]等。由于遗传算法具有隐含并行性、自适应性等特点,已应用于RNA二级结构预测问题。研究发现,遗传算法的种群进化过程缺乏记忆性,搜索过程易出现种群早熟现象,导致预测精度降低[4,8-9]。文献[10]设计并行遗传算法模拟RNA序列形成二级结构时的折叠情况,推测RNA序列的二级结构。文献[11]采用十进制编码设计遗传算法预测RNA二级结构,以改善预测效率和精度[8,11-12]。文献[5]组合模拟退火与遗传算法,以增强预测算法的局部搜索能力,避免种群早熟现象。文献[13]基于混沌差分进化算法预测RNA二级结构,利用混沌映射的伪随机性和遍历性提高算法的全局搜索能力。文献[14]提出基于免疫粒子群集成的RNA二级结构预测方法,通过并行群落优化方式实现协同演化。
本文提出基于禁忌遗传算法的RNA二级结构预测算法TGARNA。TGARNA算法给出茎区相容性检测过程,保留最长茎区构造茎区相容个体;将禁忌思想引入遗传操作,记录个体家族特征和进化过程,防止近亲繁殖,保持种群多样性。仿真实验表明,TGARNA算法能够获取最小自由能并预测RNA二级结构。
1 RNA二级结构
除少数病毒RNA为双链结构外,RNA分子通常为多核苷酸单链结构,包含腺嘌呤(adenine,A)、鸟嘌呤(guanine,G)、胞嘧啶(cytosine,C)和尿嘧啶(uralic,U)4类含氮碱基。碱基与戊糖结合形成核苷,戊糖侧链与磷酸分子结合形成核苷酸,核苷酸分子以3’和5’磷酸二酯键连接形成核苷酸序列[1]。RNA单链自身回折呈现碱基配对和单链交替出现的茎环结构,形成RNA二级结构,如图1所示。

图1 RNA二级结构示意图
RNA二级结构中,碱基以A-U、G-C或G-U互补配对形成的连续双螺旋区域称为茎区,不构成互补配对的单链结构称为环区,环区类型包括发夹环、凸环、内环和内分支环等。茎区由氢键相连的配对碱基叠加形成,氢键数量越多碱基对自由能越小,越有利于增强二级结构稳定性。环区数量增加导致自由能增长,造成二级结构稳定性下降[2]。RNA二级结构数目随序列长度增加呈指数级增长,因此有效预测RNA二级结构成为RNA分子结构研究急待解决的问题[2]。碱基是RNA二级结构的基本单元,连续配对碱基集形成茎区,未配对碱基构成环区,相容茎区集唯一确定RNA二级结构[2]。RNA二级结构的相关描述如下[5]。

2 TGARNA算法
TGARNA算法过程基于遗传算法,通过相容性检测构造合法种群,引入禁忌思想防止近亲繁殖,提高种群多样性[15],算法流程如图2所示。

图2 TGARNA算法流程
2.1 种群初始化


图3 个体编码方式
2.2 相容性检测
由于不相容茎区组合将生成非法RNA二级结构,本文设计相容性检测环节建立合法个体。常用相容性检测方法根据茎区排序位置确定茎区是否保留,未考虑其能量值,易造成低能量茎区漏选[5]。鉴于RNA二级结构中茎区数目越多,茎区长度越长,自由能越小,故应尽量保留长相容茎区。本文相容性检测步骤如下:


通过相容性检测避免非法个体,保留低能量茎区,改善种群性能。
2.3 适应度计算
本文采用文献[16]提出的最小自由能模型评价RNA二级结构稳定度,预测RNA二级结构。RNA二级结构自由能计算为:

2.4 选择操作
本文采用比例选择方式,个体适应度越大,其选择概率越大。个体Xi的选择概率为:

2.5 交叉操作
交叉操作采用融合禁忌思想的单点交叉方式,针对个体Xi的茎区组合Xsi进行。TGARNA算法将家族特征和禁忌思想引入交叉操作,防止近亲个体繁殖后代,保持种群多样性;同时,通过期望准则允许禁忌个体产生高适应度后代,增加选择压力,提高算法收敛速度。交叉操作步骤如下。


2.6 变异操作
本文采用动态变异方式产生后代个体,针对茎区组合更新变异后个体的家族特征和禁忌表。变异操作步骤如下。

2.7 TGARNA算法的实现
TGARNA算法步骤如下。
1) 给定RNA序列,建立茎区池并生成初始种群,设g=1;
2) 执行种群相容性检测;
3) 执行选择操作;
4) 执行交叉操作;
5) 执行变异操作;
6) 将父代种群中Nd个最优个体更新后代种群中适应度最低的Nd个个体;

3 实验结果
仿真实验平台采用Windows XP SP3,Matlab7.1语言,Intel Core 2 Duo 2.20 GHz CPU,2G物理内存,仿真算法每次实验运行20次。首先讨论算法参数的选择过程,然后分析TGARNA算法的预测结果。
3.1 算法参数选择


图4 不同代沟值Gg的算法进化结果

表1 不同代沟值Gg的结果比较


图5 不同交叉概率pc的算法进化结果

表2 不同交叉概率pc的结果比较

图6 不同禁忌表长度因子α的算法进化结果
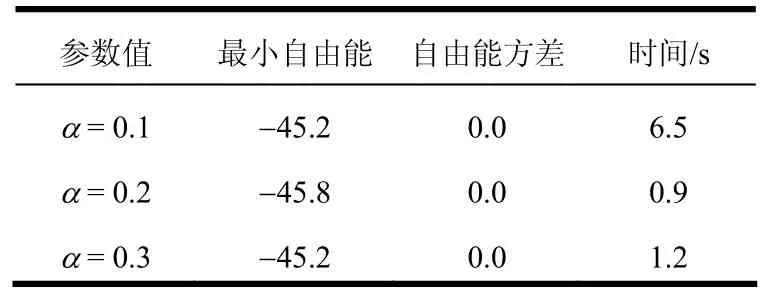
表3 不同禁忌表长度因子α的结果比较
3.2 实验结果讨论
比较基于遗传算法的RNA二级结构预测方法(genetic algorithm based prediction of rna secondary structure,GARNA)、融入相容性检测的基于遗传算法的RNA二级结构预测方法GARNA*和TGARNA算法,GARNA算法采用比例选择、单点交叉和单点变异实现遗传操作;GARNA*算法在GARNA算法的基础上改进茎区相容性检测;TGARNA算法在GARNA*的基础上融入禁忌思想。算法实验种群规模均为100。考虑到RNA序列长度差异,针对Giardia virus和Y1 scRNA序列,进化代数设为100;针对U17 snoRNA和PSTVd RNA序列,进化代数设为1 000。通过调整进化代数适应不同长度序列RNA分子的二级结构预测。
仿真实验采用Giardia virus、Y1 scRNA、U17 snoRNA和PSTVd RNA共4个RNA测试序列。Giardia virus序列源于贾第虫病毒体,由77个碱基构成,包含4个茎区、2个发夹环和1个多分支环。Y1 scRNA序列源于小家鼠的线形染色体组RNA,由111个碱基构成,包含2个茎区、1个发夹环和1个内环[17]。U17 snoRNA序列源于沼泽侧颈龟的转录RNA,由240个碱基构成,包含9个茎区、3个发夹环、3个凸环、5个内环和1个多分支环。PSTVd RNA序列源于马铃薯纺锤块茎类病毒体的线形染色体组RNA,由359个碱基构成,包含25个茎区、1个发卡环和23个内环[17]。
Giardia virus RNA二级结构自由能的计算过程如图7所示,该序列二级结构的预测结果如表4所示。预测指标包括平均最小自由能、最小自由能均方差、算法首次获得最小自由能的平均运行时间和正确预测茎区数与实际二级结构中的茎区数比。可见,GARNA算法的自由能下降缓慢,GARNA*与TGARNA算法的自由能下降迅速。GARNA*算法的运行时间下降到 0.6 s时,预测精度提高到56.52%,TGARNA算法的预测精度进一步提高到60.12%,正确预测3个茎区、1个发夹环和1个多分支环。

图7 Giardia virus序列二级结构的自由能计算
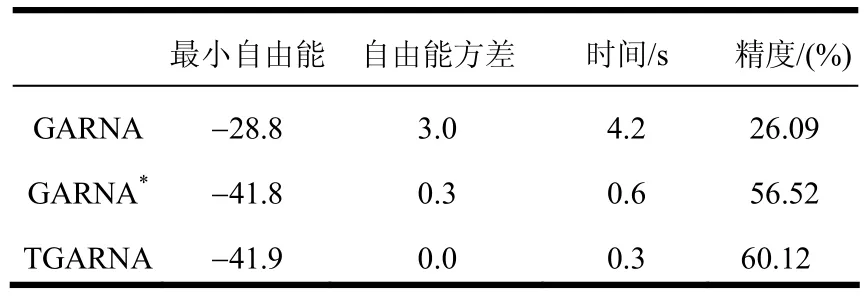
表4 Giardia virus序列二级结构的预测结果
Y1 scRNA二级结构自由能计算如图8所示。该序列二级结构的预测结果如表5所示。GARNA算法收敛速度较慢,GARNA*和TGARNA算法能较快获得最小自由能。GARNA*算法的运行时间下降到2.1 s时,预测精度达到100%,正确预测Y1 scRNA序列的二级结构。TGARNA算法的运行时间进一步下降到0.9 s。

图8 Y1 scRNA的二级结构的自由能计算

表5 Y1 scRNA序列二级结构的预测结果
U17 snoRNA二级结构自由能的计算过程如图9所示。该序列二级结构的预测结果如表6所示。GARNA*算法性能改善明显,获取最小自由能的速度较TGARNA算法快,GARNA算法预测效率最低。TGARNA算法较GARNA*算法的预测精度高,正确预测5个茎区、2个发夹环和1个凸环。

图9 U17 snoRNA序列二级结构的自由能计算

表6 U17 snoRNA二级结构的预测结果
PSTVd RNA二级结构自由能计算如图10所示。该序列二级结构的预测结果如表7所示。GARNA算法性能最差,TGARNA算法较GARNA*算法的最小自由能下降。TGARNA算法预测效率最高并达到最高预测精度,正确预测21个茎区、1个发夹环和16个内环。

图10 PSTVd RNA的二级结构自由能计算
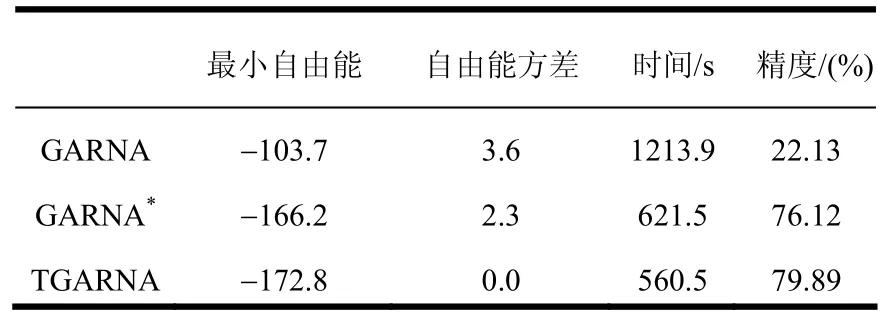
表7 PSTVd RNA二级结构的预测结果
实验表明,改进茎区相容性检测能够改善种群性能,引入禁忌思想能够防止近亲个体繁殖,预测算法的收敛速度和预测效率得到改善。
4 结 论
RNA分子结构是生物信息学领域的重要研究对象,通过RNA结构研究病毒的遗传信息,探索RNA参与合成蛋白质及转录遗传信息的过程。本文给出基于禁忌遗传算法的RNA二级结构预测方法TGARNA,通过设计茎区相容性检测,保留长茎区以降低个体自由能,将家族特征和禁忌思想融入个体编码,防止近亲繁殖,保持种群多样性。仿真实验表明,除U17 snoRNA序列外,TGARNA算法能够实现较高的预测效率和精度。
由于RNA二级结构预测的最小自由能模型忽略了某些结构(如假结),造成预测算法对U17 snoRNA序列的预测精度较低。假结结构形成复杂,文献[1]证明使用最小自由能模型预测包含任意假结的RNA二级结构为NP完全问题[1]。后续研究工作中,将考虑基于TGARNA算法将个体设计为由单位结构组成,利用TGARNA算法计算相容单元结构集合形成初始RNA二级结构,通过环区配对确定被测序列中的假结结构,预测含假结的RNA二级结构。
[1] PAR S K, BANDYOPADHYAY S, RAY S S. Evolutionary computation in bioinformatics: a review[J]. IEEE Transactions on Systems, Man, and Cybernetics, Part C:Applications and Reviews, 2006, 36(5): 601-615.
[2] COHEN J. Bioinformatics - an introduction for computer scientists[J]. ACM Computer Surveys, 2004, 36(2):122-158.
[3] 邹权, 郭茂祖, 张涛涛. RNA二级结构预测方法综述[J].电子学报, 2008, 36(2): 331-337.ZOU Quan, GUO Mao-zu, ZHANG Tao-tao. A review of RNA secondary structure prediction algorithms[J]. Acta Electronica Sinica, 2008, 36(2): 331-337.
[4] RIVAS E, EDDY S R. A dynamic programming algorithm for RNA structure prediction including pseudoknots[J].Journal of Molecular Biology, 1999, 285(5): 2053-2068.
[5] 任清华, 莫忠息, 陶玉敏. 预测RNA二级结构的一种遗传模拟退火算法[J]. 武汉大学学报(理学版), 2004, 50(1):23-28.REN Qing-hua, MO Zhong-xi, TAO Yu-min. A geneticsimulated-annealing algorithm for predicting RNA secondary structure[J]. Journal of Wuhan University(Natural Science Edition), 2004, 50(1): 23-28.
[6] VAN BATENBURG F H D, GULTYAEV A P, PLEIJ C W A.An APL programmed genetic algorithm for the prediction of RNA secondary structure[J]. Journal of Theoretical Biology,1995, 174(3): 269-280.
[7] 唐四薪, 刘艳波, 尹军. 文法推断RNA二级结构的研究进展[J]. 生物信息学, 2008, 6(4): 190-192.TANG Si-xin, LIU Yan-bo, YIN Jun. Research advances of grammatical inference of RNA secondary structure[J]. China Journal of Bioinformatics, 2008, 6(4): 190-192.
[8] WIESE K C, DESCHENES A A, HENDRIKS A G.RnaPredict—an evolutionary algorithm for RNA secondary structure prediction[J]. IEEE/ACM Transactions on Computational Biology and Bioinformatics, 2008, 5(1):25-41.
[9] LIU Y G, CHEN K F, LIAO X F, ZHANG W. A genetic clustering method for intrusion detection[J]. Pattern Recognition, 2004, 37(5): 927-942.
[10] SHAPIRO B A, WU J C, BENGALI D, POTTS M J. The massively parallel genetic algorithm for RNA folding MIMD implementation and population variation[J].Bioinformatics, 2001, 17(2): 137-148.
[11] WIESE K C, GLEN E. A permutation-based genetic algorithm for the RNA folding problem: a critical look at selection strategies, crossover operators and representation issues[J]. Biosystems, 2003, 72(1-2): 29-41.
[12] WIESE K C, GLEN E. A permutation based genetic algorithm for RNA secondary structure prediction[C]//In:Proceedings of the 2nd International Conference on Hybrid Intelligent Systems. Chile: [s.n.], 2002, 173-182.
[13] 胡桂武, 彭宏. 利用混沌差分进化算法预测RNA二级结构[J]. 计算机科学, 2007, 34(9): 163-166.HU Gui-wu, PENG Hong. An algorithm-base chaos differential evolution for predicting RNA secondary structure[J]. Computer Science, 2007, 34(9): 163-166.
[14] 胡桂武, 彭宏. 基于免疫粒子群集成的RNA二级结构预测算法[J]. 计算机工程与应用, 2007, 43(3): 26-29.HU Gui-wu, PENG Hong. Algorithm based on immune PSO ensemble for predicting RNA secondary structure[J].Computer Engineering and Applications, 2007, 43(3):26-29.
[15] TING K C, LI H T, LEE H N. On the harmonious mating strategy throug tabu search[J]. Information Sciences, 2003,156(3-4): 189-214.
[16] MATHEWS D H, SABINA J, ZUKER M, TURNER D H.Expanded sequence dependence of thermodynamic parameters improves prediction of RNA secondary structure[J]. Journal of Molecular Biology, 1999, 288(5):911-940.
