大黄鱼野生群体与养殖群体遗传多样性研究
2011-03-15陈淑吟徐士霞张志勇郭忠仁孙中响朱立静
陈淑吟,徐士霞,张志勇,郭忠仁,孙中响,3,朱立静,3
(1.江苏省海洋水产研究所,江苏 南通226007; 2.南京师范大学 生命科学院 动物遗传资源研究所,江苏 南京,210097; 3.苏州大学 医学部基础医学与生物科学学院,江苏 苏州 215123)
大黄鱼(Pseudosciaena croceaRichardson 1846)隶属鲈形目(Perciformes)、石首鱼科(Sciaenidae)、黄鱼属(Pseudosciaena),主要分布于中国黄海南部、东海和南海,为暖温性近海集群洄游鱼类,一般体长20~40 cm,最大可达75 cm,曾是中国传统的“四大海鱼”之一。20世纪 70年代以前,大黄鱼具有明显的渔场和渔汛期[1]。由于酷渔滥捕和环境污染,大黄鱼自然资源几近枯竭,到80年代以后形不成渔汛。渔业部门虽然制订了许多资源保护措施,但至今大黄鱼仍属东海区资源严重衰退的种类[2]。20世纪80年代末刘家富等[3]研究成功人工繁育大黄鱼,现在大黄鱼人工养殖产业化规模在福建、浙江等地不断壮大,但伴随着产业发展又面临着大黄鱼品质下降、病害频发的局面,进行科学管理对保护和恢复大黄鱼资源相当必要[4]。近几年来,开展的人工放流大黄鱼对恢复几近枯竭的自然资源具有积极意义,而对现有遗传资源的了解有助于进一步保护该资源的遗传多样性。
中国黄海南部的吕泗渔场为大黄鱼主要自然分布区之一,目前大黄鱼自然资源也十分稀少,有关该海区大黄鱼的种质状况及其与其他群体的遗传差异未有报道; 而且,该海区是目前尚未开发人工养殖的大黄鱼主要分布海域。线粒体COI基因序列作为功能稳定又具较多变异的序列,作为分子标记已被应用于许多水生生物种群遗传学研究中,但目前尚未见用于大黄鱼的群体遗传学研究。作者利用线粒体COI基因序列片段与ISSR标记方法研究大黄鱼江苏吕泗渔场自然群体的遗传多样性,并比较其与浙江、福建主产区两个养殖群体间遗传差异,为大黄鱼资源保护与利用提供更多依据。
1 材料与方法
1.1 样本来源与总基因组DNA提取
大黄鱼 3个群体样本取样时间在 2009年 9~10月。江苏野生群体样本(JS,30个)于吕泗渔场海区为3次捕捞得到,体长22.2~24.8 cm; 两个养殖群体样本分别来自浙江象山港(ZJ,30个)和福建宁德(FJ,30个)养殖海区,体长分别为 8.2~9.2 cm、20.3~21.6 cm。
基因组总 DNA提取采用 DNA提取试剂盒(Takara,DV811A),按试剂盒说明书要求操作,最后洗脱定溶于TE(100 mg/L)溶液中,4 ℃保存备用。
1.2 COI引物设计与PCR扩增
根据鱼类COI通用引物[5]以及从NCBI数据库下载大黄鱼的COI序列,设计扩增COI基因序列引物,序列为:COI-F:5’ -TCT ACT AAT CAC AAA GAC ATC GGC AC-3’ /COI-R:5’-GAC CTC AGG ATG GCC GAA GAA TCA-3’。引物由上海生工生物工程技术服务有限公司合成。
PCR反应总体积为50 μL,含有:DNA模板约50 ng,浓度为10 pmol引物各1 μL,25 μLPremix ExTaq聚合酶混合液(Takara,Code:D332A)。PCR扩增参数为:94℃预处理 5 min; 进行 35 循环:94℃ 30 s,55℃30 s,72℃ 1 min; 最后72℃延伸10 min。
PCR产物在1.5%琼脂糖凝胶中电泳,溴化乙锭(Ethidium bromide,EB)染色,凝胶成像系统检测。产物经试剂盒(BBI,Code:BS364)切胶纯化后送上海Sangon用PCR反应引物双向测序。测序仪器为ABI PRISM 3730,测序试剂为BigDye terminator v3.1。
1.3 COI序列数据分析
所得DNA序列在NCBI 数据库中进行BLAST同源检测确认; 利用 ClustalX1.81[6]软件进行序列比对及波形图谱校正。用MEGA4.0软件[7]统计序列的核苷酸组成、多态性位点及构建基于 Kimura’s 2-parameter模型 (Kimura,1980)的邻接法(Neighborjoining,NJ)与非加权分组平均法(Unweighted pair-group method with arithmetic mean,UPGMA)系统树(bootstrap重复1000 次)。用DNAsp 4.0软件[8]计算单倍型数量(Number of haplotypes,Nh)、单倍型多态性(Haplotype diversity,简称Hd)、平均核苷酸差异(Average number of differences,简称K)、核苷酸多样性(Nucleotide diversity,简称Pi),应用 Nei(1973)基因多样度法计算遗传分化系数(Gst)及基因流(Nm)等。
1.4 ISSR引物筛选与PCR扩增
引物为加拿大哥伦比亚大学UBC公司公布的第9套 ISSR引物序列,由上海生工合成。每个群体各选取3个DNA模板在15 μL的反应体系中进行扩增筛选,从初选的48个引物中进一步筛选10个扩增条带清晰、重复性好的引物(表2)进行分析。
PCR 反应总体积为 25 μL,引物 0.5 mmol/L,DNA 模板约 20 ng,12.5 μLPremix ExTaq 聚合酶混合液(Takara)。PCR扩增参数为:94 ℃预变性5 min后进入36个循环:94℃ 45 s,52℃ 45 s,72℃ 2 min;最后72℃延伸10 min。
1.5 ISSR扩增的电泳检测及数据分析
PCR 扩增产物在 minicell EC350 电泳仪上进行检测,电泳缓冲液为 0.5×TBE,琼脂糖凝胶浓度为 1.5%(含 0.5 μg/mL EB),电压不超过 5 V/cm,电泳2~3 h后取出,于 BIO- RAD 紫外成像仪下拍照记录。
按照电泳图谱中同一位置上DNA带的有无进行统计,有带记为“1”,无带记为“0”,记录清晰、稳定且长度在200~2000 bp范围内的扩增带,将图谱转化为 0/1矩阵。应用 Popgene1.31[9]软件在假定种群处于 Hardy-Weinberg平衡状态下,应用 Nei (1987)分析群体遗传参数方法,分别计算多态位点百分率(PPL)、群体总基因多样性(Ht)、群体内基因多样性(Hs)、群体间的遗传分化系数(Gst),并根据Nei’s遗传距离,对群体进行UPGMA树聚类分析。

以Gst估算基因流:Nm=0.5(1 -Gst)/Gst[10]
2 结果
2.1 不同群体 COI 基因片段扩增结果与序列分析
大黄鱼mt DNA基因COI片段的PCR扩增产物电泳检测结果为单一条带,长度约 700bp,空白对照没有出现扩增产物。序列经比对后得到片段长度为625 bp,经 NCBI数据库相似性检测确认为大黄鱼COI基因序列。测序成功65个序列,分别为:JS群体28个、ZJ群体19及FJ群体18个。
序列共检测到18个多态位点,其中11个为简约信息位点,无插入或缺失位点,转换与颠换比值为4.089,序列的碱基组成T、C、A及G的平均含量分别为27.7%、30.7%、22.4%及19.2%,A+T含量(50.1%)与G+C相当。共有15个单倍型,单倍型多态性(Hd)为0.714,不同单倍型多态位点分布见图1。JS野生群体28个样本中有9个群体独享单倍型,占群体14个单倍型的64.28%,这些单倍型没有共同变异位点;ZJ群体独享单倍型1个(h5),占群体单倍型的9.09%;FJ群体没有独享单倍型; 3群体间共享 3个单倍型(h1、h4及h11),其中单倍型h1拥有最多个体数,分别为来自JS群体8个、ZJ群体10个及FJ群体的16个样本; 另外,有 2个单倍型(h2及 h13)为 ZJ与 JS两群体共享。从表1几项指标中表明JS群体具有最高的遗传多样性,FJ群体的遗传多样性明显偏低。序列单倍型GenBank的登录号为:FJ851334- FJ851355和 GQ864238-GQ864250。

表1 基于COI部分序列的大黄鱼3个群体的遗传多样性Tab.1 Genetic diversity of the three populations of P.crocea based on mtDNA COI sequences

图1 基于 COI部分序列的不同单倍型的变异位点分布(“.”代表相同位点)Fig.1 Different sites among 15 haplotypes of mtDNA COI sequences (The “.” reprents the same site)
群体间的遗传分化估算值(Genetic differentiation estimates,P-value)为 0.9854(P>0.05,nt significant),且群体间的遗传分化指数(Fst)小于0(-0.00333),表明群体间没有明显遗传分化; 另外,依据Nei(1973)计算得出群体间Gst=0.05318,基因流为NmCOI=4.45。从两两群体间Gst反映出3个群体间的亲缘关系,JS与FJ、FJ与ZJ及ZJ与JS群体的Gst分别为 0.02115、-0.0125及-0.0196,即 JS与 FJ群体的亲缘关系最远,ZJ与JS群体的亲缘关系最近。由15个单倍型构建NJ与UPGMA系统树中拓朴结构相似,3个群体的多数单倍型并列在一起,见图2。
2.2 ISSR标记的引物筛选与扩增结果
从100个引物中最终筛选出10个扩增效果较好的引物,见表 2,其中有 4个引物为双碱基重复类型。这些引物扩增所得条带数多在 8~18,分子量在200~2000 bp范围内。3个群体大部分引物扩增的多态性差别较大。有 2个引物扩增得出电泳图谱在不同群体的条带分布明显不同,如引物 873扩增出 FJ群体在200~300 bp处及400~500 bp处各有3条及1条不同于其他两群体的特异性条带(图3); 在图4中,引物895扩增图谱在500~600 bp处有2条带的可以确定样品来自ZJ群体。利用引物扩增上存在的这些差异条带,可以进行不同种群样本的鉴定。

图2 大黄鱼3个群体COI单倍型的NJ与UPGMA法聚类树(节点上数字为大于50%置信度)Fig.2 NJ and UPGMA phylogenetic tree based on mt COI gene sequences across 15 haplogypes of P.crocea.(Numbers represent bootstrap percentages that above 50%)
通过 Nei’s(1987)分子进化分析,群体内的基因多样性(Hs)为 0.2494,群体总的基因多样性(Ht)为0.3131,群体内的基因多样性所占比率(Hs/Ht)为0.7965,即群体的变异大部分来自群内,群间遗传差异不大。对3个群体进行UPGMA聚类分析表明JS群体与ZJ群体系统亲缘关系较近(表3,图5)。
3 讨论
3.1 大黄鱼群体遗传多样性与种质保护
DNA序列的单倍型多样性(Hd)是衡量一个群体遗传变异程度的重要指标,Hd高表明群体具有较高的遗传多样性。本研究表明,江苏野生群体具有丰富得多的遗传多样性(Hd=0.9150),而浙江养殖群体又比福建养殖群体具有较高的遗传多样性水平,HdZJ=0.6830>HdFJ=0.3235; 人工养殖群体由于亲本个体数少及近亲杂交等因素,不可避免地丧失某些特定的等位基因,造成其遗传变异度及遗传多样性水平均低于野生种群的状况[11-13]。养殖群体原有亲本的进化潜力也对其后代遗传多样性高低有直接影响,本研究得出浙江象山养殖群体表现有较高于福建宁德养殖群体的遗传多样性,这可能与福建人工养殖亲本更早人工驯化繁育、遗传进化潜力水平偏低相关。陈锦等[14]利用同工酶技术分析得出湄洲的野生群体遗传多样性较宁德的野生群体高,而宁德野生和养殖群体及连江养殖群体遗传多样性水平几乎一致; 这可能由于人工放流和养殖逃逸的影响,致使野生群体和养殖群体间存在很大的基因交流,而当地海区野生数量稀少也加剧了这种影响。由于自然资源的稀少,除了加强对捕捞的管理,开展大黄鱼人工放流是增加、恢复其资源量的有效方法。

表2 大黄鱼ISSR引物及群体多态性Tab.2 ISSR primers sequences and the diversity of P.crocea

图3 引物873对3个群体的扩增电泳Fig.3 PCR resulte of primer 873 in three populations

图4 引物895对3个群体的扩增电泳Fig.4 PCR resulte of primer 895 in three populations

表3 大黄鱼不同群体的遗传一致度(上)及遗传距离(下)Tab.3 Nei's genetic identity (upper diagonal) and genetic distance (lower diagonal) among populations of P.crocea
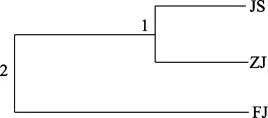
图5 基于ISSR的大黄鱼3个群体UPGMA聚类Fig.5 UPGMA dendrogram for three populations of P.crocea based on ISSR analysis
大黄鱼的集群洄游生活习性使其具有较广的扩散和分布范围。田明诚等[15]曾依据形态特征和地理分布把中国沿海的大黄鱼分为主要 3个地理群体:岱衢群(分布于黄海南部至东海中部)、闽-粤东群(分布于东海南部、台湾海峡和南海北部)及粤西群(分布于珠江口以西至琼州海峡的南海区)。目前这些海区的大黄鱼自然资源已非常罕见,瓶颈效应和基因渐渗使得该物种的群体遗传多样性越来越低。虽然,人工养殖后代会丢失一些亲本遗传信息,本研究中野生群体与养殖群体具有很高的遗传一致度,该结果与田明诚等[15]有关不同海区大黄鱼地理群体区分相一致。依据mtCOI基因序列的得出3个群体间的亲缘关系由远及近为:FJ与JS>FJ与ZJ>ZJ与JS群体。由ISSR的系统聚类树也得到与群体分布的地理距离相一致的结果。两种标记研究结果均表明群体间基因流均大于 1(NmCOI为 7.02,NmISSR为1.9563),Wright[16]认为群体间Nm>1能发挥均质化作用,从而有效抑制由遗传漂变引起的遗传分化。
3.2 不同标记方法的大黄鱼群体遗传学研究
分子遗传标记在鱼类种质资源保护与应用研究领域正发挥着越来越重要作用。RAPD、AFLP及SSR标记均被用于分析、揭示大黄鱼群体遗传多样性[12,17-18]。作者利用mtCOI基因序列揭示的大黄鱼这 3个群体的遗传特性,与利用电泳图谱分析的ISSR技术得到的结果基本一致。mtCOI基因序列与ISSR这两种标记在本文研究结果中的相同点主要为:(1)群体间的Nm皆大于 1,表明群体间存在明显的基因交流,群体间的地理距离对基因交流有一定影响;(2)JS群体具最丰富遗传多样性,且与ZJ群体亲缘关系更近,系统关系皆与地理分布距离相一致。两标记方法又有各自的不同点:(1)ISSR是以条带的扩增情况来反映结果,不但DNA模板质量及条带统计对结果估算有一定影响,而且需要较多的引物扩增、得出较多的统计数据才能更为真实的反映群体间的差异状况; 但方法简单,用于种群鉴定上有其优势,作者利用2个ISSR引物便可以简单把不同群体样品区分出来; (2)COI基因序列标记直接反映种群的DNA遗传信息,可以准确得出各群体的遗传多样性水平、群体间的系统分化、遗传差异程度,并已作为动物DNA条形码应用于物种鉴定[19],结果比 ISSR的更清晰; 但9个JS群体单倍型没有具有可以作为区分群体的特异位点。利用COI基因序列及ISSR两种标记比用单一方法反映出更为确切的群体之间的遗传进化关系。作者的这些研究结果为大黄鱼的种质保护与利用研究提供了进一步的依据。
[1]胡银茂.东海海区大黄鱼种质资源的历史演变和现状分析[J].绍兴文理学院学报,2006,26(7):49-53.
[2]徐开达,刘子藩.东海区大黄鱼渔业资源及资源衰退原因分析[J].大连水产学院学报,2007,22(5):392-396.
[3]张祖兴,李明云.大黄鱼种质资源研究进展[J].水产科学,2006,25 (7):376-378.
[4]王军,全成干,苏永全,等.官井洋野生与养殖大黄鱼同工酶的研究[J].海洋科学,2001,25(6):39-41.
[5]王中铎,郭昱嵩,陈荣玲,等.南海常见硬骨鱼类COI条码序列[J].海洋与湖沼,2009,40(5):608-614.
[6]Thompson J D,Gibson T J,Plewniak F,et al.The Clustal-X windows interface:flexible strategies for multiple sequence alignment aided by quality analysis tools[J].Nucleic Acids Research,1997,25:4876-4882.
[7]Tamura K,Dudley J,Nei M,et al.Molecular evolutionary genetics analysis (MEGA) software version 4[J].Molecular Biology and Evolution,2007,24(8):1596-1599.
[8]Rozas J,Sánchez-DelBarrio J C,Messeguer X,et al.DnaSP,DNA polymorphism analyses by the coalescent and other methods[J].Bioinformatics,2003,19:2496-2497.
[9]Yeh F C,Yang R C.POPGENE Version 1.31,Microsoft window- based freeware for population genetic analysis[CP/DK].Edmonton,Canada:University of Alberta and Tim Boyle,Centre for International Forestry Research,1999.
[10]McDermott J M,McDonald B A.Gene flow in plant pathosystems[J].Annual Review of Phytopathology,1993,31:353-373.
[11]李明云,张海琪,钟爱华,等.象山港养殖大黄鱼同工酶的分析[J].海洋学报,增刊,2003,2:231-236.
[12]王志勇,王艺磊,林利民,等.福建官井洋大黄鱼AFLP指纹多态性的研究[J].中国水产科学,2002,9(3):198-213.
[13]黎中宝,方秀,陈锦,等.大黄鱼(Pseudosciaenacrocea)养殖群体遗传多样性的降低[J].海洋与湖沼,2009,40(4):446-450.
[14]陈锦,黎中宝,方秀,等.大黄鱼野生与养殖群体遗传结构的比较研究[J].海洋科学,2010,34(2):45-48.
[15]田明诚,徐恭昭,金日秀.大黄鱼形态特征的地理变异和地理种群问题[J].海洋科学集刊,1962:79-97.
[16]Wright S.Evolution in Mendelian population[J].Genetics,1931,16:91-159.
[17]王文文,常玉梅,梁利群.微卫星分析四个大黄鱼群体的遗传多样性[J].水产学杂志,2009,22(2):6-11.
[18]丁诗华,黄丽英,张海琪,等.大黄鱼(Pseudosciaena crocea)岱衢洋选育群体和官井洋养殖群体的遗传差异分析[J].海洋与湖沼,2006,37(1):41-46.
[19]Hebert P D N,Ratnasingham S,Jeremy R W.Barcoding animal life:cytochrome coxidase subunit 1 divergences among closely related species[J].Proceedings of the Royal Society of London.Series B:Biological Sciences,2003,270:96-99.
