T淋巴瘤EL-4细胞双受体表达研究①
2011-02-06南方医科大学生物技术学院分子免疫学研究所广州510515
张 媛 罗 微 马 骊 (南方医科大学生物技术学院分子免疫学研究所,广州 510515)
T淋巴瘤EL-4细胞双受体表达研究①
张 媛 罗 微 马 骊 (南方医科大学生物技术学院分子免疫学研究所,广州 510515)
目的:检测T淋巴瘤EL-4细胞T细胞受体(T cell receptor,TCR)的表达情况,为进一步研究T细胞等位基因排斥机制奠定基础。方法:以小鼠脾细胞作为阳性对照,分别提取脾细胞和EL-4细胞mRNA,逆转录为cDNA,RT-PCR扩增24个TCRBV家族CDR3区,结合基因扫描(GeneScan)和基因测序技术,对有表达的TCRBV家族进行CDR3谱型和序列分析。结果:小鼠脾细胞24 BV家族均有表达,各TCR BV家族CDR3谱型均呈高斯分布(Gaussian distribution),表明24 BV家族均为多克隆性。EL-4细胞被同时检测到TCR BV10和BV12家族表达,CDR3谱型均呈单峰,两个家族的RT-PCR产物经测序鉴定证实确属TCR序列,且均为框内编码。结论:EL-4细胞存在两套框内重排的TCRβ链,表明其TCRβ链可能存在等位基因排斥“缺陷”现象。本研究为探索T细胞等位基因排斥机制奠定了基础。
EL-4细胞株;T细胞受体(TCR);等位基因排斥“缺陷”;框内重排
T淋巴细胞通过其表面受体(T cell receptor,TCR)特异性识别抗原。在T细胞发育成熟过程中,TCR胚系基因中可变区(Variable region,V)、多样区(Diverse region,D)、连接区(Joining region,J)基因片段经历基因重组[1]。依据Burnett的克隆选择学说,T细胞表面受体遵循“一个淋巴细胞一个抗原受体”的原则,通过等位基因排斥机制,即一个等位基因座上抗原受体链的组成和表达能够抑制另一个等位基因座上V-(D)-J基因片段的进一步重组,确保T细胞表面只存在一种TCR,表现单特异性抗原识别能力[2]。随着对等位基因排斥机制研究的不断深入,近年来,越来越多的学者们发现,TCRα链存在等位基因排斥“缺陷”现象,即单克隆T细胞有两套重排的α链基因,甚至在蛋白质水平上检测到两种TCRα的表达,而关于TCRβ链等位基因排斥“缺陷”的研究报道较少[3-8]。
EL-4细胞为C57BL/6鼠T淋巴瘤细胞,作为单一细胞系发育成熟的T细胞株,是研究T细胞等位基因排斥机制的理想模型[9]。TCRβ链互补决定区3(CDR3)是直接与抗原肽结合的位点。在TCRβ链胚系基因片段重排过程中,CDR3区基因由BV末端、BD片段、BJ前端以及重排时V-D与D-J之间的插入序列(VnDnJ)组成,既是TCR上变化最多样的区,也是在抗原特异性识别中起最关键作用的区[10]。依据CDR3可变区V基因序列的同源性,可将65个TCR BV基因分为24个基因家族(即TCR BV1-BV20基因家族,其中BV5和BV8各有三个亚家族)[11],不同T细胞克隆其TCRCDR3长度和序列有所不同,通过设计各家族上游引物和TCR BC共用下游引物,可扩增出所有TCR BV家族的CDR3区,即可检测到T细胞中所有TCRβ链的表达[12]。本研究通过对EL-4细胞24个TCR BV家族分析发现,该细胞存在两套重排的TCRβ链,表明其TCRβ链存在等位基因排斥“缺陷”。本研究为进一步探讨T细胞等位基因排斥机制奠定了基础。
1 材料与方法
1.1 动物和细胞株 实验用C57BL/6小鼠(6~8周鼠龄)由南方医科大学实验动物中心提供;CD4+T淋巴瘤细胞株EL-4由本实验室保存。
1.2 主要试剂 Ficoll淋巴细胞分离液购自上海生物制品研究所;RPMI1640培养基、胎牛血清(美国Gibco公司);RNA提取试剂盒购自美国Omega Bio-Tek公司;第一链 cDNA合成试剂盒购自德国MBI Fermentas公司;去离子甲酰胺(美国Sigma公司);凝胶回收试剂盒(美国Omega Bio-Tek公司);DL2000 DNAMarker(日本TaKaRa公司)。
1.3 小鼠脾细胞悬液的制备 将C57BL/6小鼠拉颈处死,用75%酒精浸泡5分钟,无菌条件下,取出小鼠脾脏并研磨,过200目不绣钢筛制成单细胞悬液,将单细胞悬液轻轻加于淋巴细胞分离液上层,2 000 r/min水平离心20分钟,吸取白色淋巴细胞层于一无菌离心管中,细胞计数,用含10%胎牛血清的RPMI1640全培调整细胞浓度为2×106ml-1。
1.4 有限稀释法亚克隆EL-4细胞
1.4.1 小鼠饲养层细胞的制备 拉颈处死C57BL/6鼠,70%酒精浸泡消毒10分钟,用手术剪将小鼠腹部剪开一小口,剥开皮肤,露出腹腔。用注射器将4 ml RPMI1640溶液注入腹腔,用手指轻揉腹部1分钟,用注射器回抽腹腔液体,加入离心管。1 000 r/min离心10分钟。取上清,以含 10%胎牛血清的 RPMI1640完全培养液将细胞制成悬液。台盼蓝染色计数活细胞,调整细胞浓度为4×105个/ml。将细胞种入96孔板,0.05m l细胞悬液/孔,置37℃、5%CO2孵箱培养24小时。
1.4.2 亚克隆EL-4细胞 计数EL-4细胞,取130个细胞重悬于6.5ml全培中,即20个细胞/ml,100 μl/孔加入已接种饲养细胞的96孔板A、B、C三排中,即2个细胞/孔。余下2.9ml细胞悬液补加2.9ml全培,等倍稀释,即细胞浓度为10个/ml,100μl/孔加入上述96孔板的D、E、F三排中,即1个细胞/孔。余下2.2ml细胞悬液补加2.2ml全培,再等倍稀释,即细胞浓度为5个/ml,100μl/孔,加入96孔板的G、H两排中,即0.5个细胞/孔。培养4~5天,倒置显微镜下可观察到小的细胞克隆团,补加全培100μl/孔。第8~9天时,肉眼可见克隆细胞群,挑选单克隆团的EL-4细胞,转移至24孔板扩大培养。
1.5 引物设计与合成 参照文献[13,14]合成TCR BV基因家族上游引物24条,TCR BC下游引物2条(内侧引物5′端带Fam标记,见表1);由Invitrogen上海英骏生物技术有限公司合成。
1.6 总RNA提取和cDNA合成 按试剂盒条件,提取2×106个 EL-4细胞、小鼠脾细胞总RNA,取 1μg总RNA作为模板,用OligdT作引物,cDNA反应体系为40μl,按试剂盒条件合成cDNA。
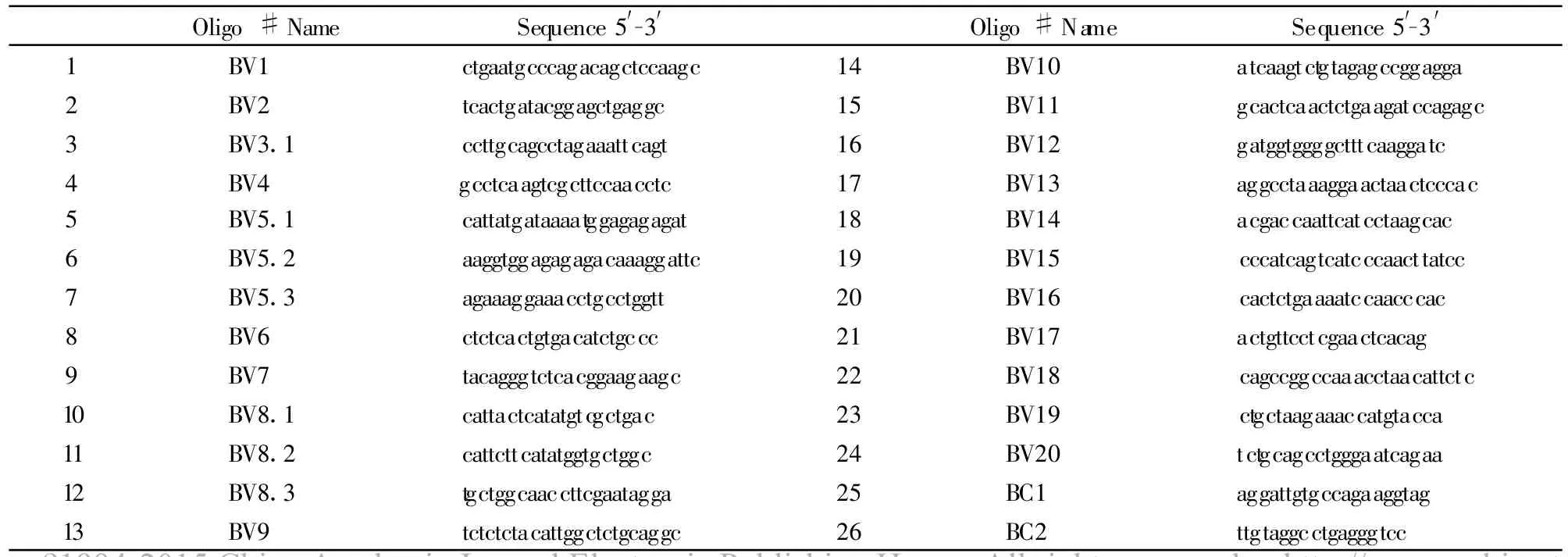
表1 TCR BV24家族、TCR BC1、TCRBC2引物设计Tab.1 The primers of TVR BV 24 fam ily,TCR BC1,TCRBC2
1.7 TCR BV半巢式PCR 第一轮PCR反应体系为25μl,含cDNA 模板1.2μl,10mmol/L dNTP 0.5μl,10×Buffer 2.5μl,25mmol/L氯化镁 1.5μl,24个TCR BV家族上游引物0.8μl和共用TCRBC下游外侧引物0.8μl,引物浓度均为10mmol/L,Taq DNA聚合酶0.65U。反应条件为:94℃变性3分钟;94℃1分钟,55℃1.5分钟,72℃2分钟,40个循环;72℃延伸10分钟。取PCR产物8μl在2%的琼脂糖凝胶上进行电泳,于-20℃保存备用;第二轮PCR反应体系25μl,含第一轮PCR产物2μl,10mmol/L dNTP 0.5 μl,10×Buffer 2.5μl,25 mmol/L氯化镁1.5μl,24个TCRBV家族上游引物0.8μl,FAM荧光标记的共用TCR BC下游内侧引物0.8μl,Taq DNA聚合酶0.65U。反应条件为:94℃变性3分钟;94℃1分钟,55℃45秒,72℃1分钟,40个循环;72℃延伸10分钟。
1.8 琼脂糖凝胶电泳 取PCR产物8μl经2%琼脂糖凝胶电泳,110 V,24分钟,采用凝胶成像系统照相,剩余产物-20℃保存备用。
1.9 GeneScan分析 取TCR BV第二轮各家族带荧光标记的PCR产物2μl,分别加入2μl去离子甲酰胺,0.5μl变性胶上样缓冲液(25 mmol/L EDTA,50 mg/ml b lue dextran),94℃变性 4分钟后,每管取2μl在373DNA序列分析仪(ABI,PerkinElmer)中电泳2小时(6%变性聚丙酰胺凝胶),计算机收集电泳过程中不同时间所出现的不同颜色和强度的荧光素,以显示出不同的位置、高度、颜色和形态的峰,Gene Scan 672软件分析。
1.10 PCR产物测序 经GeneScan分析后,取单克隆的TCR BV家族PCR产物2μl为模板,相应的上游V基因家族引物和下游不带荧光标记的BC引物各0.8μl,10 mmol/L dNTP 0.5μl,10×Buffer 2.5μl,25mmol/L氯化镁1.5μl,Taq DNA聚合酶0.65U进行二次PCR,总反应体积为25μl。产物经琼脂糖凝胶回收纯化,送Invitrogen上海英骏生物技术有限公司测序。
2 结果
2.1 琼脂糖凝胶电泳图 2%琼脂糖凝胶电泳结果显示,小鼠脾细胞24TCR BV家族PCR产物在250~500 bp位置均有清晰条带(图1),EL-4细胞株BV10和BV12基因家族PCR产物显示清晰条带(图2)。
2.2 GeneScan分析CDR3谱型图 小鼠脾细胞和EL-4细胞TCR Vβ基因家族GeneScan 672软件分析结果:小鼠脾细胞TCR BV各家族均为8个以上中间高、两端低的钟型峰图,呈高斯分布(8个以上峰型),相邻两条条带大小相差3 bp(图3)。EL-4细胞株BV10家族和BV12家族呈单峰(表2)。
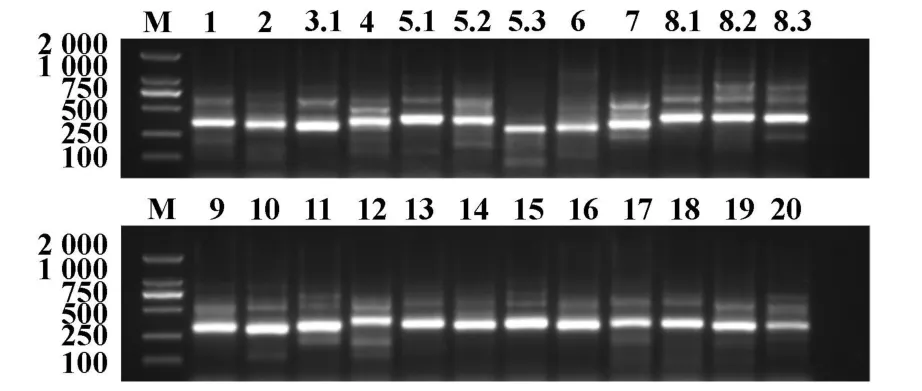
图1 小鼠脾细胞TCR BV家族PCR产物2%琼脂糖凝胶电泳图Fig.1 The products of TCR BV fam ily PCR ofmouse spleen cells analyzed on a 2%agarose gel
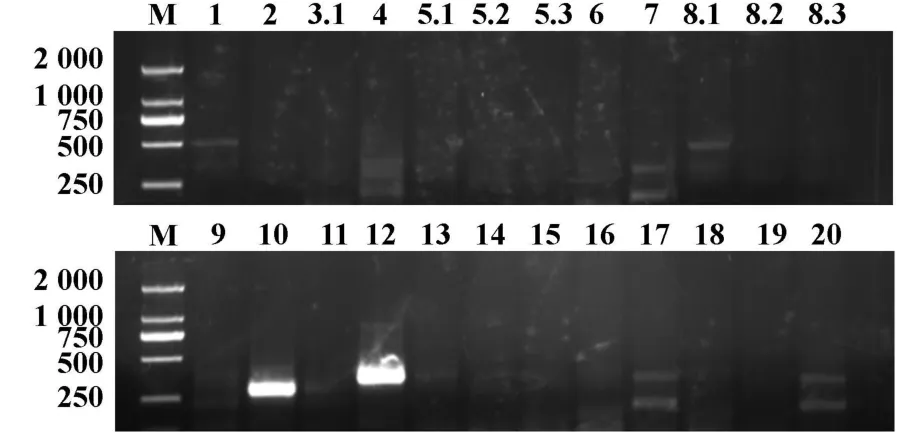
图2 EL-4细胞TCR BV家族PCR产物2%琼脂糖凝胶电泳图Fig.2 The products of TCR BV fam ily PCR of EL-4 cell line analyzed on a 2%agarose gel

图3 小鼠脾细胞TCRβ链24 BV家族CDR3谱型基因扫描图Fig.3 GeneScan analyze of CDR3 spectratype of TCRβchain 24 BV gene fam ilies ofmouse spleen cells
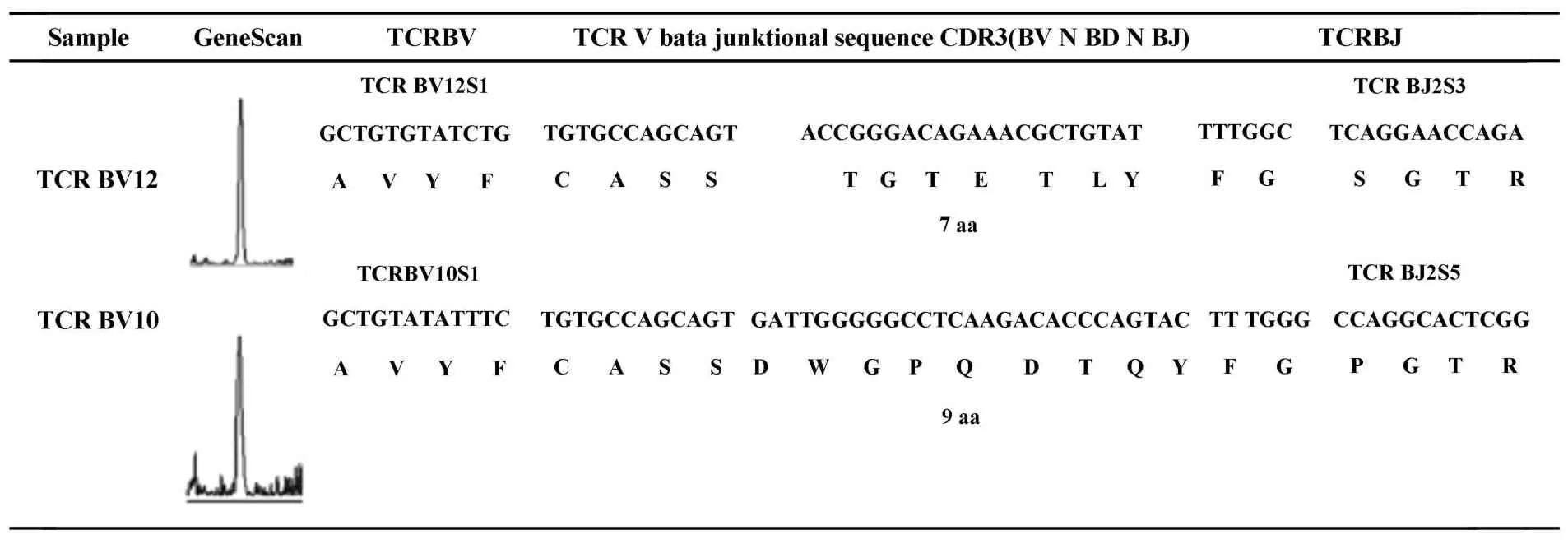
表2 TCR BV10和BV12家族CDR3基因和蛋白序列分析Tab.2 Analysis of CDR3 gene and protein sequences of the TCR BV10和BV12 in EL-4 cell line
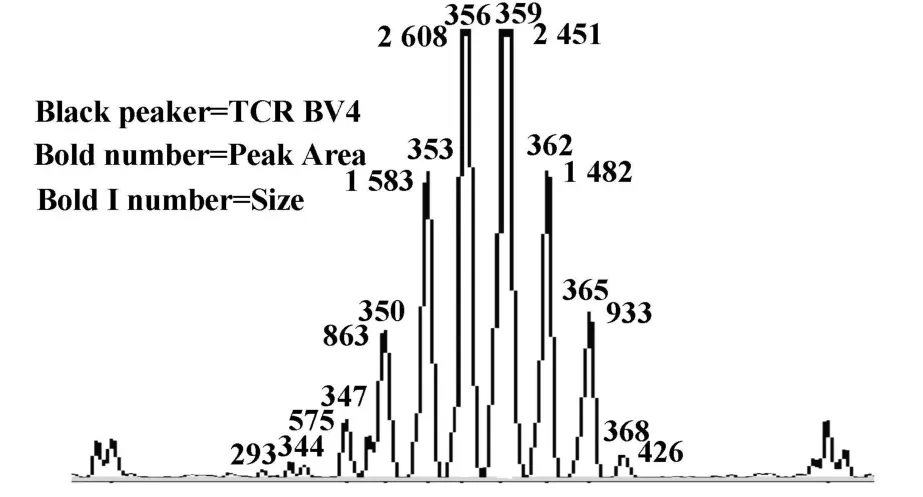
图4 小鼠脾细胞TCR BV4的PCR产物基因扫描分析图Fig.4 GeneScan distribution profiles of TCR BV4 in mouse sp leen cells
2.3 PCR产物片段大小和表达频率分析 软件自动分析标本中各TCR BV家族CDR3片段的长度和表达频率。图4列出小鼠脾细胞TCR BV4的片段大小和表达频率分析图。
2.4 EL-4细胞株TCR BV10和BV12家族PCR产物测序后CDR3基因和蛋白序列分析(见表2) 根据测序结果,按DDBJ、GeneBank及IMGT库中TCR VDJC标准基因序列,鉴定EL-4细胞TCR BV10和BV12家族PCR扩增序列均为TCR序列。
3 讨论
本研究通过分析T淋巴瘤细胞株EL-4细胞TCR的表达,检测到该细胞存在两套框内重排的TCRβ基因。为明确该两套TCR基因是同时存在于一个细胞内,还是因EL-4细胞株突变为一个混合细胞群而导致不同亚克隆株拥有不同TCRβ基因,我们对EL-4细胞株进行了亚克隆,挑选出三株生长良好的亚克隆细胞扩大培养,提取其总RNA并RT-PCR扩增24 TCR BV家族,结果表明,三个亚克隆株和母细胞株一样,均有TCRBV10和BV 12表达,且不同亚克隆株来源的BV10和BV12基因序列完全一致,表明BV10和BV12来源于单个克隆的EL-4细胞,且表达稳定。该实验结果提示,EL-4细胞株存在两套框内重排的TCRβ链,表明其TCRβ链可能存在等位基因排斥“缺陷”。
依据经典的免疫学理论,T细胞通过等位基因排斥机制确保一个淋巴细胞只表现单特异性抗原识别能力,作为早期细胞自身的耐受机制,等位基因排斥可以减少多特异性淋巴细胞的数量[15]。然而,自The Journalof Immunology杂志上报道了对白喉毒素特异的人T细胞株存在两个框内编码的TCR Vβ基因之后,T细胞存在两套重排TCR Vβ等位基因的现象才被相继报道,表明TCR Vβ链的等位基因排斥并非准确无误[16]。
为何T细胞能够同时存在两套重排的TCR Vβ链?研究者们推测可能是发生了等位基因排斥“缺陷”,具体解释为:(1)两个等位基因座上编码框内的VDJβ同时发生重排,从而逃避了等位基因排斥的抑制机制[17]。(2)在T细胞发育的双阳性阶段(CD4+CD8+),重组酶被异常激活,在TCRβ基因座上发生二次重排,使得单个细胞表达两条β链形成两种不同的TCR[18];(3)等位基因排斥取决于两条染色体上VDJβ重排的异步性,pre-TCR的作用是传导至少存在一个Vβ链信号而不是只能有一个Vβ链的信号[18];(4)第一个等位基因座上功能性重排的Vβ链表达与抑制第二个等位基因座上V-D-J重排存在时间的延迟,使得反馈抑制作用不能及时发挥作用[19]。
由于抗原受体的等位基因排斥由一系列复杂的机制调控,因此,尽管对等位基因排斥机制的研究已有数十年的历史,引起等位基因排斥“缺陷”的原因目前仍是个迷。本研究发现EL-4细胞株存在两套重排的TCRβ链,表明其TCRβ链存在等位基因排斥“缺陷”。EL-4细胞β链等位基因排斥“缺陷”的发生机制是什么?该现象究竟是等位基因排斥“缺陷”的偶然性?还是EL-4细胞作为T淋巴瘤细胞株这样一种特殊细胞群的代表所表现出来的某种特性?等位基因排斥“缺陷”机制与肿瘤发生发展之间是否存在某种关联?这些问题都有待我们更深入的研究和探讨。
1 Jackson A,Kondilis H D,Khor Betal.Regulation of T cell receptor beta allelic exclusion at a level beyond accessibility[J].Nat Immunol,2005;6(2):189-197.
2 Brady B L,SteinelNC,Bassing CH.Antigen receptorallelic exclusion:an update and reappraisal[J].JImmunol,2010;185(7):3801-3808.
3 Chou H S,BehlkeM,GodmbeSAetal.T cell receptor genes in an alloreactive CTL clone:implications for rearrangement and germ line diversity of variablegene segments[J].EMBO J,1986;5(9):2149-2155.
4 Pircher H,M ichalopoulosEE,Iwamoto Aetal.Molecular analysis of the antigen receptor of virus-specific cytotoxic T cellsand identifiction ofa new Vαfamily[J].Eur JImmunol,1987;17(12):1843-1846.
5 Malissen M,Trucy J,Letourneur Fetal.A T cell clone expresses two T cell receptorαgenes but uses one αβheterodimer for allorecognition and selfMHC-restricted antigen recognition[J].Cell,1988;55(1):49-59.
6 Malissen M,Trucy J,Jouvin-Marche Eetal.Regulation of TCRαandβ gene allelic exclusion during T-cell development[J].Immunol Today,1992;13(8):315-322.
7 FurutaniM,YanagiY,Fujisawa Ietal.Posttranscriptionalallelic exclusion of two functionally rearranged T cell receptorαgenes[J].Int Immunol,1989;1(3):281-288.
8 Eshima K,Suzuki H,Yamazaki Setal.Derivation of T-cell receptorαchain double expresser lines from normalmurinemature T cells[J].Immunology,1996;87(2):205-212.
9 Okada CY,Wong C P,Denney DWetal.TCR vaccines for active immunotherapy of T cellmalignancies[J].J Immunol,1997;159(11):5516-5527.
10 邹红云,马 骊,姚新生etal.Jurkat细胞TCR基因重排对 BV CDR3的影响[J].南方医科大学学报,2006;26(7):939-943.
11 Panneteer C,Even J,Kourilsky P.T-cell repertoire diversity and clonal expansions in normal and clinical samples[J].Immunol Today,1995;16(4):176-181.
12 Preesman AH,Hu H Z,TilanusMetal.T-cell receptor Vβ-family usage in primary cutaneousand primery nodal T-cell non-hodgkin's lymphomas[J].J Invest Dermatol,1992;99:587-593.
13 Pannetier C,CochetM,Darche Setal.The sizes of the CDR3 hypervariable regionsof themurine T-cell receptorβchains vary as a function of the recombined germ-line segments[J].Immunology,1990;90(9):4319-4323.
14 Yoshida R,Yoshioka R,Yamane Setal.A newmethod for quantitative analysisof themouse T-cell receptor V region repertoires:comparison of repertoires among strains[J].Immunogenetics,2000;52(1-2):35-45.
15 Ji Q,Perchellet A,Goverman JMetal.Viral infection triggers central nervous system autoimmunity via activation of CD8+T cells expressing dual TCRs[J].Nat Immunol,2010;11(7):628-334.
16 Triebel F,Breathnach R,GrazianiMetal.Evidence forexpression of two distinctT cell receptorβ-chain transcripts in a human diphtheria toxoidspecific T cell c lone[J].JImmunol,1988;140(1):300-304.
17 Matis LA,Ezquerra A,Coligan JE.Expression of two distinct T cell receptorα/β heterodimers by an antigen-specific T cell c lone[J].JExp Med,1988;168:2379-2384.
18 Davodeau F,Peyrat M A,Romagne Fetal.Dual T cell receptor beta chain expression on human T lymphocytes[J].JExp Med,1995;181(4):1391-1398.
19 Schittek B,Unkelbach E,Rajewsky K.Violation ofallelic exclusion of the T cell receptor beta genes in a helper T cell clone[J].Int Immunol,1989;1(3):273-280.
[收稿2010-11-12]
(编辑 倪 鹏)
Expression of two distinct T cell receptorsby EL-4 T lymphoma cells
ZHANGYuan,LUOWei,MALi.InstituteofMolecularImmunology,SchoolofBiotechnology,SouthernMedicalUniversity,Guangzhou510515,China
Objective:To investigate the expression of T cell receptor(TCR)inEL-4 T lymphoma cells.Methods:Mouse spleen cells were used as positive control,total RNA was isolated from mouse spleen cells and EL-4 cell line.Comp lementarity Determining Region 3(CDR3)of24 TCR BV gene subfamilieswas amplified by the reverse transcription polymerase chain reaction(RT-PCR).To determ ine the spectratypes and sequences of CDR3 region of TCRBV subfam ilies expressed by EL-4,PCR productswere analyzed by TCRGeneScan Technique and DNA sequencer,respectively.Results:24 TCRBV subfam ilies ofmouse spleen cellswere polyclonal,the spectratypes of TCRCDR3 regionwere showed Gaussian distribution.EL-4 was shown detected the expression of TCR BV10 and BV12 simultaneously,spectratypes of which showed single peak,and the TCR sequences of BV10 and BV12 were confirmed by DNA sequencer and they were both in-frame rearrangements.Conclusion:Two sets of in-frame rearrangements of TCRβchain exist in EL-4,and theremaybe‘defects'in allelic exclusion in TCRβchain.It provides abasis for future research of allelic exclusionmechanism.
EL-4 cell line;T cell receptor(TCR);‘Defect'in allelic exclusion;In-frame rearrangement
R392.12
A
1000-484X(2011)05-0404-05
10.3969/j.issn.1000-484X.2011.05.005
①本文受国家自然科学基金(30972680、30771952)和教育部新世纪优秀人才支持计划(NCET-07-0410)资助
张 媛(1987年-),女,在读硕士,E-mail:zhangy_1987@126.com;
及指导教师:马 骊(1971年-),女,教授,博士生导师,主要从事T细胞识别活化的基础和应用研究,E-mail:maryhmz@126.com。