滇牡丹分类处理的细胞学与分子生物学证据
2010-12-05成仿云
孔 红,成仿云
(1.廊坊师范学院 生命科学学院,河北 廊坊 065000;2.北京林业大学 园林学院,北京100083)
滇牡丹Paeonia delavayi属于芍药科Paeoniaceae芍药属Paeonia牡丹组(Sect.Moutan)肉质花盘亚组(Subsect.Delavayanae)[1],为中国西南地区特有种。主要分布于云南中部至西北部、四川西南部和西藏东南部,是芍药属分布最南的一个牡丹类群,在芍药属的起源、演化和地理分布研究上有重要的学术价值,且滇牡丹拥有黄色花基因,在杂交育种上具有重要应用价值[2]。有关滇牡丹的研究非常广泛,而且滇牡丹的分类一直存在争议,始终是研究的热点问题。笔者主要就近年来国内外开展的有关滇牡丹分类的细胞学及分子生物学方面的研究进行了总结,目的是指出问题,供今后研究参考。
1 分类简史和分类处理
滇牡丹叶裂片数目、叶裂片宽度、花色、苞片数量具丰富的多样性[2],这些性状是滇牡丹形态分类的主要依据。1886年,Franchet根据Delavay采自云南丽江的紫色花标本和洱源的黄色花标本发表了滇牡丹和黄牡丹P.lutea Delavay ex Franch.[3]。1904年,Finet和Gngnepain将黄牡丹降为滇牡丹的变种 P.delavayi var.lutea Finet ex Gngnepain[4]。1913年,Rehder和Wilson根据采自四川康定叶裂片狭窄的标本发表了狭叶牡丹P.delavayi var.angustiloba Rehder et Wilson[5],而在1921年,Komarov根据原地采集的标本发表了保氏牡丹P.potanini Komarov[6]。1943年及1946年,Stern分别将Stapt和Stern发表的金莲牡丹P.trollioides Stapt ex Stern以及Bean发表的银莲牡丹P.delavayi var.alba Bean处理为保氏牡丹的一个变种 P.potanini var.trollioides(Stapt ex Stern)Stern[7]和一个变型 P.potanini f.alba(Bean)Stern[8]。这些类群构成了滇牡丹复合群,以后关于滇牡丹复合群的分类一直存在争议,归纳起来有以下4种观点:①认可3种1变种 1变型[8-11],即滇牡丹P.delavayi Franch.,黄牡丹P.lutea Delavay ex Franch.,保氏牡丹 P.potanini Komarov,金莲牡丹 P.potanini var.trollioides(Stapt ex Stern)Stern,银莲牡丹 P.potanini f.alba(Bean)Stern;②认可 2种 1变种[12],即滇牡丹 P.delavayi Franch.,黄牡丹 P.lutea Delavay ex Franch,狭叶牡丹 P.delavayi var.angustiloba Rehder et Wilson;③认可 1 种 2 变种[13-14],即滇牡丹 P.delavayi Franch.,黄牡丹 P.delavayi var.lutea Finet ex Gngnepain,狭叶牡丹 P.delavayi var.angustiloba Rehder et Wilson;④将滇牡丹复合群归并为1个种,即滇牡丹P.delavayi Franch.,理由是这一类群在居群内花色的变化从白色、纯黄色、黄色有紫红色斑块、橙色、红色至红紫色;花瓣数目4~10枚,花丝和花药颜色从黄色变至橙色、紫红色;心皮数目2~5或3~6个,颜色绿色至紫红色;叶裂片数68~312片,叶裂片宽度为0.40~2.86 cm;苞片1~5片;萼片2~5,或4~6,或7~9片,绿色至紫红色;花盘绿色、黄色、红色或紫红色。另外,这个类群一致地兼有性生殖和无性繁殖,根纺錘状加粗。没有一个性状或性状的组合可以把它划分为不同种甚至种下分类群[1,15]。这一观点得到一些研究者的赞同[2,16-26],并广泛开展了细胞学及分子生物学方面的研究。
2 细胞学证据
2.1 染色体核型
染色体是基因的载体,是生物进化发育、遗传变异的物质基础。染色体核型是植物体细胞染色体所有可测定的表型特征的总称,主要指染色体数目和形态特征[27]。
滇牡丹的染色体基数x=5,染色体数目2n=2x=10,为二倍体,属大型染色体[28-30]。关于滇牡丹的核型分析研究报道较多[28-31],核型的主要特征归纳于表1[2,32]。龚洵等[2]认为,滇牡丹 12 个居群中出现了8种核型。核型的差异不仅表现在不同染色体类型数目上,而且表现在随体的数目(0~6)和位置上,以及次缢痕的数目(0~5)和位置上,这种核型的多样性反映出了滇牡丹居群水平上的遗传多样性。这与牡丹组所有种的染色体核型没有明显差异的结论[33-34]不太一致。李懋学等[35]报道,牡丹染色体的随体属于微型随体,在牡丹同一种或同一品种中,不同作者报道的随体数目不一致的现象常见。另外,Ag-Nor数普遍多于常规染色可见到的随体数目,表明常规染色所见的随体数目并不是一个稳定可靠的核型特征。滇牡丹的染色体随体很小[28-30],同样存在以上问题,随体数目和位置差异很不可靠。因此,仅凭随体的有无及数目进行种及种下类群的核型结构比较是很难的。
由表1可知,滇牡丹核型由中部着丝点染色体(m),近中部着丝点染色体(sm)和近端部着丝点染色体(st)等3种染色体类型组成。根据染色体类型的数目不同,滇牡丹不同居群的核型可分为3种:①2n=10=6m+2sm+2st,②2n=10=7m+1sm+2st,③2n=10=5m+2sm+3st。其中 10个滇牡丹居群属于核型①,为Stebbins[36]核型分类的 2A型[28-30],只有禄劝居群、丽江鲁甸居群染色体类型的数目略有不同,表明滇牡丹复合群核型基本一致。
2.2 染色体带型
肖调江等[31]对滇牡丹复合群5个类群的Giemsa C-带进行了全面研究,龚洵等[32]对滇牡丹黄色花类型8个居群的Giemsa C-带进行了研究,结果表明所有染色体都在着丝点附近显示出了Giemsa C-带,长臂上没有显带,只有短臂上的Giemsa C-带的数量和位置表现了一定的差异。可见滇牡丹各居群关系非常密切,可能由同一祖先演化而来,短臂上Giemsa C-带的差异说明了各居群分别对其生态环境形成了适应性而产生了遗传分化[2]。
3 分子生物学证据
于玲等[37]对滇牡丹复合群中的黄牡丹和狭叶牡丹叶片蛋白谱带进行了相似性定量分析,结果表明两者之间蛋白谱带排序间距较小,具有较近的亲缘关系。邹喻苹等[38]对滇牡丹复合群中的紫牡丹和黄牡丹进行了随机扩增多态性DNA标记(RAPD,random amplified polymorphic DNA)分析,结果表明两者遗传距离很近,亲缘关系紧密,可看作同一种的2个居群。对滇牡丹进行分子标记最全面的报道是杨淑达等[18]应用简单重复序列区间扩增多态性(ISSR,inter-simplesequencerepeat)标记对滇牡丹的遗传多样性开展的研究,结果表明,滇牡丹在分子水平上遗传多样性水平较高,居群间表现出一定的遗传分化。非加权配对算术平均法(UPGMA,unweighted pair-group method with arithmetic means)聚类分析结果完全支持Hong等[1]对滇牡丹的分类处理。
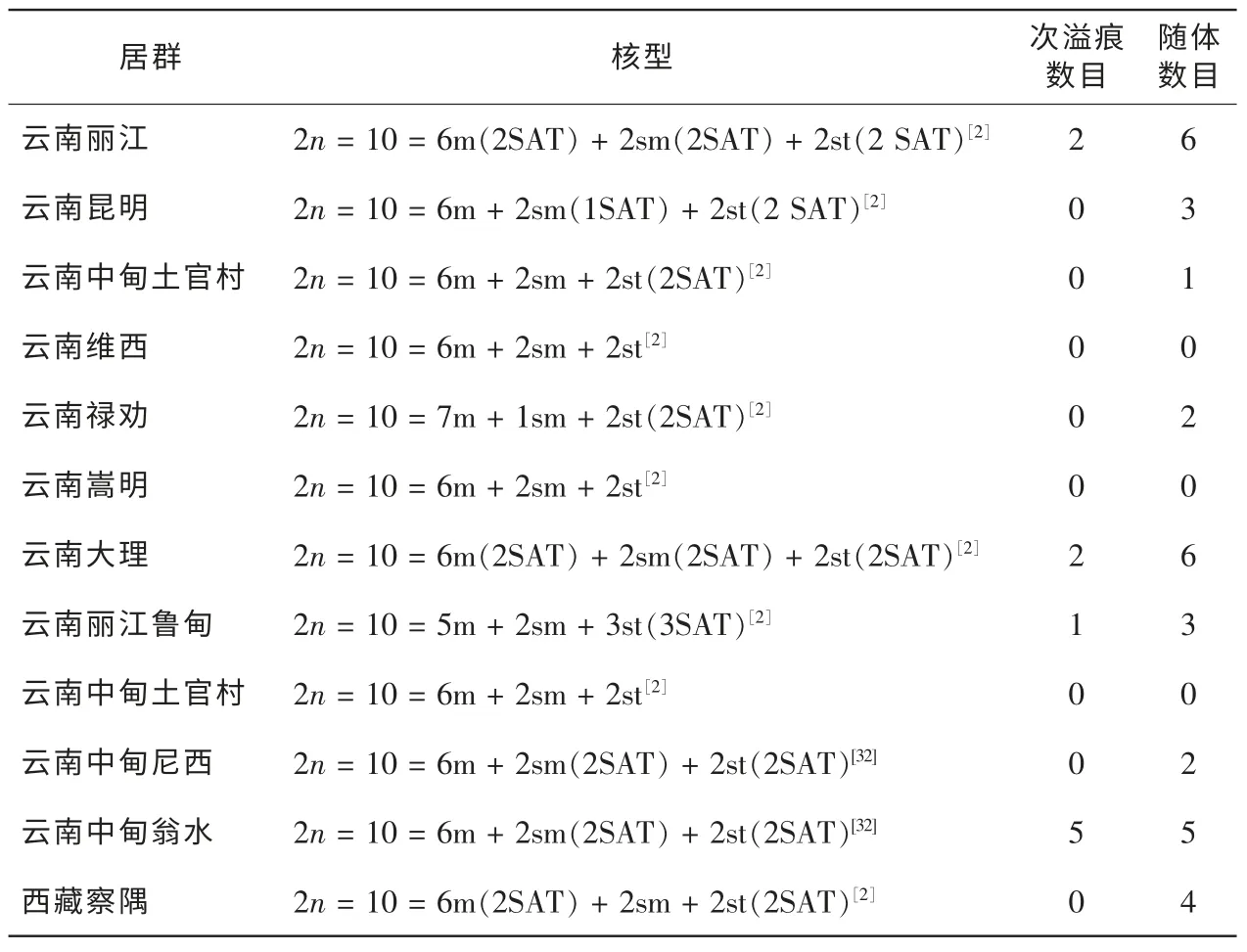
表1 12个滇牡丹居群的核型Table 1 Karyotype of 12 population of Paeonia delavayi
近年来,基因序列分析开始用于滇牡丹的研究中。Sang 等[39-41],Ferguson 等[24],Tank 等[26],赵宣等[22]分别对滇牡丹的2个居群(滇牡丹复合群中的P.delavayi和P.lutea)进行了核糖体DNA的ITS序列、叶绿体matK基因、乙醇脱氢酶基因、GPAT基因序列分析,结果表明两者亲缘关系很近。但遗憾的是,上述研究中只涉及了滇牡丹的2个居群,代表性有限,因此还需要加强滇牡丹不同居群基因序列的研究,为滇牡丹复合群合并为滇牡丹一个种提供更全面的分子依据。
4 讨论
滇牡丹分类研究经历了100多年的发展,从形态分类开始,到逐渐开展的染色体核型、Giemsa C-带、分子标记及基因测序,逐渐证明了滇牡丹复合群合并为滇牡丹一个种的合理性,但滇牡丹基因测序所取材料代表性有限,染色体银染带及原位杂交等研究尚属空白,还有待进一步研究。将染色体核型、Giemsa C-带、银染带、染色体原位杂交、分子标记及基因测序等研究相结合,可为滇牡丹复合群合并为滇牡丹1个种提供充分的科学依据。滇牡丹种内形态变异很大,因此,洪德元等[1,15]认为滇牡丹不宜划分任何种下类型。由于滇牡丹复合群中P.delavayi和P.lutea等不同居群在引种中表现出实生后代的遗传稳定性以及花色素组成上的显著差异等,李嘉珏[42]对滇牡丹复合群的完全合并而没有任何种下类群的处理持有保留意见。Haw[25]赞同滇牡丹复合群归并为滇牡丹1个种,并在种下设了2个变种5个变型,但这一种下等级的处理未见被采用。滇牡丹是否应建立种下类群,还有待于进一步研究和探讨。
[1]HONG Deyuan,PAN Kaiyu,YU Hong.Taxonomy of the Paeonia delavayi complex(Paeoniaceae)[J].An Missouri Bot Gard,1998,85(4):554-564.
[2]龚洵,潘跃芝,杨志云.滇牡丹的多样性和现状评估[J].西北植物学报,2003,23(2):218-223.GONG Xun,PAN Yuezhi,YANG Zhiyun.The diversities and value of present situation of Paeonia delavayi[J].Acta Bot Boreal-Occident Sin,2003,23(2):218-223.
[3]FRANCHET A.Paeonia delavayi et Paeonia lutea[J].Bull Soc Bot France,1886,33:382-383.
[4]FINET F C,GAGNEPAIN F.Contributions a La Flore de I’Asie Orientale[J].Bull Soc Bot France,1904,51:524.
[5]REHDER A,WILSON E H.Paeonia delavayi var.angustiloba[M]//SARGENT C S.Plantae Wilsonianae.Cambridge:The University Press,1913:318.
[6]KOMAROV V L.Paeonia potanini[J].Noth Syst ex Herb Horti Bot Petropolitani,1921,2(2):7.
[7]STERN F C.Paeonia potanini var.trollioides[J].J Roy Hort Soc,1943,68:125.
[8]STERN F C.A Study of the Genus Paeonia[M].London:The Royal Horticulture Society,1946:46-47.
[9]方文培.中国芍药属的研究[J].植物分类学报,1958,7(4):297-323.FANG Wenpei.Notes on Chinese paeonies in China[J].Acta Phytotaxon Sin,1958,7(4):297-323.
[10]龚洵.野牡丹复合群的研究[D].昆明:中国科学院昆明植物研究所,1990.GONG Xun.A Study of Paeonia delavayi Comples[D].Kunming:Chinese Academy of Sciences.Kunming Institute of Botany,1990.
[11]李嘉珏.中国牡丹与芍药[M].北京:中国林业出版社,1999:54-55.
[12]吴征镒.云南植物名录[M].昆明:云南人民出版社,1984:122-123.
[13]潘开玉.中国植物志:第27卷[M].北京:科学出版社,1979:37-48.
[14]潘开玉.芍药科分布格局及其形成的分析[J].植物分类学报,1995,33(4):340-349.PAN Kaiyu.The analysis of distribution pattern in the Paeoniaceae and its formation[J].Acta Phytotaxon Sin,1995,33(4):340-349.
[15]洪德元,潘开玉.芍药属牡丹组的分类历史和分类处理[J].植物分类学报,1999,37(4):351-368.HONG Deyuan,PAN Kaiyu.Taxonomical history and revision of Paeonia Sect.Moutan[J].Acta Phytotaxon Sin,1999,37(4):351-368.
[16]李保印,周秀梅,蒋细旺,等.牡丹种及品种亲缘关系的分子生物学研究进展[J].河南农业大学学报,2008,42(1):121-126.LI Baoyin,ZHOU Xiumei,JIANG Xiwang,et al.Research advancement on the relationship between Chinese tree peony species and cultivars at the molecular level[J].J Henan Agric Univ,2008,42(1):121-126.
[17]林启冰,周志钦,赵宣,等.基于Adh基因家族序列的牡丹组(Sect.Moutan DC.)种间关系[J].园艺学报,2004,31(5):627-632.LIN Qibin,ZHOU Zhiqin,ZHAO Xuan,et al.Interspecific relationships among the wild species of Paeonia Sect.Moutan DC.based on DNA sequences of Adh gene family[J].Acta Hortic Sin,2004,31(5):627-632.
[18]杨淑达,施苏华,龚洵,等.滇牡丹遗传多样性的ISSR分析[J].生物多样性,2005,13(2):105-111.YANG Shuda,SHI Suhua,GONG Xun,et al.Genetic diversity of Paeonia delavayi asrevealed by ISSRs[J].Biodiversity Sci,2005,13(2):105-111.
[19]ZHOU Zhiqin,PAN Kaiyu,HONG Deyuan.Phylogenetic analyses of Paeonia section Moutan based on morphological data[J].Acta Phytotaxon Sin,2003,41(5):436-446.
[20]周志钦,潘开玉,洪德元.牡丹组野生种间亲缘关系和栽培牡丹起源研究进展[J].园艺学报,2003,30(6):751-757.ZHOU Zhiqin,PAN Kaiyu,HONG Deyuan.Advances in studies on relationships among wild tree peony species and the origin of cultivated tree peonies[J].Acta Hortic Sin,2003,30(6):751-757.
[21]ZHOU Zhiqin.Taxonomy,geographic distribution and ecological habits of tree peonies[J].Genet Resour Crop Evol,2006,53(1):11-22.
[22]赵宣,周志钦,林启冰,等.芍药属牡丹组(Paeonia sect.Moutan)种间关系的分子证据:GPAT基因的PCR-RFLP和序列分析[J].植物分类学报,2004,42(3):236-244.ZHAO Xuan,ZHOU Zhiqin,LIN Qibin.et al.Molecular evidence for the interspecific relationships in Paeonia section Moutan:PCR-RFLP and sequence analysis of glycerol-3-phosphate acyltransferase(GPAT)gene[J].Acta Phytotaxon Sin,2004,42(3):236-244.
[23]赵宣,周志钦,林启冰,等.基于多基因序列和形态性状的牡丹组种间关系[J].植物分类学报,2008,46(4):563-572.ZHAO Xuan,ZHOU Zhiqin,LIN Qibin.et al.Phylogenetic analysis of Paeonia sect.Moutan(Paeoniaceae)based on multiple DNA fragments and morphological data[J].Acta Phytotaxon Sin,2008,46(4):563-572.
[24]FERGUSON D,SANG T.Speciation through homoploid hybridization between allotetraploids in peonies(Paeonia)[J].Proc Nat Acad Sci USA,2001,98(7):3915-3919.
[25]HAW S G.Paeonia delavayi,a variable species[J].New Plantsman,2001,8(4):251-253.
[26]TANK D C,SANG T.Phylogenetic utility of the glycerol-3-phosphate acyltransferase gene:evolution and imphcations in Paeonia(Paeoniaceae)[J].Mol Phylogenet Evol,2001,19(3):421-429.
[27]侯小改,段春燕,刘素云,等.中国牡丹染色体研究进展[J].中国农学通报,2006,22(2):307-309.HOU Xiaogai,DUAN Chunyan,LIU Suyun,et al.Advances on chromosome study of Chinese tree peony[J].Chin Agric Sci Bull,2006,22(2):307-309.
[28]龚洵,顾志建,武全安.黄牡丹7个居群的细胞学研究[J].云南植物研究,1991,13(4):402-410.GONG Xun,GU Zhijian,WU Quanan.A cytological study of seven populations in P.delavayi var.lutea[J].Acta Bot Yunnan,1991,13(4):402-410.
[29]李思锋,于兆英,周俊彦.黄牡丹的核型分析[J].武汉植物学研究,1989,7(2):107-111.LI Sifeng,YU Zaoying,ZHOU Junyan.Karyotype analysis of P.delavayi var.lutea[J].J Wuhan Bot Res,1989,7(2):107-111.
[30]杨涤清,朱燮桴.草芍药、野牡丹和黄牡丹的核型研究[J].云南植物研究,1989,11(2):139-144.YANG Diqing,ZHU Xiefu.Karyotypic studies of Paeonia obovata,P.delavayi and P.delavayi var.lutea[J].Acta Bot Yunnan,1989,11(2):139-144.
[31]肖调江,龚洵,夏丽芳,等.滇牡丹复合群的Giemsa C-带比较研究[J].云南植物研究,1997,19(4):395-401.XIAO Tiaojiang,GONG Xun,XIA Lifang,et al.C banding patterns in Paeonia delavayi complex of genus Paeonia[J].Acta Bot Yunnan,1997,19(4):395-401.
[32]龚洵,肖调江,顾志建,等.黄牡丹 8个居群的 Giemsa C-带比较研究[J].云南植物研究,1999,21(4):477-482.GONG Xun,XIAO Tiaojiang,GU Zhijian,et al.Giemsa C-banding patterns in 8 populations of Paeonia delavayi var.lutea[J].Acta Bot Yunnan,1999,21(4):477-482.
[33]裴颜龙.牡丹复合体的研究[D].北京:中国科学院植物研究所,1993:1-80.PEI Yanlong.A Study of Paeonia suffruticosa Complex[D].Beijing:Chinese Academy of Sciences.Institute of Botany,1993:1-80.
[34]洪德元,张志宪,朱相云.芍药属的研究(1)国产几个野生种核型的报道[J].植物分类学报,1988,26(1):33-43.HONG Deyuan,ZHANG Zhixian,ZHU Xiangyun.Studies on the genus Paeonia(1)Report of karyotypes of some wild species in China[J].Acta Phytotaxon Sin,1988,26(1):33-43.
[35]李懋学,张赞平.作物染色体及其研究技术[M].北京:中国农业出版社,1996:286-290.
[36]STEBBINS G L.Chromosomal Evolution in Higher Plant[M].London:Edward Arnold,1971:87-90.
[37]于玲,何丽霞,李嘉珏,等.牡丹野生种间蛋白质谱带的比较研究[J].园艺学报,1998,25(1):99-101.YU Ling,HE Lixia,LI Jiajue,et al.Comparative studies on protein zones of wild tree peony species[J].Acta Hortic Sin,1998,25(1):99-101.
[38]邹喻苹,蔡美林,王子平.芍药属牡丹组的系统学研究—基于RAPD分析[J].植物分类学报,1999,37(3):220-227.ZOU Yuping,CAI Meilin,WANG Ziping.Systematic studies on Paeonia section Moutan DC.based on RAPD analysis[J].Acta Phytotaxon Sin,1999,37(3):220-227.
[39]SANG T,CRAWFORD D J,STUESSY T F.Documentation of reticulate evolution in peonies(Paeonia)using internal transcribed spacer sequences of nuclear ribosomal DNA:implications for biogeography and concerted evolution[J].Proc Nat Acad Sci USA,1995,92(15):6813-6817.
[40]SANG T,CRAWFORD D J,STUESSY T F.Chloroplast DNA phylogeny,reticulate evolution and biogeography of Paeonia(Paeoniaceae)[J].Am J Bot,1997,84(8):1120-1136.
[41]SANG T,DONOGHUE M J,ZHANG D.Evolution of alcohol dehydrogenase genes in peonies(Paeonia):phylogenetic relationships of putative nonhy-brid species[J].Mol Biol Evol,1997,14(10):994-1007.
[42]李嘉珏.中国牡丹品种图志[M].北京:中国林业出版社,2005:4-30.
