12种寄主来源的茄科雷尔氏菌16S-23S rDNA间隔区序列比较
2010-11-30佘小漫何自福李华平
佘小漫, 何自福, 李华平, 虞 皓
(1.广东省农业科学院植物保护研究所,广州 510640; 2.华南农业大学资源环境学院,广州 510642)
茄科雷尔氏菌[Ralstonia solanacearum(Smith)Yabuuchi et al.]是世界上最重要的植物病原细菌之一,广泛分布于热带、亚热带及温带地区。该病原细菌的寄主范围很广,可侵染55科数百种植物[1]。在我国,目前已报道有番茄、辣椒、茄子、马铃薯、烟草、花生、生姜、沙姜、桑、空心菜和桉树等 10余种作物受到该病原细菌的危害。
茄科雷尔氏菌种内存在明显生理分化和遗传差异。根据对不同寄主植物种类的致病性不同,将该病原细菌其划分为5个生理小种(race)[2-4];而根据菌株对3种二糖和3种己醇氧化能力的差异,又可以将其划分为6个生化变种(biovar)[3,5-6]。Li等[7]通过对16Sr RNA基因序列分析将茄科雷尔氏菌分为2个簇(或亚种),并与 Cook等[8]的 2个区组(division)相对应。另外,分子系统进化分析还可以将该病原细菌区分为亚洲组(Asiaticum)、美洲组(Americanum)和非洲亚组(African subdivision)等3个地理组[9-11]。因此,分类意义上的茄科雷尔氏菌明显是一个复合种。
对于茄科雷尔氏菌这个复合种,不同来源,尤其是不同寄主植物来源的菌株间系统进化在分子水平上有无差异?先前的研究结果显示,不同地理区域和寄主植物来源的茄科雷尔氏菌菌株存在丰富的遗传多样性[12-13],但其16S rDNA十分保守[14]。在茄科雷尔氏菌的分类与系统进化研究时,16S-23S rDNA ITS序列分析可以作为16S rDNA序列分析的补充[15]。因此,本文通过对来源于12种寄主植物的茄科雷尔氏菌代表菌株的16S-23S rRNA基因间隔区序列的研究,进一步探讨这些不同寄主来源菌株的分子特征,全面认识该病原菌的生理分化和遗传变异,为控制该病原菌的危害奠定基础。
1 材料与方法
1.1 供试菌株
供试菌株共21株,分别分离自广东不同寄主植物,并鉴定其所属生化变种[16](表1)。所有菌株均在含2,3,5-氯化三苯基四氮唑(TTC)平板培养基上,28℃培养3 d。选取白边宽中间粉红色的单菌落用于试验。

表1 供试的21个茄科雷尔氏菌菌株
1.2 菌株基因组DNA的提取
按Boucher等[17]方法提取各菌株基因组DNA。
1.3 PCR引物
用原核生物16S-23S rDNA ITS通用引物[18]进行 PCR,其序列为:1493f(5′-AGTCGTAACAAGGTAGCCGT-3′)和 23r(5′-GTGCCAAGGCATCCACC-3′)。上述引物均由上海生工生物技术工程服务有限公司合成。
1.4 PCR扩增体系
以提取的基因组DNA为模板进行扩增。PCR反应体系(25μL)含如下成分:DNA模板约 20~25 ng,10×PCR buffer 2.5μL,dNTPs 200μmol/L,2个引物各0.4μmol/L和3 U Taq酶(TaKaRa)。PCR扩增条件为:94℃预变性4 min,之后94℃变性30 s,48℃退火30 s,72℃延伸1 min,30个循环,72℃最终延伸10 min。PCR反应结束后,取6μL扩增产物在0.8%琼脂糖凝胶上电泳,用全自动凝胶成像系统(SYNGENE公司)观察和分析电泳结果。
1.5 序列分析
对PCR扩增获得的茄科雷尔氏菌21个株菌16S-23SrDNA ITS进行双向测序,以获得各菌株全序列,并将这些序列分别与19个茄科雷尔氏菌菌株、2个 blood disease bacterium(BDB)菌株(表 2)的16S-23S rDNA ITS进行比较分析,应用DNAstar 5.0软件构建系统发育树。

表2 参与分析的茄科雷尔氏菌和BDB菌株及其16S-23Sr DNA ITS序列1)
2 结果分析
2.1 PCR扩增16S-23Sr DNA ITS序列结果
应用原核生物通用引物23r和1 493f分别对21个茄科雷尔氏菌菌株进行PCR扩增,其产物电泳结果显示,从21个菌株基因组DNA中均能成功地扩增出约590 bp特异片段,与预期片段大小一致(图 1)。
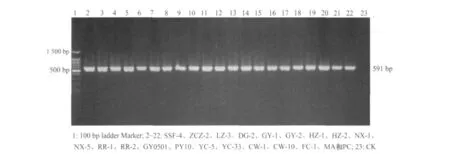
图1 PCR扩增21个菌株16S-23Sr DNA ITS的产物电泳结果
2.2 16S-23SrDNA ITS序列分析结果
对21个菌株PCR产物进行双向测序,将测序结果进行拼接,结果表明:除菌株HZ-1扩增的片段长度为586 bp(GenBank登录号:FJ744134)外,其他20个菌株扩增的片段大小均为591 bp(GenBank登录号见图2)。去除两端16S rDNA和23 rDNA序列,菌株HZ-1的ITS序列长度为498 bp,其他20个菌株的ITS序列全长均为503 bp。
序列比较结果显示,21个菌株之间ITS序列相似性最高为100%,最低为95.4%;菌株HZ-1与R.solanacearum GMI1000的16S-23SrDNA ITS序列相似性为 95.8%,而其他 20个菌株序列与 R.solanacearum GMI1000的16S-23SrDNA ITS序列相似性均大于99.2%(表3)。进一步比对发现,菌株HZ-1与其他20个菌株分别有33~37 bp不等的碱基差异;在其余20个菌株中,菌株 DG-2、HZ-2、GY-2 、RR-1 、RR-2 、NX-1 、NX-5 、PY-10 、GY0501 和FC-1 之间 ,菌株 GY-1、LZ-3、YC-33 、ZCZ-2、MA 和PC之间,以及菌株 YC-5、CW-1和CW-10之间,其ITS序列均完全一致,其余菌株间也仅有1~4个不等的碱基差异。
根据21个菌株以及来源于GenBank的22个菌株(包括 P.pickettii外围菌株)16S-23S rDNA ITS,利用DNAstar软件构建系统发育树,结果显示:40个茄科雷尔氏菌株、2个BDB菌株共42个菌株可聚类为3个分支,其中第1分支包含了生化变种Ⅲ、Ⅳ和Ⅴ菌株,对应于Li等[7]的茄科雷尔氏菌区组1;第2分支包含了生化变种Ⅰ和Ⅱ菌株,对应于Li等[7]的茄科雷尔氏菌区组2;两个BDB菌株自成一个分支。本研究的21个菌株中,只有菌株HZ-1聚类在区组2,其余20个菌株聚类在区组 1(图 2)。
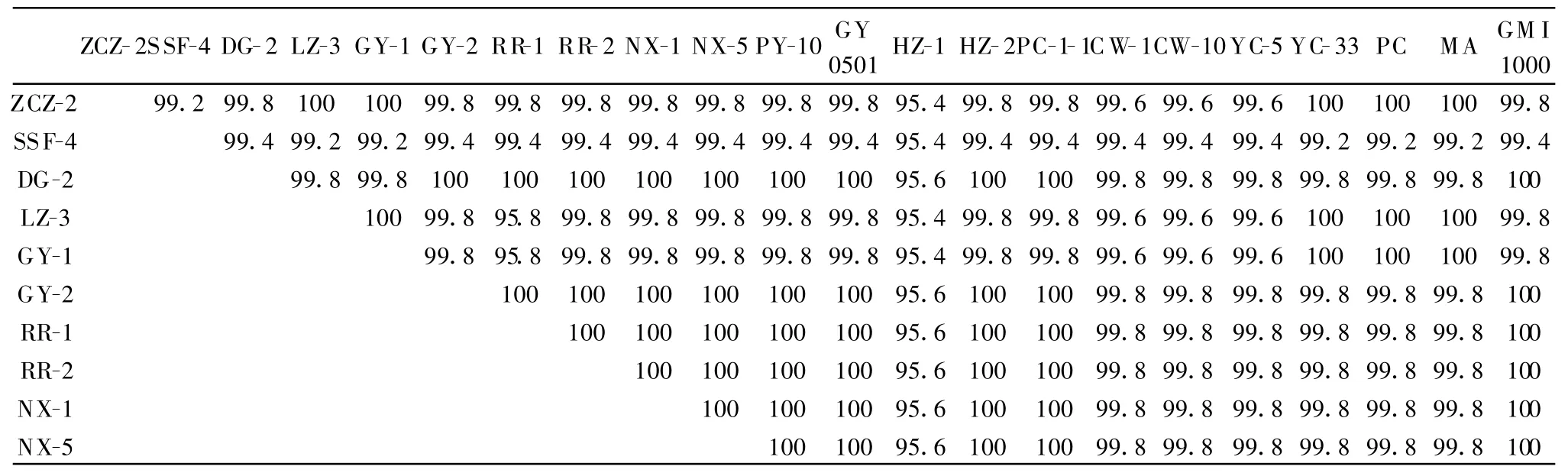
表3 21株茄科雷尔氏菌16S-23S rDNA ITS的序列相似性比较 %

续表3%

图2 茄科雷尔氏菌及相关菌株的16S-23Sr DNA ITS系统发育树
3 讨论
16S-23S rDNA ITS序列的进化速度是16S rDNA的10倍[19],该区间经常有序列的缺失和插入,造成不同种和同种不同菌株的16S-23S rDNA ITS在数目、长度和序列组成上的异质性。因此,在细菌分类、系统进化和分子特征等研究中,16S-23S rDNA ITS序列分析具有重要价值。
本研究分析了来源于12种寄主植物21株茄科雷尔氏菌的16S-23S rDNA ITS全长序列,除菌株HZ-1外,其余20个菌株的序列长均为503 bp,序列间相似性为99.2%~100%;而HZ-1菌株的序列长为498 bp,与其他 20个菌株间的序列相似性为95.4%~95.6%。所有菌株 ITS序列所包含的tRNAala和 tRNAile核苷酸序列完全一样,高度保守。因此,来源于不同寄主植物的茄科雷尔氏菌菌株间的16S-23S rDNA ITS序列也十分保守。
本研究21个菌株中,仅HZ-1菌株属生化变种Ⅱ,并与已登录GenBank的茄科雷尔氏菌生化变种Ⅰ、Ⅱ菌株聚类在区组2分支中;而其余20个菌株分别属生化变种Ⅲ和Ⅳ,且与已登录GenBank的茄科雷尔氏菌生化变种Ⅲ、Ⅳ和Ⅴ各菌株聚类在区组1分支中。由此可见,茄科雷尔氏菌的生化变种与区组间存在对应关系。这与对16S rDNA分析结果一致[16]。茄科雷尔氏菌种内以及一些关系密切的相关菌如 blood disease bacterium(BDB)的 16S rRNA基因的序列高度相似,有时也难以区分[8-9]。但利用ITS序列,BDB菌自成一个分支,可以将其与茄科雷尔氏菌分开。
对于分离自广东惠州马铃薯上的茄科雷尔氏菌菌株 HZ-1,其16S-23S rDNA ITS与 HZ-2及其他茄科雷尔氏菌菌株均存在较大差异,推测该菌株与其他20个菌株的起源不同。16S-23S rDNA ITS系统进化分析也支持这一推测,即HZ-1菌株属于茄科雷尔氏菌区组2,而其他20个菌株均属于茄科雷尔氏菌区组1。但该菌株的真正起源及与其他菌株间致病性差异等还有待于进一步研究。
[1] Hayward A C.Biology and epidemiology of bacterial wilt caused by Pseudomonas solanacearum[J].Annual Review Phytopathology,1991,29:65-87.
[2] Buddenhgen I,Sequeira L,Kelman A.Designation of races in Pseudomonassolanacearum[J].Phytopathology,1962,52:726.
[3] He L Y,Sequeira L,Kelman A.Characteristics of strains of Pseudomonassolanacearum[J].Plant Dis,1983,67:1357-1361.
[4] Pegg K,Moffett M.Host range of the ginger strain of Pseudomonas solanacear um in Queensland[J].Aust J Ex p Agric Anim Husb,1971,11:696-698.
[5] Hayward A C.Characteristics of Pseudomonassolanacearum[J].Journal of Applied Bacteriology,1964,27:265-277.
[6] Hayward A C,El-Nashaar H M,Nydegger U,et al.Variation in nitrate metabolism in biovars of Pseudomonas solanacear um[J].J Appl Bacteriol,1990,69:269-280.
[7] Li X,Dorsch M,Del Dot T,et al.Phylogenetic studies of the rRNA group II pseudomonads based on 16S r RNA gene sequences[J].Journal of Applied Bacteriology,1993,74:324-329.
[8] Cook D,Barlow E,Sequeira L.Genetic diversity of Pseud omonas solanacear um:detection of restriction fragment length polymorphisms that specify virulence and the hypersensitive response[J].Molecular Plant-Microbe Interactions,1989,2:113-121.
[9] Cook D,Barlow E,Sequeira L.Genetic diversity of Pseud omonas solanacear um:detection of restriction fragment length polymorphisms that specify virulence and the hypersensitive response[J].Molecular Plant-Microbe Interactions,1989,2:113-121.
[10]Taghavi M,Hayward A C,Lindsay I et al.Analysis of the phylogenetic relationships of strains of Burkholderia solanacearum,Pseudomonas syzy gii,and the blood disease bacterium of banana based on 16S r RNA gene sequence[J].International Journal of Systematic Bacteriology,1996,46:10-15
[11]Poussier S,Trigalet-Demery D,Vandewalle P,et al.Genetic diversity of Ralstonia solanacear um as assessed by PCR-RFLP of the hr p gene region,AFLPand 16S r RNA sequence analysis and identification of an African subdivision[J].Microbiology,2000,146:1679-1692.
[12]何自福,虞皓,罗方芳.广东茄科青枯菌致病力分化及其 DNA多态性分析[J].植物病理学报,2003,33(5):415-420.
[13]陈永芳,何礼远,等.我国植物青枯菌菌株的遗传多样性和组群划分[J].植物病理学报,2003,33(6):503-508.
[14]佘小漫,何自福,虞皓,等.广东茄科雷尔氏菌16S r DNA序列分析[J].华南农业大学学报,2009,30(4):24-28.
[15]Pastrik K H,Elphinstone J G,Pukall R.Sequence analysis and detection of Ralstonia solanacear um by multiplex PCR amplification of 16S-23S ribosomal intergenic spacer region with internal positive control[J].European Journal of Plant Pathology,2002,108:831-842
[16]佘小漫,何自福,李华平,等.广东茄科雷尔氏菌菌株生化变种的测定[G]∥中国植物病理学会2008年学术年会论文集.北京:中国农业科学技术出版社,2008:337-339.
[17]Boucher C A,Van Gijsegem F,Barberis P A,et al.Pseud omonas solanacear um genes controlling both pathogenicity on tomato and hy persensitivity on tobacco are clustered[J].Journal Bacteriology,1987,169:5626-5632.
[18] Li Xiang,De Boer SH.Selection of polymerase chain reaction primers from an RNA intergenic spacer region for specific detection of Clavibacter michiganensis subsp.sepedonicus[J].Phytopathology,1995,85:837-842.
[19]郑雪松,杨虹,李道棠,等.基因间隔序列(ITS)在细菌分类鉴定和种群分析中的应用[J].应用与环境生物学报,2003,9(6):678-684.
