一种基于有监督奇异值分解和随机森林的卵巢癌磷脂代谢物特征提取方法
2010-11-27来海锋厉力华SUTPHENRebecca
来海锋 韩 斌 厉力华 陈 岩 SUTPHEN Rebecca 祝 磊 代 琦
1(杭州电子科技大学生物医学工程与仪器研究所,杭州310018)
2(南佛罗里达大学H.Lee Moffit癌症研究中心,坦帕 FL 33612,美国)
引言
卵巢癌是一种常见的妇科肿瘤,发病率仅次于宫颈癌和子宫体癌,列居第三位,但因卵巢癌致死者,却占各类妇科肿瘤的首位,对妇女生命造成严重威胁。据统计,我国每年新发卵巢癌病人数为19.2万,但死亡人数却高达11.4万;美国每年发病人数为 2.2万,死亡达到 1.5万[1]。
肿瘤生物标志物是指在组织、血浆或其它体液中能够检测到的有关生化物质,这些物质达到一定水平时就能揭示某种肿瘤的存在,因此对肿瘤的诊断、分类、预后判断以及治疗提供指导。已有很多研究以基因谱或质谱数据为基础来提取卵巢癌等肿瘤标志物[2-3],最近研究者尝试开展了利用磷脂代谢物数据提取肿瘤标志物的工作,磷脂代谢物标志物从血液中提取,具有提取方便,检测快速,价格低廉等优点。在本研究中,针对合作方美国H.Lee Moffitt癌症研究中心提供的卵巢癌磷脂代谢物数据,研究利用磷脂代谢物数据主要解决以下两个方面问题:一为选取的磷脂代谢物特征标志物与样本采自于血浆或者血清是否有关;二为选取磷脂代谢物特征标志物用于卵巢癌诊断。
根据特征选择过程与分类器设计的关系,常用的特征选择算法一般可分为过滤法(filter)和缠绕法(wrapper)[4-5]。过滤法是一种基于判断准则的特征选择方法,如秩、信息增益等,他们独立于分类器;缠绕法是将分类器与特征选择结合使用,以最大分类率为引导的特征选择算法。在分类器的选择上,可以分为无监督分类和有监督分类。无监督的样本聚类方法通过样本的相似性度量,能够发现肿瘤样本集的结构特征,即相似的样本自然聚成一类[6],像自组织映射(self organizing maps,SOM)[7]和均值聚类(means clustering)[8]等;有监督分类主要有 k-近邻法(nearest neighbor,KNN)[9]、决策树(decision tree)[10,11]、支 持 向 量 机 (support vector machines,SVM)[12-13]、人 工 神 经 网 络 (artificial neural network,ANN)[14]等经典通用的分类方法,其优点是能够根据已知的样本类别信息进行学习,提取样本分类知识。有监督分类相比于无监督分类虽具有一定的优势,但也存在着一些不足之处,比如容易产生过拟合,当数据集中存在大量噪声时分类性能大大下降等。
针对以上存在的一些问题,本研究给出了一种Filter-Wrapper混合方法用于卵巢癌样本数据的特征标志物的提取。首先采用Filter方法从众多的生物标志物中粗选出一定数量的标志物,以大幅降低标志物搜索空间,然后采用Wrapper方法精选出满足目标条件的特征标志物集。在粗选过程中,使用有监督奇异值分解[15-16](supervised singular value decomposition,SSVD)方法对卵巢癌磷脂代谢物数据进行分析;在精选过程中,使用基于信息增益的随机森林决策方法,根据各肿瘤标志物的信息增益进行排序得到特征标志物[17-18],最后基于选取的特征标志物用SVM在测试数据上进行分类测试,并进行排列组合检验(Permutation Test)和寻找其所具有的生物学关联意义,以证实此方法用于特征标志物提取的可行性。
1 数据与方法
1.1 数据
本研究中所采用的磷脂代谢物数据来自于美国H.Lee Moffitt癌症研究中心,共有139个样本,分别采自于血清和血浆,其中卵巢癌组织样本70例(case),正常卵巢组织样本69例(control),其中70例case样本中分别有35例采自于血清和血浆,69例control样本中分别有35例和34例采自于血清和血浆。每个样本包括34个生物标志物,包括肿瘤抗原125(CA125)和磷脂代谢物,磷脂代谢物分别有缩醛磷脂类 (PPEs)、溶血磷脂胆碱 (LPCs)、溶血磷脂酸 (LPAs)、溶血磷脂酰肌醇 (LPIs)、溶血磷脂酰乙醇胺 (LPEs)、1-磷酸-鞘氨醇 (S1P),这些脂类信息分子包含一定的生物学关联信息,在人体各种机理反应中起着重要作用并显示人体生理状态,有研究显示这些信息分子与癌症之间存在着密切联系,应用这批磷脂代谢物数据,找出一组标志物用于卵巢癌诊断是本研究的一个重要目的。
1.2 特征选择方法
1.2.1 有监督奇异值分解(SSVD)
矩阵奇异值分解(SVD)是“对称矩阵正交相似于对称矩阵”的推广。设X是任意一个m×n阶实矩阵,X的第 i行是一个 n维向量 bi,它表示第 i个标志物在各样本上的反应值,X的第j列是一个m维向量aj,它表示第j个样本的表达谱。对X的奇异值分解的方程如公式(1)所示,通过SVD把X从m-markers × n-arrays 空 间 减 为 L-eigenarrays × L-eigenmarkers空间,其中 L=min{m,n}。

式中,S是一个L×L的对角矩阵,其中非零的值称为奇异值,因此,S=diag(s1,…,sL),其中当 1≤k≤r时 sk>0,而当 r+1≤k≤L 时,sk=0,其中 r为秩。U的每一列(uk)表示一个特征阵列(eigenarray),VT的每一行(vk)表示一个特征标志物(eigenmarker)。在这里,每一个eigenmarker唯一的和一个eigenarray对应,即uk唯一与vk应对,同时对应着作为特征表达水平的sk。
通过对X的奇异值分解得到的对角矩阵S中的sk是按从大到小排列的,传统的奇异值分解把第一分量作为区分样本的主要特征,但是无法得知这个主要特征是否是区分所关注的样本属性的特征。因此本研究采用一种有监督的奇异值分解(SSVD)来选取区分样本(case与 control)的特征标志物。作为SVD分解公式的一部分,aj可以表示为
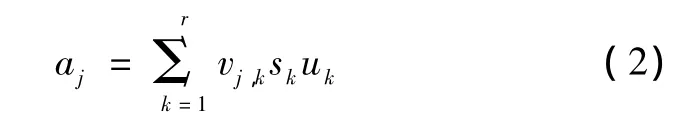
aj是uk是一个线性组合。为了真正找到区分case和control的特征向量 aj,本研究引入映射散点图和柯尔莫诺夫-斯米尔诺夫检验(Kolmogorov-Smirnov test)[23]构成 SSVD,其中 K-S 检验基于累计分布函数,用以检验两个经验分布是否不同或一个经验分布与另一个理想分布是否不同。pj,k=aj·uk表示样本aj映射到uk后的值,通过式(1)可以得到式(3),因此pjk可以用式(4)表示。

这样,如果 case和 control可以在散点图上被eigenarray分开,那么相应的 eigenmarker以及各个标志物在其中所具有的权重值也可以得到,选取前面的标志物作为粗选的结果。
1.2.2 基于信息增益的随机森林决策理论
随机森林是由Leo Breiman提出,是一类专门为决策树分类器设计的组合方法,通过自助法(bootstrap)重采样技术,从原训练集N中有放回地选取k个样本产生新的训练样本集,然后通过自助法产生m个决策树分类器,最后通过投票机制综合分量分类器的结果得到最终分类结果。在构建分量分类器时,未被选中的样本组成袋外数据集,通过袋外数据集进行性能测试。
随机森林中的每一棵决策树为一二叉树(二元分类),其生成遵循自顶向下的递归分裂原则,即从根节点开始依次对训练集进行划分;在二叉树中,根节点包含全部训练数据,按照节点不纯性度量最小原则,分裂为左节点和右节点,它们分别包含训练数据的一个子集,按照同样的规则节点继续分裂,直到满足分支停止规则而停止生长。不纯性度量有熵、Gini指数、分类误差等,本研究选用 Gini指数作为不纯性度量[24],其定义如下

式中p(i|t)表示给定节点t中属于类i的记录所占的比例,c表示类的个数。对于二类问题,任意结点的类分布都可以记作(p0,p1),其中 p1=1-p0。为了确定测试条件的效果,需要比较父结点(划分前)的不纯程度和子女结点(划分后)的不纯程度,它们的差越大,测试条件的效果就越好。增益G是一种可以用来确定划分效果的标准,其定义如下:

式中,I(·)是给定结点的不纯性度量(这里为Gini指数),N是父结点上的记录总数,k是属性值的个数,N(vj)是与子女结点vj相关联的记录个数。
用于构建分类器的标志物的重要性的一个主要表现为分类率,但对于决策树分类器,不光考虑分类率,还可以考虑标志物在构建决策树时所处结点的重要性。本研究中,基于信息增益的随机森林特征选择方法如图1所示。通过随机地从候选标志物中选取几个标志物构建成千上万个决策树来选取特征肿瘤标志物。具体地说,从d个候选标志物中选取m个标志物s次,即产生s个子集,而每一个子集随机按6∶4的比例构建训练集和测试集 t次。这样总共产生了st棵决策树。其中,s和t必须足够大,尤其是s,因为这样保证了每一个标志物有机会出现在不同的子集中,使选取的特征标志物更加准确。

图1 基于随机森林的特征选择框图Fig.1 Block diagram of feature selection with Random Forest
为了测定肿瘤标志物的相对重要性,首先引入加权分类率来评估决策树的性能。与普通分类率不同,加权分类率考虑了每类样本的大小,对于一个c类问题,定义 ni,j为第 i类样本分类成第 j类的数量,其中 i,j=1,2…,c,因此定义加权分类率为:

在本研究中c=2,因此可以简化为式(8)。

在决策时中,如果某个标志物作为一棵决策树的分裂结点的属性,那么如果包含的信息量越多,对决策树分类率的作用也越大,同样在这个结点上也会获得更多的信息增益。因此定义某个标志物的相对重要性指标为RIbk,具体定义为
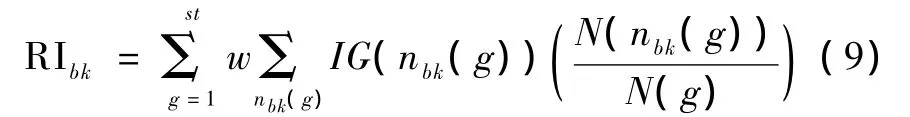
式中,st为整个处理过程中所构建的决策树的数量,w为某棵决策树的分类率,nbk(g)为第 g棵树上标志物bk作为分类属性的结点,IG(nbk(g))表示结点nbk(g)的信息增益,N(nbk(g))表示在结点 nbk(g)中的样本数量,N(g)表示第g棵树根结点的数量。
这样,应用随机森林思想计算每棵决策树的信息量得到每个标志物的RI值,即相对重要性。
1.3 具体实验方法与过程
1.3.1 辨识血清与血浆在特征选择中的区别
辨识血清与血浆在特征选择中的区别主要是通过SSVD方法,即通过样本在散点图上与生物特征的关系来辨识与诊断结果相关的特征向量。首先对数据进行预处理操作,第一将数据取对数,对数据进行压缩但又不失去原来数据间的意义;第二,对各样本进行归一化处理,具体操作是将各样本的表达数据分别减去其对应样本向量的均值。在预处理的基础上,经过SSVD得到样本散点图和K-S检验结果,通过对结果的分析来辨识血浆和血清对特征的选择是否有影响。
1.3.2 特征标志物的选取过程
作为本研究的重要部分,特征标志物选择的过程如图2所示。在对原始数据预处理的基础上,case和control分别按7:3的比例分为训练数据和测试数据,训练数据用来挑选特征标志物,测试数据用来对挑选出来的特征标志物进行测试。
首先使用SSVD方法进行粗选。通过SSVD找到能够辨识case和control的特征向量,按照一一对应的关系,对所有标志物按其权重值进行排序,最后选取其中的前N个标志物作为粗选的结果。
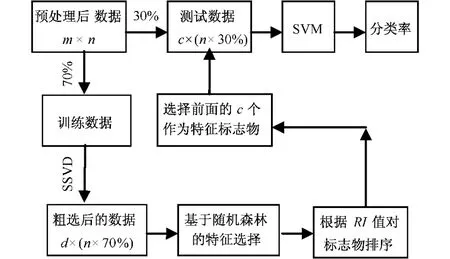
图2 特征标志物选择实验框图Fig.2 The experiment diagram of feature selection
其次,基于信息增益的随机森林特征选择过程如图2中所示,具体参数设置如下:d=20,s=1000,t=10,c=10。整个过程在Matlab平台上编程实现。
1.4 有效性验证方法
1.4.1 t-test
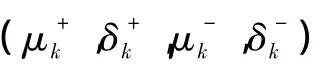

式中,n+,n-是各类样本的数量。根据每个标志物的T值,选出T值最高的几个标志物作为选取的特征标志物。
1.4.2 SVM-RFE
基于支持向量机的递归特征减少法 (support vector machine-recursive feature elimination,SVMRFE)是由Guyon等提出,是一种经典的有监督方法[21]。SVM-RFE 基于 SVM,对于训练样本,如果将其中的一个特征排除,则以SVM训练时的权向量W将发生变化,根据这一权向量的变化大小,可以确定相应特征的重要程度。如果每次去掉分类函数中关联权重值最小的肿瘤标志物,并将这一过程递归进行,从而得到各标志物的相对重要性。
1.4.3 Permutation Test
Permutation Test是根据所研究的问题构造一个检验统计量[22],并利用现有样本,按排列组合的原理,导出检验统计量的理论抽样分布;若难以导出确切的理论分布,则采用抽样模拟的方法估计其近似分布。然后求出从该分布中获得现有样本及更极端样本的概率(P值),并界定此概率值,做出推论。
本研究中,分类率差作为统计量应用于Permutation test中,目的是为了比较本研究方法与ttest和SVM-RFE有无显著性差异。在本实验中,将数据集重采样分成10组训练数据和测试数据后,分别使用以上三种方法在训练集上提取前10个特征标志物,然后用SVM分类器对提取的标志物在10组测试集上测试得到分类率,随机对得到的分类率平均分组100000次,计算两组之间的平均分类率差,与原有的分类率差进行比较,得到两组方法比较的显著性差异水平的P值。
1.4.4 变标号实验过程
应用变标号实验是为了验证所选取的卵巢癌磷脂代谢物特征标志物是否具有随机性,在实验中选取所选出的前3、6、9个特征标志物分别做置信水平为0.95的permutation test验证。具体做法是从总共84个训练样本中,随机挑选42个样本标记为case,剩余42个标记作为 control,在训练集上做相似处理,共进行100000次实验,得到分类率的概率分布图。
2 结果
2.1 血清与血浆在特征选择中的区别
在对数据预处理的基础上,通过SSVD得到样本散点图如图3所示。
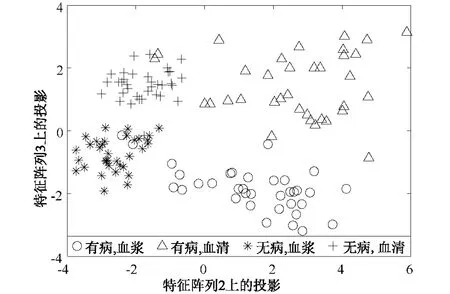
图3 样本通过SSVD分析后所得到的散点图Fig.3 The scatter plot of samples after SSVD
从图中可以看到除了个别点外,case样本集(圆和三角)和control样本集(加号和星号)分别沿某一坐标(样本“特征向量”)聚集在一起,而 case样本集和control样本集又沿着这一“特征向量”分开,在本研究中,这一特征向量为 eigenarray-2。已知分别团聚而又彼此分开的点分别表征两种卵巢癌诊断结果,因此第二个特征向量表征了与卵巢癌诊断结果相关的生化特征。在图中还可以看到不管样本采自于血浆还是血清,case与control都能被eigenarray-2很好的分开。K-S检验也同样印证了这点,当设置信水平为0.95时,K-S检验得到 P值小于0.05,接受零假设即两组样本服从不同分布,也就是样本集case和control在这一特征向量上服从不同的分布。从图3和K-S检验的结果可以得出在辨识的特征向量上,不同的样本服从不同分布,相同的样本聚类,说明这一特征向量表征了样本的类别信息,这也充分说明样本的采集不管是血浆还是血清,对于样本的所属类别没有影响,对于病人的诊断也没有影响。
2.2 特征标志物的选取结果
在使用SSVD方法进行粗选的过程中,实验一的结果表明case和control能够很好的被eigenarray-2分开,最后选取权重值最高的前20个作为粗选的结果。
在基于信息增益的随机森林特征选择过程中,得到前 10个标志物为 CA125、PPE5、PPE8、LPC0、LPE4、LPA2、PPE6、PPE7、PPE4、LPE5。
2.3 变标号实验验证
应用变标号实验得到分类率概率分布见图4。

图4 前3、6、9个特征标志物的 Permutation testFig.4 The permutation test of first 3,6,9 biomarkers
从图中可以看到,在变标号实验中所得到的分类率与原分类率相比较所得到的显著性水平P值≪0.05,这表明所选取的卵巢癌磷脂代谢物特征标志物具有显著的对卵巢癌的分类作用。
2.4 分类率比较
将本方法所选出的特征标志物与 t-test和SVMRFE所选出的特征标志物进行比较,通过SVM对测试数据进行测试,观察各方法的分类精度,其结果如图5所示。

图5 随机森林,t-test,SVM-RFE所选择的标志物在SVM分类率上的比较。(a)原始训练数据中包含 CA125,但SVM-RFE方法中CA125没有入选前10个标志物,而另两种方法将其包含其中;(b)原始训练数据中不包含CA125Fig.5 The classification rate comparison of Random Forest.t-test and SVMRFE on SVM.(a)the original training data include CA125,random forest and t-test selected CA125 as one of the first ten biomarkers,but SVMRFE excluded it;(b)the original training data does not include CA125
从图5中可以看到,基于信息增益的随机森林特征选择方法在分类率上来说要优于 t-test和SVMRFE,特别是SVMRFE。(a)中SVMRFE的分类率之所以比较低是因为其选出的前10个标志物中不包括CA125,而其他两种方法包括了 CA125,(b)的结果进一步证实了这个原因,这反过来说明CA125在样本分类中具有的重要作用,也印证了其本身作为肿瘤标志物所具有的作用。从图中也可以看到当选取相同数量的特征标志物时,本方法所选出的特征标志物包含了更多的分类信息。
2.5 分类率差异显著性比较
在定量比较本研究方法与T-test和SVMRFE有无显著性差异的实验中,通过Permutation Test得到本研究方法对 t-test的 P值为0.282,表明本研究方法好于t-test但是不显著;而在对 SVMRFE的比较中,P值远远小于0.05,表明本方法对 SVMRFE方法具有明显优势,这与图5(a)中的结果吻合。
3 讨论
3.1 生物学关联意义解释
特征提取不仅要求提取的特征能够获得较高的分类精度,同时提取的特征基因应该具有生物学关联。本研究方法所提取的特征标志物为CA125、PPE5、PPE8、LPC0、LPE4、LPA2、PPE6、PPE7、PPE4、LPE5。
通过查阅相关生物学资料,发现本方法所提取的特征标志物与卵巢癌的诊断有着很大的相关性。通过与已有文献的结果比较,也发现了所选取的特征标志物中包含了一些已被实验证实的与卵巢癌相关的重要生物标志物。
肿瘤抗原125(CA125)是存在于卵巢癌细胞表面的一种糖蛋白,是随访卵巢癌患者有价值的检测指标,正常组织或良性卵巢组织中不存在CA125,若发现 CA125 值升高,多见于卵巢癌组织中[25-27]。最近众多学者对其进行了广泛深入研究,Hogdall等的结果均表明,手术后CA125值的升降标志着肿瘤的复发与痊愈,且具有较高的判断符合率[28]。Geary等认为手术后第3个月的CA125含量测定最有意义,即术后3个月的CA125值仍大于35 U/mL的43例患者在9~l2个月进行二次探查术中均发现有癌组织的存在[29]。总之,CA125在卵巢癌的诊断、疗效监测和免疫治疗方面显示出可喜的应用前景。
溶血磷脂酸 (LPA)是新近发现的一种细胞间磷脂类信号分子,它通过G蛋白来耦联受体引起多种生物学效应,被称为多功能的“磷脂信使”。Xu等的研究发现,LPA可以作为诊断妇科恶性肿瘤的生物学指标,特别是早期诊断卵巢癌[30-31]。Shen等研究发现,卵巢癌患者血浆LPA水平显著升高,而其他肿瘤患者 LPA水平升高不明显[32]。另外,卵巢癌细胞能分泌LPA,促进血管内皮生长因子、尿激酶型纤溶酶原激活物、白介素-8、环氧合酶-2的产生,有利于卵巢癌细胞的浸润和转移。正常人血浆LPA水平很低,卵巢癌患者的腹腔积液及血浆LPA水平显著升高,而其他肿瘤患者的 LPA增高不明显[33]。总之,LPA水平对卵巢癌的诊断尤其是早期诊断和术后病情监测有重要价值。LPA2作为LPA中的一种,有着重要作用,Yun等发现LPA2是LPA的主要受体,细胞信号主要的通过 LPA2介导[34]。Huang等也同样提到了LPA2在卵巢癌诊断中的重要作用[32,35-36]。
Zhao等提出在卵巢癌病人中,可以检测到溶血磷脂胆碱 (LPC)值明显提高[37],Zhu等也同样提到LPC在卵巢癌诊断中起着重要的作用[38]。其他文章中也有对LPC在卵巢癌诊断中所起作用的描述[31,39]。
溶血磷脂酰乙醇胺 (LPE)虽然在卵巢癌诊断中的报道还不是很多,但是Kyoung等的研究表明,LPE在卵巢癌病人的不同时期里的反应有不同的表现,这充分说明LPE可能成为一种卵巢癌诊断的生物标记物[40]。
缩醛磷脂类 (PPEs)。缩醛磷脂类包括缩醛磷脂酰乙醇胺和缩醛磷脂酰胆碱等,研究表明其在肿瘤样本中的含量变化显著。Rachel等的研究表明胆碱代谢的增多和缩醛磷脂酰胆碱合成的减少可作为乳腺癌发生的代谢标记[41];Shan等的研究表明缩醛磷脂酰胆碱在卵巢癌血清样本中含量显著降低,而选取的部分 PPE对样本分类,敏感性高于80%,而特异性则达到 100%[42],充分说明 PPE可以作为一种潜在的生物标记物用于卵巢癌诊断。
3.2 方法总结
对生物数据分析的目的是为了找出具有生物相关性的标志物,而不仅仅是一个单纯的高分类率的组合。实际上已有大量的研究中出现分类率高达90%甚至100%的报道[43-44],而结果能应用于临床的很少,其中一个主要原因就是生物数据是高噪声数据,而且由于采样成本问题,样本量不大而出现“过拟合”,单纯提高分类率实际上是在拟合噪声,而且以分类率衡量生物相关性更多的是出于直觉和方便,并无证据表明分类率和生物相关性的关联;而差异检验方法的问题是差异有可能由非生物特性或者非感兴趣的生物特性造成。就其本质而言,SVM-RFE是一种完全利用分类率为监督的数据拟合方法,不能直接衡量标志物和诊断类别的关联,而且易产生过拟合;t-test是一种经典的无监督方法,通过检测标志物表达差异来选择标志物,而非生物原因有可能造成差异。
本研究中以“与肿瘤类别的关联”为指标来筛选标志物,然后用分类率做修正,关联度大的优先被选上,在图2中可以看到,本研究找到的特征向量与卵巢癌类别具有很强的相关性,在此特征向量上,不同的样本(case/control)服从不同分布,而相同的样本聚类,其表达情况完全符合肿瘤类别表达,而且这种按照与肿瘤类别相关性为准则的监督学习方法,排除了差异检验方法的弊端,同时由于本方法兼顾了分类率要求,从而保持了较高的分类率,在此意义上,笔者认为本方法比其他两种方法具有优势。在具体实验中,应用;t-test所提取的特征 标 志 物 为:CA125、PPE7、PPE8、PPE5、PPE6、PPE2、LPE4、LPC0、LPE5、PPE4,与本研究方法相比,提取的前几个标志物更多为PPE系列,且缺少了在卵巢癌诊断研究中具有重要意义的LPA2标志物[32,34-36];在 SVM-RFE 实验结果中缺少了在癌症诊断中具有重要作用的CA125。虽然三种方法在分类率上有一定的相似性,但本方法兼顾了与医学诊断信息的关联和分类的要求,辨识的特征向量保持了较高分类率的同时与医学诊断结果具有很强的关联,因而挑选的标志物对数据扰动更加不敏感,结果更稳定。最后决定选取前6个生物标志物作为特征标志物用于卵巢癌联合检测,这样既保证较高的分类率,又不缺乏生物学关联。
4 结论
本研究针对卵巢癌磷脂代谢物数据,提出了融合有监督奇异值分解和基于信息增益的随机森林特征选择方法研究肿瘤标志物特征提取问题,综合疾病分类率和标志物生物学关联意义挖掘出了CA125及5个磷脂代谢物特征标志物用于卵巢癌诊断。
首先应用有监督奇异值分解对生物标志物进行粗选;其次应用基于信息增益的随机森林特征选择方法对标志物进行精选,最后结合对测试样本的分类率和所选的标志物的生物学关联意义决定最后的特征标志物。有监督奇异值分解应用了包含诊断类别信息的特征向量以及K-S test以更好的选出区分类别的特征标志物;基于信息增益的随机森林方法与其他常用方法的对比在分类率不存在劣势,而选出的标志物更加具有生物学关联。因而本方法对研究寻找既具有较高疾病模式分类能力又有生物学关联的特征标志物具有重要意义,且方法简单可行,所提取的特征标志物对卵巢癌的临床诊断和医学研究起到有益的参考和借鉴作用。
[1]American CancerSociety. CancerFacts & Figures2009[R],2009.
[2]Tchagang AB,Tewfik AH,SkubitzAPN,etal. Groupbiomarkers identification in ovarian carcinoma [A]. In:Proceedings of 2007 IEEE International Conference on Acoustics,Speech and Signal Processing(ICASSP'07)[C].New York:IEEE,2007.1341-1344.
[3]Hibbs K,Skubitz KM,Pambuccian SE,et al.Differential gene expression in ovarian carcinoma-Identification of potential biomarkers[J].American Journal of Pathology,2004,165(2):393-141.
[4]Ke CH,Yang CH,Chuang LY et al.A Hybrid Filter/Wrapper Approach of Feature Selection for Gene Expression Data[A].In:Proceedings of IEEE International Conference on System,Man,and Cybernetic[C].New York:IEEE,2008.2663-2669.
[5]Zhang Liang,Zhang Fengming,Hu Yongfeng.A two-phase flight data feature selection method using both filter and wrapper[A].In:Feng WY,Gao F,eds.Proceedings of 8th ACIS International Conference on Software Engineering,Artificial Intelligence,Networking and Parallel/Distributed [C].Los Alamitos:IEEE Computer Society,2007.447-452.
[6]Mohsin S.Unsupervised Learning based feature points detection in ECG [A].In:SainzDelaMaza JMZ,Espi PLL,eds.Proceedings of 8th WSEAS International Conference on Signal,Speech and Image Processing[C].Athens:World Scientific and Engineering Acad and Society,2008.157-160.
[7]Essenreiter R,Karrenbach M,Treitel S.Identification and classification of multiple reflections with self-organizing maps[J].Geophysical Prospecting,2001,49(3):341– 352.
[8]LiGuangrong,Hu Xiaohua,Shen Xiajiong. A novel unsupervised feature selection method for bioinformatics data sets through feature clustering[A].In:Lin TY,Hu X,Liu Q,Shen X,et al.eds.IEEE International Conference on Granular Computing[C].New York:IEEE,2008.41-47.
[9]Li Yun,Lu Baoliang.Feature selection for identifying critical variables of principal components based on K-nearest neighbor rule [A].In:Qiu GP,Leung C,Xue XY et al,eds.Proceedings of 9th International Conference on Visual Information Systems[C].Berlin:Springer-Verlag,2007.193-204.
[10]Van UNT,Chung TC.An efficient decision tree construction for large datasets[A]. In:Proceedingsof 4th International Conference on Innovations in Information Technology[C].New York:IEEE,2007.502-506.
[11]Ge Guangtao,Wong GW. Classification ofpremalignant pancreatic cancer mass-spectrometry data using decision tree ensembles[J].Bmc Bioinformatics,2008,9(1):275 -287.
[12]Lu C,Van Gestel T,Suykens JAK.Preoperative prediction of malignancy of ovarian tumors using least squares support vector machines[J].Artificial Intelligence in Medicine,2003,28(3):281-306.
[13]Dash M,Liu H.Feature Selection for Classification [J].Intelligent Data Analysis,1997,1(3):131-156.
[14]Ledesma S,Cerda G,Aviña,G et al.Feature selection using artificial neural networks[A].In:Alexander G,Eduardo FM,eds.Proceedings of 7th Mexican International Conference on Artificial Intelligence[C].Berlin:Springer-Verlag,2008.351-357.
[15]Orly A,Patrick OB,David B.Processing and modeling genomewide expression data using singular value decomposition[A]In:Bittner ML,Chen Yidong,Dorsel AN,eds.Microarrays:Optical Technologies and Informatics[C].Bellingham:SPIE,2001,171-186.
[16]WallM,Rechtsteiner A,Rocha LM. Singular value decomposition and principal component analysis[A]. In:Daniel PB,Werner D,Martin G,et al,eds.A practical approach to microarray data analysis[M].New York:Springer US,2003.91-109.
[17]Draminski M,Rada-Iglesias A,Enroth S,et al.monte carlo feature selection for supervised classification [J].Bioinformatics,2008,24(1):110-117.
[18]Han Bin,Zhu Lei,Yanchen,etal. An across factor normalization based SVD approach to analysis of gene expression profiles for uncovering biomarkers in ovarian carcinoma chemotherapy responses [A]. In:Proceedings of2nd InternationalConference on Bioinformatics and Biomedical Engineering(iCBBE 2008)[C].Piscataway:IEEE,2008.334-337.
[19]Li ST,Liao C,James K.Gene Feature Extraction using T-Test Statistics and Kernel Partial Least Squares[A].In:King I,Wang J,Chan L,et al,eds.Proceedings of International Conference on Neural Information Processing[C].Berlin:Springer-Verlag,2006.11 - 20.
[20]Ding Yuanyuan,Wilkins D.Improving the performance of SVMRFE to select genes in microarray data[A].In:Jonathan DW,Stephen WH,Yuriy G,et al,eds.Proceedings of 3rd Annual MidSouth ComputationalBiology and BioinformaticsSociety(MCBIOS)Conference[C].London::BMC Bioinformatics,2006.S12.
[21]Guyon I,Weston J,Barnhill S,et al.Gene selection for cancer classification using support vector machines [J]. Machine Learning,2002,46(1-3):389–422.
[22]荀鹏程,赵杨,易洪刚,等.Permutation Test在假设检验中的应用[J].数理统计与管理,2006,25(5):616-621.
[23]Kar C,Mohanty AR.Application of K-S test in ball bearing fault diagnosis[J].Journal of Sound and Vibration,2004,269(1–2):439–454.
[24]Tan PN,Steinbach M,Kumar V.Introduction to Data Mining[M].Reading:Addison-Wesley,2006.
[25]Bouanene H,Harrabi I,Ferchichi S,et al.Factors predictive of elevated serum CA125 levels in patients with epithelial ovarian cancer[J].Bull Cancer,2007,94(7):E18 - E22.
[26]Kobayashi H,Ooi H,Yamada,Y,et al.Serum CA125 level before the development of ovarian cancer[J].International Journal of Gynecology& Obstetrics,2007,99(2):95-99.
[27]SchollerN,Urban N. CA125 in ovarian cancer [J].Biomarkers in Medicine,2007,1(4):513-523.
[28]Hogdall E.Cancer antigen 125 and prognosis[J].Current Opinion in Obstetrics& Gynecology,2008,20(1),4-8.
[29]Geary M,Folery M,Lenehan P,et al.Recurrent ovarian carcinoma:diagnosis and second-line therapy [J].Ir Med J,1995,88(2):68 -70.
[30]Yang Kun,Zheng Danhua,Deng Xiaoli,et al.Lysophosphatidic acid activates telomerase in ovarian cancer cells through hypoxiainducible factor-1 alpha and the PI3K pathway [J].Journal of Cellular Biochemistry,2008,105(5):1194 -1201.
[31]Sutphen R,Xu Y,Wilbanks GD,et al.Lysophospholipids are potential biomarkers of ovarian cancer [J]. Cancer Epidemiology Biomarkers and Prevention,2004,13(7):1185-1191.
[32]Shen ZZ,Belinson J,Morton RE,et al.Phorbol 12-myristate 13-acetate stimulates lysophosphatidic acid secretion from ovarian and cervical cancer cells but not from breast or leukemia cells[J].Gynecol Oncol,1998,71(1):364–368.
[33]Shen Zhongzhou,Wu Minzhi,Elson P,et al.Fatty acid composition of lysophosphatidic acid and lysophosphatidylinositol in plasma from patients with ovarian cancer and other gynecological diseases[J].Gynecol Oncol,2001,83(1):25-30.
[34]Yun C.Chris,Sun Hong,Wang Dongsheng,et al.The LPA2 receptor mediates mitogenic signals in human colon cancer cells[J].Am J Physiol Cell Physiol,2005,289(1):C2 – 11.
[35]Huang Meichuan,Lee HY,Yeh CC,et al.Induction of protein growth factor systems in the ovaries of transgenic mice overexpressing human type 2 lysophosphatidic acid G proteincoupled receptor(LPA2)[J].Oncogene,2004,23(1):122–129.
[36]WangPing,Wu Xiaohua,Chen Wenxue,etal. The lysophosphatidic acid(LPA)receptors their expression and significance in epithelialovarian neoplasms [J]. Gynecol Oncol,2007,104(3):714– 20.
[37]Zhao Zhenwen,Xiao Yijin,Elson P,et al. Plasma lysophosphatidylcholine levels: Potential biomarkers for colorectal cancer[J].Journal of Clinical Oncology,2007,25(19):2696-2701.
[38]Zhu Kui, Baudhuin LM, Hong Guiying, et al.Sphingosylphosphorylcholine(SPC)and lysophosphatidylcholine(LPC)are ligands for GRP4 [J].The Journal of Biological Chemistry,2001,276:41325-41335.
[39]Zhao Zhenwen,Xiao Yijin,Elson P,et al. Plasma lysophosphatidylcholine levels: potential biomarkers for colorectal cancer[J].Journal of Clinical Oncology,2007,25(19):2696–2701.
[40]Park KS, Lee HY, Lee SY, et al.Lysophosphatidylethanolamine stimulates chemotactic migration and cellular invasion in SK-OV3 human ovarian cancer cells:involvement of pertussis toxin-sensitive G-protein coupled receptor[J].FEBS Letters,2007,581(23):4411 - 4416.
[41]Rachel KB,Dalia S,Dalia RS.Metabolic markers of breast cancer:enhanced choline metabolism and reduced choline-etherphospholipid synthesis[J].Cancer Research,2002,62(7):1966-1970.
[42]Shan L,Davis L,Hazen SL.Plasmalogens,a new class of biomarkers for ovarian cancer detection[A].In:David E,Bruns YM,Dennis L,et al,eds.Proceedings of 59th Annual Meeting of the American Association for Clinical Chemistry[C].Washington DC:Amer Assoc Clinical Chemistry,2007,53(6):A110-A110.
[43]Antonov AV,Tetko IV,Mader MT et al.Optimization models for cancer classification:extracting gene interaction information from microarray expression data[J].Bioinformatics,2004,20(5):644-652.
[44]Wang hongqiang,Wong HS,Huang Deshuang,et al.Extracting gene regulation information for cancer classification[J].Pattern Recognition,2007,40(12):3379-3392.
