DNA序列广义3-D耦合数及其应用
2010-11-07潘琳玉马弘吕龙君李春渤海大学数学系121000
潘琳玉 马弘 吕龙君 李春 渤海大学数学系 121000
DNA序列广义3-D耦合数及其应用
潘琳玉 马弘 吕龙君 李春 渤海大学数学系 121000
本文在Z-曲线基础之上,通过扩大化的节点间的邻接关系,构造DNA序列的广义的3-D耦合数,并将其应用到DNA序列的刻画与比较研究中,对13个物种的 -球蛋白基因第一个外显子序列的相似性分析证实了这种方法的有用性。
DNA Z-曲线; 广义3-D耦合数; 相似性分析
1 引言
在DNA序列的图形表示中,很多图形表示伴随着一定程度上的简并/退化(指图形的交叉或重叠),这种简并现象则可能会伴随某些信息的丢失,因而许多学者都致力于非简并的图形表示研究,并且取得了很好的结果。但退化的图形表示并非不可利用,关键在于能否借其退化的形式引入更多的数学工具,进而挖掘其潜在的价值。本文正是以其为桥梁,通过扩大化的节点间的邻接关系,构造DNA序列的广义3-D耦合数。并将其应用到13个物种的相似性分析中。
2 DNA序列广义3-D耦合数
(a)0Q是一个列向量,,其中n表示图中节点的个数,它小于或等于序列中氨基酸的个数。qj为节点i所包含的电荷总量或权值,它等于在i点处的所有氨基酸的电量之和。
(b)0.这个向量的分量表示绝对初始概率,即,这里。
(c) 在文献1Π被称为一阶随机电子转移矩阵,它是一个n阶方阵,其中元素1pij表示为:步长k=1,从带电量为qi的节点ni移动到带电量为qi的节点ni的概率。

为截断函数。Gonzalez-diaz等人[5]曾指出,在1Π中,忽略距离较远的氨基酸之间的直接相互作用,并不是否认它们会以一种间接地方式相互作用。事实上,这种长程相互作用也是可能发生的(非禁止)。鉴于此,本文对aij做如下规定:如果ni与nj为同一节点或邻接两个节点则令aij=1,不然令aij=1/|i-j|。
我们知道,每一条DNA序列可分成三条“编码位置序列”,亦即相位序列,这三条序列分别由DNA序列中第1,4,7…;2, 5, 8…;3, 6, 9…位置上的碱基组成。按照文献[6],我们将每条相位序列转化为一条Z-曲线,进而,每一条相位序列对应一个点列。在向量0Q的构造中,本文并不是直接取核苷酸的电荷,而是将其推广到数学上的某种量,并从形式上称之为广义的“电荷”,这个所谓的“电荷”可以按照公式(4)确定:
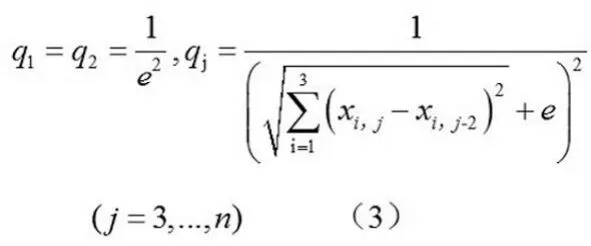
值得注意的是,Z曲线并不是DNA的真实3维空间结构,在这个3-D图中节点的个数小于或等于DNA序列中核苷酸碱基的实际个数。考虑到这点,我们用来代替ζk,其定义如下:
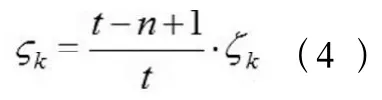
其中n为相位序列的Z曲线中的节点数,t为该相位序列中的碱基的实际个数。
综上所述,由任意一条DNA序列可得到三个不同的点列,同时每个点列对应4个耦合数。这样,一条DNA序列可以用一个12元的向量来刻画,这个向量的分量是相应的广义3-D耦合数。
3 结果及讨论
生物序列一旦以向量的形式表示出来,序列间的比较就可以转化为向量间的比较。通常认为,如果两个向量方向相近并且具有相近的量级,那么由这两个向量所代表的两条序列就是相似的[7]。因此,两个向量vi和vj之间的相似度可以由公式(5)计算得出:
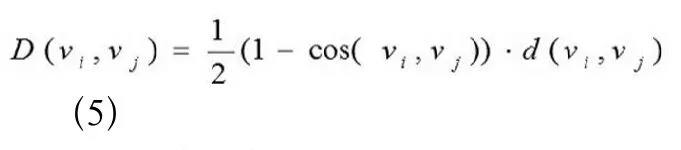
其中d(vi,vj)是向量vi和vj之间的欧式距离,cos(vi,vj)是向量vi和vj之间的夹角余弦值。显然,D(vi,vj)越小,两条序列越相似。
为了验证我们的方法的有用性,我们取n个不同物种的β-球蛋白基因的第一个外显子序列进行比较,它们被认为是非常保守的序列,即进化很慢的序列,能够较稳定的保持生物体的遗传性状。因此,许多相似性分析的文献都以此为例。
根据公式(5),我们计算出13个物种彼此间的距离,并用MEGA软件构造出13个物种之间的系统发育树(见图1)。
从图1可以看出,Gallus位于独立的一个分支,这表明gallus与其它物种间的相似程度都比较低,这与Gallus是非哺乳动物而其它物种都是哺乳动物相吻合。仔细观察哺乳动物所对应的子树,我们可以看到human, gorilla, and chimpanzee聚集在一起,同样有(Opossum,mouse,rat)和(goat,sheep,mouflon and bovine)也各自聚集在一起。本章所得结果与文献[7,8]中的结果基本一致。

图1 13个物种 -球蛋白基因第一个外显子序列的关系树Fig.1: the relationship tree of the first exon of -globin genes of 13 species
[1] 杨柳.蛋白质序列的图形表示及其应用.渤海大学硕士学位论文.2009.
[2] G.Aguero-Chapin, H.Gonzalez-Diaz, R.Molina, J.Varona-Santos, E.Uriarte, Y.Gonzalez-Diaz, FEBS Lett.580 (2006) 723_730.
[3] E.R.Collantes, W.J.Dunn, Amino acid chain descriptors for QSAR studies of peptides analogues, Journal of medicinal chemistry, 38, 1995, 2705-2713.
[4] Z.H.Lin, Y.Wu, Y.Wei, B.Ni, B.Zhu, L.Wang, A rapid method for quantitativeprediction of high affinity CTL epitopes∶ QSAR studies on peptides having affinity with the class I MHC molecular HLAA0201, Letters in peptide science,10, 2003, 15-23.
[5] H.Gonzalez-Diaz, R.Molina, E.Uriarte, Markov entropy backbone electrostatic descriptors for predicting proteins biological activity,Bioorganic& Medicinal chemistry letter, 14, 2004,4691-4695.
[6] R.Zhang, C.T.Zhang, Z curves, an intuitive tool for visualizing and analyzing DNA sequences, J.Biomol.Str.Dyn., 11, 1994, 767-782.
[7] M.Randic, M.Vracko, N.Lers, D.Plavsic, Analysis of similarity/dissimilarity of DNA sequences based on novel 2-D graphical representation, Chem.Phys.Lett., 371, 2003, 202-207.
[8] M.Randic, M.Vracko, N.Lers, D.Plavsic, Novel 2-D graphical representation of DNA sequences and their numerical characterization, Chem.Phys.Lett., 368, 2003, 1-6.
10.3969/j.issn.1001-8972.2010.11.012
潘琳玉 出生日期:1983年5月6日
性别:女 籍贯:吉林省公主岭市
渤海大学在读研究生 专业:应用数学
研究方向:生物信息学
