植物细胞DNA提取与PCR实验的设计及其关键参数的优选
2010-08-29闵运江
闵运江
(皖西学院化学与生命科学系,安徽六安 237012)
植物细胞DNA提取与PCR实验的设计及其关键参数的优选
闵运江
(皖西学院化学与生命科学系,安徽六安 237012)
以草本类型的蓼科5种植物为实验材料,研究了DNA提取方法。探讨了nrDNA ITS片段的PCR扩增方法,通过正交试验法,对该实验的关键步骤中PCR体系主要成分的最适含量进行了优化。为植物细胞DNA提取和PCR扩增这一现代生物学必不可少的实验教学和相关科学研究提供方法借鉴。
DNA提取;PCR扩增;实验设计;正交试验;草本植物
植物细胞DNA的提取与扩增实验是生物学等相关专业本科生必需掌握的实验技能之一[1](P271-284)[2](P53-55)[3-4],属于综合性、设计性实验。植物细胞DNA的提取与扩增技术又是以上专业学生进一步进入研究生学习阶段必备的而又较难掌握的实验技能之一。
DNA分子双螺旋结构的发现以及PCR(多聚酶链式反应,polymerase chain reaction)技术的建立,分子生物学得到了迅猛的发展,并已渗透到生物学的各门学科领域,已成为生物学各学科中发展速度最快,当今最热门的研究领域之一[5-12][13]。
DNA提取和PCR扩增操作是研究许多生物学问题必不可少的实验环节,而大多数学生和研究者常常在此环节伤透脑筋。随实验材料的种类、材料保存、运输条件的差异,以及实验操作过程的繁杂的实验条件的改变,其中任何一个参数设置或控制不好,结果就会导致实验的失败——无扩增产物或产物不理想,无法测序。
因此,本文以草本类型的蓼科5种植物为例,对其进行DNA提取、PCR扩增nrDNA的 ITS片段(核糖体基因的间隔区 ITS1和 ITS2,包含5.8S基因)等实验进行设计,实验中的关键步骤的参数,通过正交试验法进行探索,找到最佳实验条件,以期对植物分子生物学实验和研究提供方法借鉴。
1 材料与方法
1.1 实验材料(见表1)
1.2 实验设计
1.2.1 实验步骤
参照已发表的相关文献资料[14-15]以改良CTAB (Cetyltrimethyle Ammonium Bromide,十六烷基三甲基溴化铵)法,DNA提取与PCR扩增nrDNA ITS片段步骤如下:
(1)改良CTAB法提取DNA
称取干样品10~20mg→放入研钵中→加1.5角匙60~80目(AR)的石英砂→加适量液氮→研磨成粉末→将材料转入2ml离心管中→加4×CTAB液900μl→强烈振荡混匀 →置 65℃水浴保温 60~90min,每10min振荡一次→吸取上清液及部分样品浆液(除去石英砂)→转入 1.5ml离心管中 →加600μl氯仿:异戊醇 =24:1的溶液 →强烈振荡 →1200°r/min离心10min→取上清液→重复一次(即:再次加600μl24:1的氯仿:异戊醇→强烈振荡混匀→离心)→取上清液→加异丙醇450μl(上下移动30S轻摇)→在-20℃(或4℃)冰箱保存15~30min(材料不好可为4~6h或过夜)→1200°r/min离心5min→此时,应看到离心管底部有白色沉淀→将此沉淀用75%乙醇洗涤1~2次,倾去乙醇→在37℃恒温金属浴上风干→加50μlddH2O(无菌超纯水)溶解沉淀→加2μl的RNA酶(50mg/μl)→37℃水浴30min→加5~10μl的3M的NaAC和120μl无水乙醇,-20℃保存30min→1200°r/min离心10min→400μl75%乙醇洗涤1次→400μl无水乙醇洗涤1次→离心10S→37℃恒温金属浴上风干→加50μl的ddH2O溶解→电泳法检查DNA纯度→-20℃保存,备用。
至此即得到材料DNA(总DNA)模板。
(2)nrDNA ITS片段的扩增
配制好 PCR扩增体系,于 PCR扩增仪(Biorad DNA Engine,上海瑞亿仪器有限公司)上,扩增程序是:
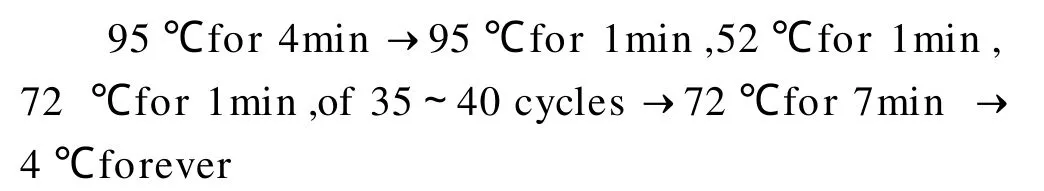
扩增结果用1%琼脂糖凝胶电泳,紫外检测器检查PCR产物。
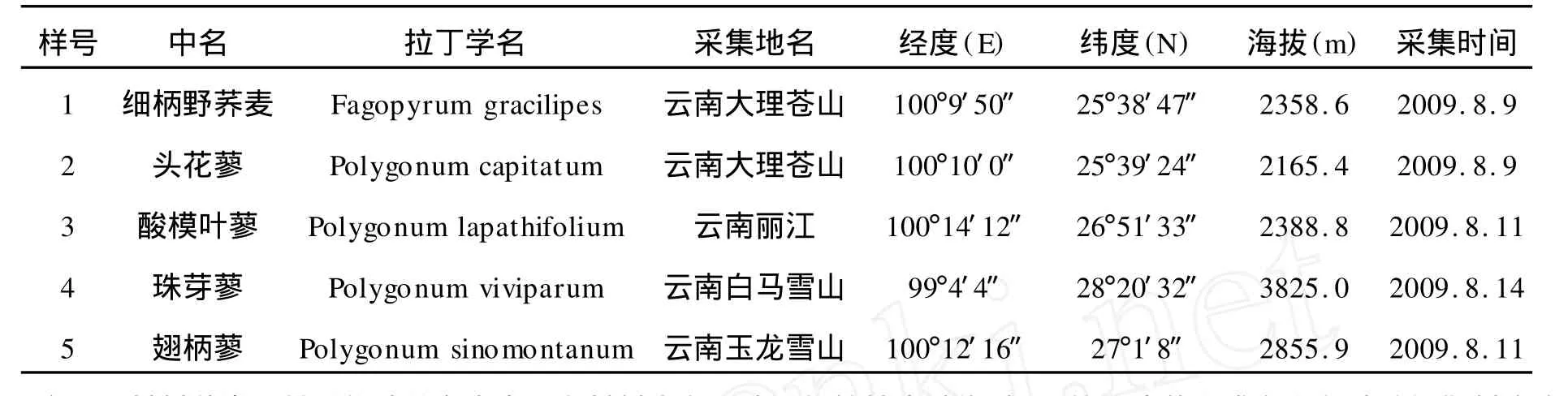
表1 实验材料采集地信息表
1.2.2 正交试验法优选PCR扩增条件
在 PCR扩增中,PCR扩增体系是否适宜,是PCR扩增能否成功的关键之关键,综合已发表的相关论文中其他学者所采用的条件,用DPS v7.05软件设计一个L16(54)正交试验,即5因素4水平16次试验的正交试验(表2、表3),优选出各成分的最佳条件。
正交试验设计如表3,按正交试验设计表配制好各体系,10×Buffer各 5μl,并以 dd H2O补足至50μl,按上述nrDNA ITS片段的扩增程序扩增,产物以1%琼脂糖凝胶电泳,紫外检查电泳有无亮带,根据亮带的亮度(与扩增产物中DNA片段含量呈正相关)判断PCR结果是否符合要求,每组试验结果中以每样品有清晰明亮的电泳带定量为1,无亮带为0,暗带定义为0.1~0.9,试验结果见表3和图1、2。
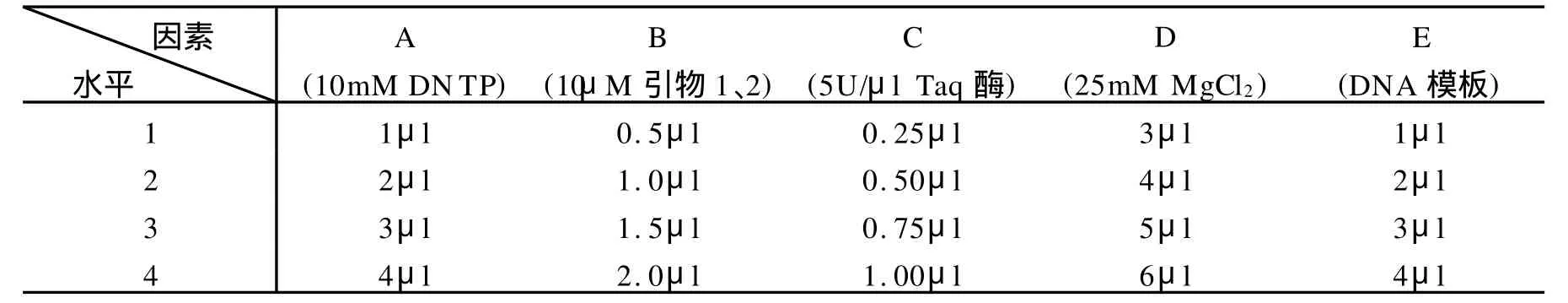
表2 L16(54)正交试验因素和水平表(50μl/体系)
2 结果与分析
2.1 DNA提取结果及其关键影响因素
按照以上DNA提取方法,只要样品材料干燥及时,低温保存,可以提取出植物细胞总DNA。
DNA提取的关键控制环节是防止DNA降解,离体的新鲜材料提取过程中,DNA极易降解。因此,采集的新鲜材料应立即用硅胶脱水干燥,并低温保存,提取DNA时用液氮研磨等提取过程中,保持低温等措施,均是防止DNA降解。
同时,采样和提取DNA过程中防止污染也是试验必须的控制因素之一。
2.2 PCR扩增体系优选结果及其分析
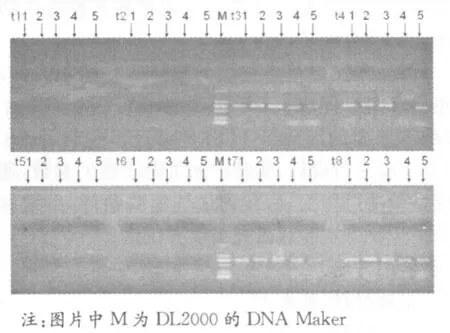
图1 ITS片段扩增正交试验t1-t8组结果
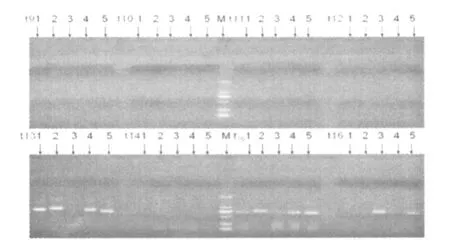
图2 ITS片段扩增正交试验t9-t16组结果
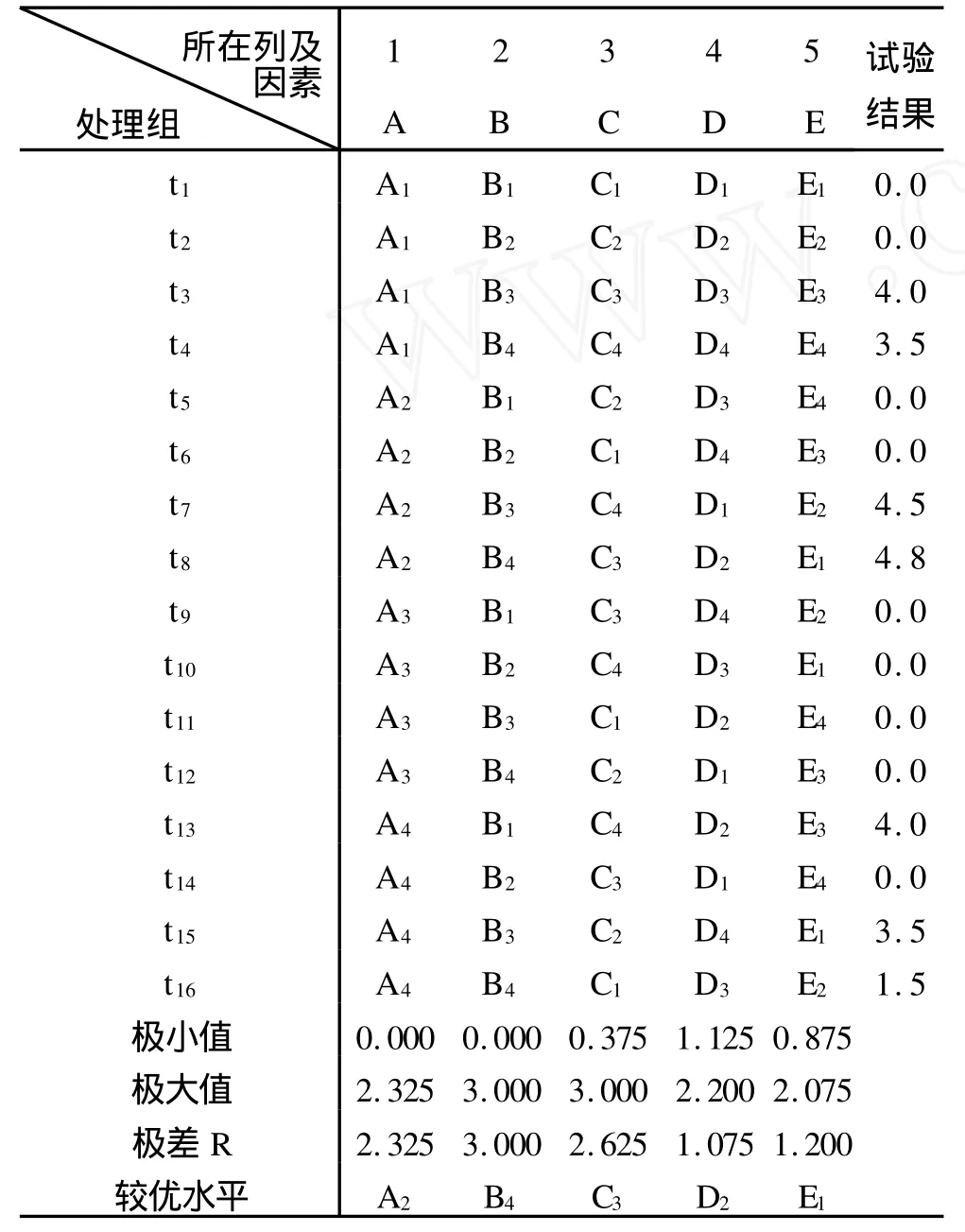
表3 L16(54)正交试验设计及电泳检查PCR结果及其方差分析结果
各PCR条件的正交试验结果,经1%琼脂糖凝胶电泳后,紫外检测,根据电泳带的清晰度,可直观判断各组每个样品的PCR产物量(见图1、2),经量化处理和方差分析结果见表3。
从图1、2和表3可见,处理组t8的5个样均有较清晰的电泳带,其他各处理组或不理想或无 PCR产物,说明t8组条件最佳,即A2B4C3D2E1组合的条件最佳,因此,每50μl PCR体系最佳配方是:10μM引物(1、2)各2.0μl、5U/μl的 Taq酶0.75μl、10mM的dNTP 2μl、DNA模板1μl、25mM的MgCl24μl、10 ×Buffer 5μl,并以dd H2O补足至50μl。
由表3的方差分析结果的极差大小可见,B>C >A>E>D,说明在本试验采用的各浓度范围内, PCR体系中,引物量对PCR结果影响最大,其次是Taq酶和dNTP的量。
3 讨论
植物DNA提取的方法很多,如十六烷基三甲基溴化铵(CTAB)法、十二烷基磺酸钠(SDS)法、PVP法、高盐法、尿素提取法和WizardTM clean-up system试剂盒等各种试剂盒法等等[16][17](P60-67),PCR扩增条件也随着材料和扩增片段不同而千差万别[18][19]。然而,其基本原理和大致过程是相同或相似的,尤其是同一类材料如草本植物,DNA提取方法、PCR体系组成和合理搭配及PCR程序基本可以通用。本文根据现有本科实验教学条件,摸索了适合于草本类型的植物细胞DNA提取方法——改良CTAB手工提取法的提取条件,并用正交试验找出了其PCR扩增的关键因素的最佳配比水平,可为相关学科的DNA提取与扩增实验教学和研究提供方法借鉴。
至于PCR扩增的另一关键因素——退火温度,本试验设计前已进行反复摸索,采用的是其最佳退火温度(52℃),该文中未作详述。
[1]杨安钢,毛积芳,药立波.生物化学与分子生物学实验技术[M].北京:高等教育出版社,2001.
[2]F.M.Ausubel,R.E.Kingston,J.G.Seidman,et al.精编分子生物学实验指导(第四版)[M].马学军,舒跃龙,译.北京:科学出版社,2005.
[3]丛峰松.生物化学实验[M].上海:上海交通大学出版社,2005.
[4]胡晓燕,张孟业.生物化学与分子生物学实验技术[M].济南:山东大学出版社,2005.
[5]A.Sanchez,T.M.Schuster,K.A.Kron.A Large-scale Phylogeny of Polygonaceae Based on Molecular Data[J]. International Journal of Plant Sciences,2009,170(8):1044 -1055.
[6]L.M.Dávalos,A.L.Porzecanski.Accounting for Molecular Stochasticity in Systematic Revisions:Species limits and Phylogeny of Paroaria[J].Molecular Phylogenetics and Evolution,2009,53:234-248.
[7]L.A.Johnson,L.M.Chan,T.L.Weese,et al.Nuclear and cpDNA Sequences Combined Provide Strong Inference of Higher Phylogenetic Relationships in the Phlox Family (Polemoniaceae)[J].Molecular Phylogenetics and Evolu-tion,2008,48:997-1012.
[8]M.L.Zhang,P.W.Fritsch,B.C.Cruz.Phylogeny of Caragana(Fabaceae)Based on DNA Sequence Data from rbcL, trnS-trnG,and ITS[J].Molecular Phylogenetics and Evolution,2009,50:547-559.
[9]郑小艳,曹家树,滕元文.DNA序列分析在植物分子系统学研究中的应用现状——以蔷薇科为例[J].园艺学报, 2009,36(12):1827-1836.
[10]严寒静,房志坚,余世孝.不同种源何首乌的 ITS序列分析及其亲缘关系研究[J].西北植物学报,2008,28(5):922 -927.
[11]陈生云,吴桂莉,张得钧,等.高山植物条纹狭蕊龙胆的分子亲缘地理学研究[J].植物分类学报,2008,46(4):573 -585.
[12]孟丽华,杨慧玲,吴桂丽,等.基于叶绿体DNA trnL-F序列研究肋果沙棘的谱系地理学[J].植物分类学报, 2008,46(1):32-40.
[13]A.Falchi,J.Paolini,J.M.Desjobert,et al.Phylogeography of Cistus creticus L.on Corsica and Sardinia inferred by the trnL-F and rpl32-trnL sequences of cpDNA[J]. MolecularPhylogenetics and Evolution,2009,52:538 -543.
[14]童大跃,伍新尧,陆惠玲,等.DNA提取方法的比较及改良[J].中山大学学报,2003,24(4):411-412.
[15]李玥莹,赵姝华,刘世强.高粱DNA提取纯化方法的比较及RAPD反应条件的建立与优化[J].杂粮作物,2001,21 (2):12-15.
[16]侯义龙,曹同,蔡丽娜,等.苔藓植物DNA提取方法研究[J].广西植物,2003,23(5):425-428.
[17]彭学贤.植物分子生物技术应用手册[M].北京:化学工业出版社,2006.
[18]C.Y.Mak,K.S.Cheung,P.Y.Yip,et al.Molecular Evidence for the Hybrid Origin of Bauhinia blakeana(Caesalpinioideae)[J].Journal of Integrative Plant Biology,2008, 50(1):111-118.
[19]J.L.Grimsby,D.Tsirelson,M.A.Gammon,et al.Genetic Diversity and Clonal VS.Sexual Reproduction in Fallopia spp.(Polygonaceae)[J].American Journal of Botany, 2007,94(6):957-964.
The Experiment Design and Its Key Parameters Optimization of Isolation and PCR Amplification DNA Fragment from Plant Cell
MIN Yun-jiang
(Department of Chemistry and L if e Science,West A nhui University,L u’an237012,China)
The experiments had been designed which plant cell DNA isolation and its nrDNA ITS fragment PCR amplification,with 5 species herbaceous materials of Polygonaceae.The key step of the experiment the main components of the PCR system optimum concentration had been optimized by the orthogonal test method.The results of this experiment will provide an example to our experimental teaching and related research.
DNA isolation;PCR amplification;experiment design;the orthogonal test method;herbaceous
Q94-33
A
1009-9735(2010)02-0089-04
2010-03-18
安徽省教育厅2010年度高校省级科学研究重点项目(KJ2010A327)。
闵运江(1963-),男,安徽金寨人,硕士,在读博士,副教授,研究方向:植物分子生态。
