细菌人工染色体基因组文库的构建及应用
2010-04-25任斐
任斐
(河南科技学院,河南新乡 453003)
DNA克隆技术是分子生物学研究中重要的技术手段之一,特别是大片段DNA克隆是复杂基因组分析中的第一步.而用于克隆DNA片段的载体系统则一直是分子生物学中的重要工具.1983年,Murry[1]把酵母染色体的着丝点、自主复制序列和端粒子连接在一起,构建成了第一个人工染色体,称为酵母人工染色体(Yeast Artificial Chromosome,YAC).在此基础上,Burke[2]构建了第一个 YAC 载体,并以此来克隆人基因组中的大片段DNA分子,它可插入上千kb左右的外源DNA.YAC的插入片段比噬菌体、cosmid载体在细菌宿主中的插入片段至少达十倍以上,能够覆盖完整的基因组,但是其自身有难以克服的缺点:存在嵌合现象,克隆的稳定性差,插入片段存在重排和丢失现象,插入片段的分离和纯化困难,转化效率低[3].因此,一种大容量、并能独立稳定存在和遗传的人工染色体显得尤为重要.
1 细菌人工染色体载体的优点
细菌人工染色体(Bacterial Artificial Chromosome,BAC)的构建基础是大肠杆菌(E.Coli)的F因子,其至少应具备三种功能元件的类似组份:复制原点(origin of replication),着丝点(centromere)和端粒(telomere).BAC相对YAC而言,具有很多优势:①通过电击方法转化大肠杆菌比YAC转化酵母的转化率提高了10~100倍,降低了文库构建所需的DNA量,尤其适合于目的DNA资源有限的文库构建;② 杂交筛选文库对于细胞中存在的单拷贝BAC载体都是可行的;③YAC以线形分子存在,BAC载体则以环形超螺旋状态存在,使得后者在分离操作上相对容易,使用常规的碱裂解方法即可做到;④BAC载体在大肠杆菌宿主中稳定复制,一般保持低拷贝,减少了所携带DNA片段的重组,使外源DNA片段保持稳定;⑤YAC克隆中普遍存在20%~40%的嵌合现象是YAC文库的最大缺陷,这主要是自由连接和同源重组形成的,而BAC克隆由于大肠杆菌重组缺陷型菌株的获得和大肠杆菌只允许一个细胞接受一个外源DNA分子,几乎不存在嵌合现象.
2 细菌人工染色体文库的构建及鉴定
基因组文库是用重组DNA技术将某种生物细胞的核基因组DNA的全部片段随机地连接到克隆载体上,再转移至适当的宿主细胞中,通过细胞增殖而形成的各个片段的无性繁殖系的总集.通常基因组文库所包含的克隆数目多到可以覆盖某种生物基因组的数倍.BAC文库构建主要有以下几个步骤.
2.1 BAC载体的制备
BAC载体的好坏直接关系到文库中空白载体的比例和文库的质量.载体DNA的提取采取碱裂解法,用氯化铯—溴化乙锭梯度平衡离心法纯化闭环载体DNA[4].用限制性内切酶对载体DNA进行酶切消化,为减少空白载体的比例还应用脱磷酶对载体进行充分的脱磷处理[5],再用连接酶对脱磷后的载体DNA进行自连,最后回收完全脱磷的线性化载体.
2.2 高分子量核DNA的制备
一般是在液氮中将样品组织研磨成粉末,在整个研磨过程中均使组织浸在液氮中[6];也可以是从血液中提取白细胞[7].为防止核DNA降解,一般采用2种低熔点琼脂糖包埋核DNA的方法,一是将核DNA包埋在低熔点琼脂糖中形成栓塞(plugs),二是将核DNA包埋在低熔点琼脂糖和矿物油混合物中形成微粒(microbead).前者在制备方法、对核DNA的保护作用和操作上明显优于后者.之后对核DNA进行酶切,一般通过两次脉冲场电泳进行片段选择以获得大小比较集中的片段[8].凝胶中回收DNA片段有两种方法:琼脂糖酶消化和电洗脱.采用电洗脱得到的DNA比用β-琼脂糖酶I处理得到的更完整,对DNA分子粘性末端破坏程度小于琼脂糖酶消化方法,有效提高连接效率和插入片段的大小.但是电洗脱方法更加耗时,回收过程中DNA的损失也较多.
2.3 大片段核DNA与载体连接
大片段核DNA能否有效地连接到载体上主要取决于插入DNA片段与载体的比例,对于不同生物采用的比例不同,大多数BAC文库采用1∶5~1∶15摩尔比[9].连接之后将连接产物在ddH2O上透析脱盐可以去除其中的高盐成分,避免电击转化过程中的电击穿现象.然后在含PEG 8000的0.5×TE上透析浓缩处理,可以提高转化效率数倍.但是透析之后的连接产物应立刻进行转化,因为比起透析之前,透析后的连接产物更容易降解和积聚.
2.4 重组载体转化受体感受态细胞
对于转化大肠杆菌而言,电转化是目前使用最普遍的一种方法.电转化效率与插入片段的大小有关,插入片段越大,转化效率越低.此外,转化条件也直接影响转化效率、插入片段大小及假阳性克隆的含量.因此,在大规模转化前需进行转化预试验,以选择最佳的转化条件.
2.5 挑取阳性克隆构建文库
可通过lac Z的α互补所形成的蓝白菌落筛选阳性克隆,阳性克隆的分检有人工和全自动分子生物学工作台技术两种.
阳性克隆可以接种至无菌的含有氯霉素LB培养基的384孔板中,37℃培养16 h后,用带有384个塑料针头的复制器复制接种可以得到复制板.原板和复制板文库可以在-80℃中长期保存.
2.6 BAC文库的筛选
文库构建完成后,需要对文库进行评价,评价一个BAC文库质量高低的标准是文库中克隆的数量、插入片段大小、细胞器DNA的含量,及假阳性克隆的含量.
用与目的基因紧密连锁的单拷贝标记探针筛选BAC文库,检验构建的BAC文库是否具有很好的代表性和实用性
2.7 BAC克隆的质量鉴定
从文库中随机挑取一定量的BAC克隆,用碱裂解法提取BAC DNA,经限制性内切酶酶切后,用脉冲场凝胶电泳(PFGE)检查插入外源DNA片段的大小.文库的平均插入片段大小以及所包含的重组克隆数决定了从中筛选出某个基因的概率,可以通过Clarke L.和Carbon J.在1975年提出公式计算:
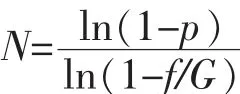
其中P是从建成的基因文库中可能选出某一基因的概率.f是该基因文库中每一克隆所含外源DNA片段的平均长度,可根据该生物基因组大小和所用载体可容纳的外源DNA片段的长度决定.G是该生物基因组的大小,基因组大小和DNA片段长度的单位可用道尔顿或碱基对表示.N是这一基因文库所应包含的克隆数目.
最高质量的BAC文库是那些插入片段大小为150 kb,覆盖基因组10倍,非重组克隆少于5%的文库[10].
2.8 BAC克隆的稳定性鉴定
挑选几个较大的BAC克隆(分子量分别为140、250 kb),分别接种在培养基上,连续继代培养后,分别提取第0代和第100代BAC克隆的质粒DNA,经限制性内切酶酶切后,脉冲电泳检查BAC克隆中外源DNA在继代培养中是否存在和发生突变[6,7].
2.9 检查BAC文库是否含有细胞器DNA的污染
核DNA文库混杂细胞器DNA的污染,不仅会降低实际库容量,还可能会误导染色体步移走向错误方向[11].采用脉冲电泳纯化法制备高纯度大分子量DNA,进行BAC文库的构建,可以从根本上避免细胞器DNA的污染.同时以线粒体基因为探针与BAC克隆进行菌落原位杂交,检验BAC克隆有无细胞器DNA的污染.
到目前为止,已经成功构建了很多种BAC文库.大致可以分为三种类型:第一种是整个核基因组的BAC 文库,第二种是部分基因组的 BAC 文库[12],第三种是构建 BAC 池[13].
3 BAC文库的应用
BAC文库许多对现代生物学研究都非常有用,比如分离完整的基因或基因簇,构建基因组的物理图谱,研究基因功能结构,定位克隆,长距离DNA测序和锚定,鉴定假设的顺式调节元件以及比较基因组学的研究等.
3.1 重要性状基因的克隆
对于已知序列的目的基因的筛选,通常采用2种方法:PCR筛选和分子杂交方法.
PCR方法是将各个单克隆混合组成各个混合池,Asakawa S.等提出了两步-4维(4 D)方法筛选[14].以96孔板为例,每个96孔板混合成1个混合物,然后每100个混合物排列成10×10的矩阵,每行混合成1个混合物,共10个组成1维(1 D),分别是1 D-1…1 D- 10;每列混合成1个,共10个组成2维(2 D),分别是2 D-1…2 D-10.每个96孔板由12×8个孔组成,每100个96孔板同一位置上的孔混合,这样将100个96孔板压缩成1个96孔板,然后按照前面的方法分别组成3 D和4 D.1 D~2 D混合或3 D~4 D混合组成1个超级池,含有9 600个克隆.第一步筛选出阳性超级池,第二步筛选1 D~4 D:筛选1 D、2 D可以知道哪块96孔板含有阳性克隆,筛选3 D、4 D可以知道这100个96孔板中哪个孔含有阳性克隆,二者结合就可以得知阳性克隆的具体位置.单个超级池中阳性克隆的数(n)存在的可能性(r)可以通过下面公式计算得出
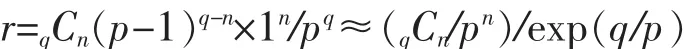
其中n<<p,q.p是文库覆盖基因组一倍所需的克隆数,q是这个超级池中的克隆数.以Asawaka S.等筛选的人基因组BAC文库的平均插入片段110 kb,覆盖基因组一倍需要34 000个克隆,一个超级池含有9 600个克隆,根据上述公式一个超级池中含有一个阳性克隆的可能性r=0.213,概率为86.6%.这种PCR筛选文库的方法非常简便,所需时间很短.
分子杂交分为膜杂交和Southern blot.膜杂交可以是高密度复制(high-density replica,HDR)膜或低密度膜,是用尼龙膜制作文库的复制品,然后用质粒、YACs或cDNAs作为探针进行HDR膜的菌落杂交(colony hybridization),最后通过自显影显示出阳性克隆.Southern blot是将每张HDR膜作为一组,用限制性内切酶消化此组的总BACDNA作为一张southern膜上的一条泳道,因此一张southern膜上可以检测很多张HDR膜.在筛选的开始使用Southern blot可以有效减少菌落杂交所需要的HDR膜的数量.
3.2 基因的图位克隆
基因的图位克隆(map-based cloning)又称为定位克隆(positional cloning),是用于分离尚不知道编码产物的目的基因的一种有效的方法.这种方法是根据目的基因在染色体上的位置进行的,基本包括以下环节:首先建立目的性状的分离群体,利用分子标记技术在目的基因两侧寻找与之紧密连锁的标记,利用遗传图谱和物理图谱将目的基因定位在染色体的特定位置上;其次构建大片段基因组文库(BAC或YAC),用与目的基因紧密连锁的分子标记为探针筛选文库,并用获得的阳性克隆构建目的基因区域的重叠群;找到与目的基因紧密连锁的分子标记和鉴定出分子标记所在的大片段克隆以后,以该克隆为起点用染色体步移或染色体登陆方法(chromosome landing)逐渐靠近目的基因,以该克隆的末端做为探针筛选基因组文库,鉴定和分离出邻近的基因组片段的克隆,再将这个克隆的远端末端做为探针重新筛选基因组文库,继续这一过程,直到获得具有目的基因两侧分子标记的大片段克隆或重叠群[15],随后通过压克隆获得含有目的基因的小片段克隆,最后通过遗传转化试验证实目的基因的功能.由此可以看出,基因图位克隆成功实施的关键因素之一是构建大片段基因组文库并筛选阳性克隆.BAC载体已在许多基因的图位克隆中做出了贡献.Song[16]等构建了水稻的BAC文库,并通过图位克隆获得了Xa21抗病基因.
3.3 基因组物理图谱的构建
BAC载体具有插入片段大、易分离等特点,在物理图谱的构建中发挥了重要作用.物理图谱所容纳的信息不仅包括基因的编码区也包括了内含子及基因的调控区.基因组物理图谱不仅是基因组测序的底物和基因图位克隆的桥梁,而且对研究基因的结构、功能、时空表达以及基因间的关系研究都极为重要.一些模式生物的全基因组物理图谱一旦得以构建,即可通过基因组序列分析找到大量的功能基因,还能发展新的分子标记以增强遗传图谱上分子标记的密度,加速基因图位克隆的进程.
3.4 基因组测序
基因组测序是发现、克隆、解码大范围基因的有力工具,是确定包括功能未知基因在内所有基因的完整序列和内、外显子结构所必需的.BAC文库性质稳定,保真度高,并且对普通的自动化DNA制备纯化程序相容,所以成为基因组分析中优先选择的克隆系统.BACs既可以通过亚克隆到质粒上进行测序,也可以直接作为原始底物进行末端测序及内部核苷酸分析.
3.5 在比较基因组学中的应用
比较基因组学(comparative genomics)是对来自不同的物种的已知基因和基因组结构进行比较,以了解基因的功能、表达机理和物种进化.比较基因组学研究的主要方法有生物信息学、DNA微阵列和BAC阵列(BAC arrays)3种,这些方法均与BAC有关.DNA测序量的突飞猛进改变了生物学研究方法.使得研究者可以不再依赖于“通过实验”来推断基因组在漫长的进化过程中是如何分化的.研究者可以直接观察基因组的变化,更清楚地了解基因组的组成和调节方式,了解是哪些变化造就了新物种.作为分析非编码顺式调控区是如何对基因组功能起作用的第一步,“targeted comparative sequencing”策略是对来自不同物种的直系同源的基因组区域的BACs进行测序.最终目的是得到不同基因组垂直同源区的序列信息并进行比对,可以推断出基因的非编码顺式调控元件的保守性.
4 结语
从第一个BAC载体构建至今,BAC由于其自身的一些优点已广泛应用于转基因和基因组研究,诸如基因组文库的构建、基因的图位克隆、基因组物理图谱的构建、基因组测序、基因的组织结构分析以及基因表达调控和基因转化等研究.BAC文库是克隆进BAC载体的大量外源基因插入片段的集合,并在大肠杆菌中复制.这些文库在各类生物的基因组计划中发挥了重要作用.随着更加完美的BAC载体的发展以及更多生物BAC文库的构建完成,必将对基因组研究产生巨大的影响.
[1]Murray N E, Murray,K.Manipulation of Restriction Targets in Phage Lambda to form Receptor Chromosomes for DNA Fragments [J].Nature, 1974, 251:476-481.
[2]Burke D T, Carle G F, Olson M V.Cloning of large segments of exogenous DNA into yeast by means of artificial chromosome vectors [J].Science, 1987, 236: 806-812.
[3]Shizuya H, Kouros-Mehr H.The development and applications of the bacterial artificial chromosome cloning system [J].Keio Journal of Medicine, 2001, 50(1): 26-30.
[4]Sambrook J, Fritsch E F, Maniatis T.Molecular cloning: a laboratorymanual (2nd Ed) [M].New York: Cold Spring Harbor Laboratory Press,1992.
[5]Kim U J,Birren BW,Slepak T, et al.Construction and characterization of a human bacterial artificial chromosome library [J].Genomics, 1996, 34:213-218.
[6]Zhang Y, Zhang X J, Chantel F, et al.Construction and Characterization of Two Bacterial Artificial Chromosome Libraries of Zhikong Scallop, Chlamys farreri Jones et Preston, and Identification of BAC Clones Containing the Genes Involved in Its Innate Immune System [J].Marine Biotechnol, 2008,10:358-365.
[7]Zhang X J, Zhang Y,Scheuring C, et al.Construction and Characterization of a Bacterial Artificial Chromosome (BAC)Library of Pacific White Shrimp, Litopenaeus vannamei[M].Marine Biotechnol, 2009.
[8]d′Alenc,on E, Piffanelli P, Volk of f A N, et al.A genome BAC library and a new BAC-GFP vector to study the holocentric pest Spodoptera frugiperda [J].Insect Biochemistry and Molecular Biology, 2004, 34(4):331-341.
[9]Frijters A C J,Vandamme M,Wang G L,et al.Construction of a bacterial artificial chromosome library containing large EcoRIand Hind IIIgenomic fragment of lettuce [J].Theor Appl Genet, 1997, 94(3):390-399.
[10]Miyake T, Amemiya C T.BAC libraries and comparative genomics of aquatic chordate species [J].Comparative Biochemistry and Physiology, Part C., 2004, 138(3):233-244.
[11]Zhang H B, Choi SD, Woo SS, et al.Construction and Characterization of Two Rice Bacterial Chromosome Libraries from the Parents of a Permanent Recombinant Inbred Mapping Population [J].Mol Breed, 1996, 2(1):11-24.
[12]Nam YW,Penmetsa R V,Endre G,et al.Construction of a bacterial artificial chromosome library of Medicago truncatula and identification of clones containing ethylene-response genes [J].Thero Appl Genet, 1999, 98: 638-646.
[13]Ma Z Y, Song W N, Sharp P J, et al.Non-gridded library: a new approach for BAC (bacterial artificial chromosome)exploitation in hexaploid wheat(Triticum aestivum) [J].Nucleic Acids Research, 2000, 28(24):1-5.
[14]Asakawa S, Abe I, Kudoh Y.et al.Human BAC library: construction and rapid screening [J].Gene, 1997, 191(1):69-79.
[15]Nakamura S, Asakawa S, Ohmido N, et al.Construction of an 800kb contig in the near centromeric region of the rice blast resistance gene Pi-ta2 using a highly representative rice BAC library [J].Mol Gen Genet,1997, 254(6):611-620.
[16]Song W Y, Wang G L, Chen L L, et al.A receptor kinase-like protein encoded by the rice disease resistance gene,Xa21 [J].Science, 1995, 270( 15): 1804-1806.
