鹿科动物茸角组织Osteopontin基因全长cDNA的克隆及表达分析
2010-03-07郝丽
郝 丽
(东北林业大学野生动物资源学院,哈尔滨 150040)
骨桥蛋白(Osteopontin,OPN)为一种分泌型的磷酸化糖蛋白,是成骨细胞的表型之一。最早由Franzen等研究骨基质成分时发现,因其介导骨组织细胞与骨基质的连接而得名。OPN在动物体中分布是有限的,如骨、肾、肌肉、膀胱及自然杀伤细胞等[1],其中骨和肾脏中含量最多。OPN主要在骨组织形成的早期产生。体外试验发现在骨基质矿化开始后,成骨细胞中OPN mRNA水平开始升高[2]。因此,OPN被认为是成骨细胞成熟分化的标志,是骨形成和重塑过程中非常重要的细胞因子。
鹿茸为雄鹿(驯鹿除外)未骨化密生茸毛的幼角,在中医临床占有重要地位,鹿茸入药距今已有两千多年的历史。鹿茸依靠其他动物器官无可比拟的生长速度、再生机理和神奇的药用价值吸引了众多科学家的高度关注[3-9],现已成为研究哺乳动物细胞增殖、生长与分化的理想材料,可作为模拟人和动物软骨内成骨过程的最佳实验模型。生长与骨化是鹿茸生长发育的两个方面。有学者将鹿茸角从脱盘到生长停止划分为生长期及骨化期。生长期内,生长占优势,骨化缓慢。骨化期内,鹿茸迅速骨化,沉积大量矿物质,阻碍鹿茸的生长。鹿茸骨化程度越高,矿物质含量就越高,有机成分比例下降。因此,鹿茸的骨化程度反映茸质的老嫩和成分含量的变化,决定着茸的等级。如能抑制或减弱鹿茸骨化而又不影响鹿茸的生长,则可全面提升鹿茸的产量,使养鹿业的效益大大提高。阐明生长与骨化的关系是实现这一目标的理论依据[10]。
目前GenBank中尚无鹿源OPN基因序列的任何记录。本研究从梅花鹿鹿茸尖端组织的全长cDNA文库中首次成功克隆了具有完整编码区的OPN基因的全长cDNA序列,并对其序列进行了生物信息学分析,通过RT-PCR技术研究了鹿茸尖端不同组织层中OPN基因的表达差异,为进一步研究OPN基因的功能及其在鹿茸骨化过程中的作用机制,指导鹿茸生产实践奠定了试验基础。
1 材料与方法
1.1 材料
选取人工驯养的健康4岁成年东北梅花鹿公鹿为实验动物(该年龄段的公鹿鹿茸生长发育相对稳定,以便缩小采样误差),在其生茸至二杠茸型时采集鹿茸顶端组织(该处为鹿茸生长发育中细胞增殖、生长和分化中心[11])为试验样品。取样时,将鹿麻醉,用手术刀迅速切取鹿茸顶端约4 cm组织(见图1),用酒精棉擦去茸皮表面污物,对照文献[12]处理赤鹿生长顶端的分层方法(见图2),切取皮肤层、间充质层、前软骨层和软骨层组织各25 mg,总共100 mg,于液氮保存。

图1 取材位置Fig.1 Position of the sample in deer antler
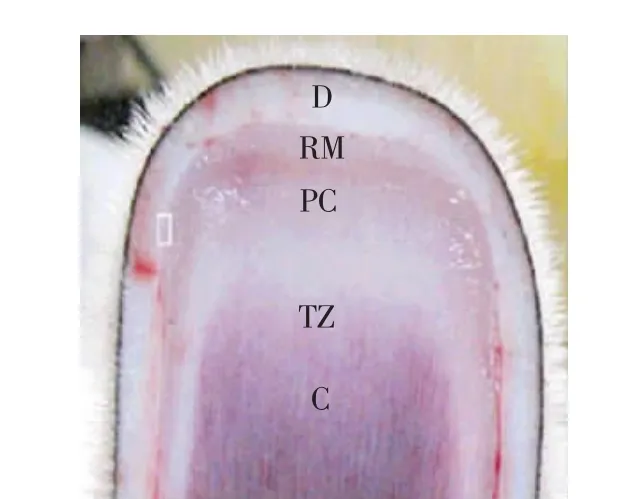
图2 组织分离位置Fig.2 Position of isolated tissue in deer antler
1.2 方法
1.2.1 OPN基因全长cDNA的获得
按试剂盒SV Total RNA Isolation System(Promega公司)操作说明提取总RNA。依照CreatorTMSmaRTTMcDNA文库构建试剂盒(Clontech公司)的说明,进行文库构建。在3730XL测序仪(Applied Biosystems,Foster City,CA,USA)上使用 M13 通用引物对鹿茸尖端组织原始文库中大量随机挑取的单菌落进行了5'端EST测序。利用Phrap软件对高质量的EST进行聚类和拼接,对拼接后的一致序列采用BLASTX和BLASTN程序,在GenBank非冗余蛋白质和核酸库中进行序列同源性比对及功能注释。挑取OPN基因对应的阳性菌落进行PCR鉴定,摇菌制备质粒,于测序仪上对阳性菌落质粒DNA进行双向测序。
1.2.2 OPN的生物信息学分析
采用ORF程序查找序列的开放阅读框,利用ExPASy服务器上的软件ProtParam和ProScal分别计算蛋白的分子量、理论等电点及分析蛋白的疏水性,SignalP3.0预测蛋白的信号肽,TMpred预测此蛋白的跨膜区,应用PSORTⅡ软件进行蛋白的亚细胞定位,BlastP软体进行蛋白保守区预测,Motif Scan软件进行基序搜索,使用软件DNAStar和GENEDOC进行氨基酸的多重序列比对和系统进化树的绘制。
1.2.3 OPN的实时荧光定量RT-PCR分析
分别提取鹿茸尖端不同组织层的总RNA,反转录合成cDNA。根据OPN基因的cDNA序列设计用于定量PCR的特异性引物(OPN-S:5'CATCT CAGAAGCAGACTTTC 3';OPN-A:3'-TCGTCATC ATCATCTAGGTC 5'),以18S rRNA为内参(18S-S:5'GGACTCTTAGCGGTGGATC 3';18S-A:3'ATC GTCAAGCGACGCTCAG 5'),对鹿茸尖端不同组织层的OPN基因表达量进行定量分析。在MJ Research公司的DNA Engine OpticonTM2实时荧光定量PCR仪上进行RT-PCR,PCR反应体系:第1链cDNA 3μL,SYBR PremixExTaqTM(2×)10μL,上、下游引物各1μL(10 pmol),去离子水5μL。PCR反应条件为:94℃预变性30 s,94℃12 s,57℃30 s,72℃30 s,45个循环,读板温度为81℃。
1.3 数据分析
联机运行软件为MJ Opticon MonitorTMAnalysis Software Version 3.1。通过普遍使用的 2-△△Ct法[13-14]来计算分析基因在不同组织中的相对表达量:每个反应做3次重复,取Ct值平均数;计算目标基因的平均Ct值与内标基因18S rRNA平均Ct值的差(△Ct值);通过每个测试样品的△Ct值减去对照样品的△Ct来确定△△Ct值。
2 结果与分析
2.1 OPN基因全长cDNA的获得
从梅花鹿茸尖端组织全长cDNA文库中获得的OPN基因的阳性菌落经PCR鉴定,得到一条约1400bp的DNA插入片段(见图3),进一步制备质粒进行双向测通,测序结果为1406bp,与预期片段大小一致。经进一步与GenBank数据库进行Blast比对分析,证实为OPN基因的全长cDNA序列(见图4)。

图3 梅花鹿OPN基因cDNA菌落PCR结果Fig.3 Results of colony PCR of OPN gene cDNA from Sika Deer
2.2 OPN的序列分析
ORF程序显示:OPN全长cDNA的5'非翻译区为105bp,3'非翻译区为461bp,开放读码框为840bp,编码279个氨基酸;利用NCBI的BlastP在蛋白保守区数据库(Conserved domain database,CDD)对OPN基因进行蛋白保守区预测表明,与此基因匹配的蛋白保守区有1个Osteopontin结构域,其匹配区段从第1个氨基酸到279个氨基酸(见图5);使用SignalP 3.0预测显示该基因氨基端第1~16位氨基酸具有典型信号肽特征;ProtParam在线软件预测OPN基因编码蛋白的相对分子质量为30.99 ku,理论等电点为4.40,不稳定系数54.42,属于不稳定蛋白。整个氨基酸组成中丝氨酸和谷氨酸残基均占很高比例,这就赋予了OPN具有高的酸性特征;利用软件TMpred进行跨膜区预测得知此蛋白为跨膜蛋白;ProtScale软件对OPN氨基酸序列进行疏水性分析发现,在1~25区域的氨基酸具有较大峰值,且这个区域位于跨膜区,可见跨膜区的氨基酸多数是疏水性的,N端的强疏水信号可能与该蛋白的跨膜运输有关;使用PSORTⅡ软件进行亚细胞定位分析表明,该蛋白为胞外蛋白,推测前16个氨基酸残基作为信号肽引导OPN编码蛋白定位于细胞外,在胞外发挥作用。
2.3 不同物种间OPN的同源性分析
将梅花鹿与GenBank中选取的9种动物的OPN氨基酸全序列进行BlastP,发现与欧洲牛、山羊的OPN具有很高相似性,分别为86%和85%;与野猪、人、马、金仓鼠及家犬的相似性较高,分别为65%、59%、57%、53%和50%;而与鹌鹑、原鸡的OPN序列之间的变异程度较大,相似性较低,分别为32%和31%。

图4 OPN基因的全长cDNA序列和推定的氨基酸序列Fig.4 Nucleotide and deduced amino acid sequences of OPN cDNA

图5 推导的OPN氨基酸保守区查找Fig.5 Search for the conserved domains in deduced sequence of amino acid of OPN
应用软件DNAStar和GENEDOC对9个物种的OPN氨基酸序列进行多重序列比对并绘制系统进化树(见图6、7)。结果表明,不同种属的OPN氨基酸表现出较高的同源性,说明OPN基因作为功能基因在进化中比较保守。树图显示,在该基因座位上梅花鹿与山羊和欧洲牛在进化关系上较近,而与鸟类(如鹌鹑、原鸡)亲缘关系较远。
2.4 OPN基因的实时荧光定量RT-PCR分析
RT-PCR结果显示,OPN的表达量从鹿茸皮肤层到前软骨层逐渐上升,到软骨层表达量又下调,且前软骨层和软骨层的表达量均高于间充质和皮肤层(见图 8)。

图6 梅花鹿与其他动物OPN氨基酸的多重序列比对Fig.6 Multiple alignment of amino acid sequences of the Sika Deer and other animal OPN

图7 动物OPN系统进化树Fig.7 Phylogenetic tree of animal OPN

图8 鹿茸尖端不同组织层中OPN基因的表达差异Fig.8 Expression of OPN genes in different tissues of velvet tip
3 讨论与结论
结合Motif Scan软件对功能位点的查找及不同物种间的多重序列比对结果,发现OPN氨基酸在26-29[SSEE]位酪蛋白激酶Ⅱ磷酸化位点、110-126位的富含天门冬氨酸的编码区及151-156位含RGD细胞黏附序列区域呈高度保守(见图6)。羧基端RGD(Arg-Glu-Asp)序列是OPN能够结合大量Ca2+以及与羟磷灰石具有高吸附作用的原因[15-16]。现已确定该序列通过与整合素家族中受体的识别,从而介导了细胞粘附和细胞迁移。OPN的系统进化树分为两大支,1支为哺乳动物,另1支为鸟类,其分析结果与物种的传统分类学地位十分一致,且符合进化关系。
在梅花鹿鹿茸尖端组织的ESTs中OPN基因属于高丰度表达基因,Apparao等[17]研究认为OPN基因的表达具有波动性。本研究也发现,在鹿茸尖端不同组织层中OPN表达水平的波动性变化。OPN有促进成骨细胞样细胞和成纤维细胞贴附于细胞外基质上的功能[18-19],产生该作用的机制是它的分子内部含有一特殊结构RGD序列,该序列是促进细胞黏附的蛋白质中的特有结构。另外,OPN作为由单核细胞衍生的破骨细胞的调节器,能够指导最初的破骨细胞识别并黏附到骨上,通过与αvβ3整合素相互作用刺激破骨细胞迁移,从而提高了骨的吸收[20]。组织钙化的一个重要事件就是蛋白的降解,由此,我们推测OPN可能通过介导破骨细胞与矿化组织的黏附,从而参与了鹿茸组织的矿化。
OPN是在骨代谢过程中起重要调节作用的细胞因子,本研究首次从梅花鹿鹿茸尖端组织cDNA文库中成功克隆了具有完整编码区的OPN基因的全长cDNA序列,并且由EST分析得知OPN基因在所构建的鹿茸尖端组织cDNA文库中为高丰度表达基因,提示它可能是鹿茸发育过程中的重要调节因子。研究鹿茸骨化机制不仅可以揭示茸质的老嫩和成分含量的变化,直接决定鹿茸最佳采摘时间、等级,同时对建立以鹿茸为骨发育模型具有重大意义。鹿茸的骨化机理非常复杂,深入研究其各种调控机制,不仅可以提高鹿茸的产量和质量,还有助于骨病的治疗,而且也为新药开发提供理论基础。
[1]Denhardt D T,Guo X.Osteopontin:a protein with diverse functions[J].FASEB J,1993,7(15):1475-1482.
[2]Owen T A,Aronow M,Shalhoub V,et al.Progressive development of the rat osteoblast phenotype in vitro:reciprocal relationships in expression of genes associated with osteoblast proliferation and differentiation during formation of the bone extracellularmatrix[J].J Cell Physiol,1990,143(3):420-430.
[3]Han M,Yang X,Taylor G,et al.Limb regeneration in higher vertebrates:Developing a roadmap[J].Anat Rec B New Anat,2005,287(1):14-24.
[4]Mescher A L.The cellular basis of limb regeneration in urodeles[J].Int J Dev Biol,1996,40(4):785-795.
[5]Brockes J P,Kumar A.Plasticity and reprogramming of differentiated cells in amphibian regeneration[J].Nature Rev Mol Cell Biol,2002(3):566-574.
[6]Brockes J P,Kumar A.Appendage regeneration in adult vertebrates and implications for regenerative medicine[J].Science,2005,310:1919-1923.
[7]Odelberg S J.Cellular plasticity in vertebrate regener-ation[J].Anat Rec B New Anat,2005,287(1):25-35.
[8]Li C,Waldrup K A,Corson I D,et al.Histogenesis of antlerogenic tissues cultivated in diffusion chambers in vivo in red deer(Cervus elaphus)[J].J Exp Zool,1995,272:345-355.
[9]Price J,Allen S.Exploring the mechanisms regulating regeneration of deer antlers[J].Philos Trans R Soc Lond B Biol Sci,2004,359:809-822.
[10]高志光.梅花鹿鹿茸生长与骨化关系的研究[J].特产研究,1999(3):51-53.
[11]赵世臻.中国养鹿大成[M].北京:中国农业出版社,2001.
[12]Li C,Suttie J M.Tissue collection methods for antler research[J].European Journal of Morphology,2003,41(1):23-30.
[13]Livak K J,Schmittgen T D.Analysis of relative gene expression data using real-time quantitative PCR and the 2-ΔΔCt tmethod[J].Methods,2001,25(4):402-408.
[14]Ingham D J,Beer S,Money S,et al.Quantitative real-time PCR assay for determining transgene copy number in transformed plants[J].Biotechniques,2001,31(1):132-140.
[15]Steitz S A,Speer M Y,McKee M D,et al.Osteopontin inhibits mineral deposition and promotes regression of ectopic calcification[J].Am J Pathol,2002,161(6):2035-2046.
[16]Ohri R,Tung E,Rajachar R,et al.Mitigation of ectopic calcification in osteopontin-deficient mice by exogenous osteopontin[J].Calcif Tissue Int,2005,76(4):307-315.
[17]Apparao K B,Illera M J,Beyler S A,et al.Regulated expression of osteopontin in the peri-implantation rabbit uterus[J].Biol Reprod,2003,68(5):1484-1490.
[18]Oldberg A,Franzen A,Heinegard D.Cloning and sequence analysis of rat bone sialoprotein(osteopontin)cDNA reveals an Arg-Gly-Asp cell-binding sequence[J].Prog Natl Acad Sci USA,1986,83(23):8819-8823.
[19]Somerman M J,Prince C W,Sauk J J,et al.Mechanism of fibroblast attachment to bone extracellularmatrix:role of a 44 kilodelton bone phosphoprotein[J].Bone Miner Res,1987,2(3):259-265.
[20]Uemura T,Liu Y K,Kuboki Y.Preliminary communication.MRNA expression of MT1-MMP,MMP-9,cathepsin K,and TRAP in highly enriched osteoclasts cultured on several matrix proteins and ivory surfaces[J].Biosci Biotechnol Biochem,2000,64(8):1771-1773.
