自然发酵东北酸菜中乳杆菌的分离与鉴定
2010-03-07张鲁冀孟祥晨
张鲁冀,孟祥晨
(东北农业大学乳品科学教育部重点实验室,哈尔滨 150030)
酸菜是一种中国北方传统的腌渍蔬菜制品,酸菜汁中富含丰富的乳酸菌[1-2],是巨大的乳酸菌资源库。目前人们从酸菜中分离出的乳杆菌主要有植物乳杆菌,短乳杆菌[3-5],而分离得到的菌株主要集中在优势菌群范围内。除了这些优势菌群外,酸菜中还包含许多其他菌群,因此酸菜成为分离乳酸菌,尤其是乳杆菌的良好材料。本试验是从酸菜中分离乳杆菌,并采用传统生理生化方法与分子生物学方法,对分离菌株进行鉴定,为多角度开发利用东北酸菜中的微生物资源,发展相应微生态制剂及深入研究其开发利用价值奠定基础。
1 材料与方法
1.1 材料
1.1.1 标准菌株及来源
嗜酸乳杆菌(Lactobacillus acidophilus)ATCC 4356,购自中科院微生物研究所菌种保藏中心。
1.1.2 自然发酵东北酸菜
15个自然发酵东北酸菜样品采集自中国哈尔滨地区,样品风味口感良好。
1.1.3 仪器设备
生化培养箱(上海佳胜实验设备有限公司);BCN-1360生物洁净工作台(北京东联哈尔滨仪器制造有限公司);光学显微镜(OLMPUS);GL-2M离心机(上海市离心机械研究所);HIRAYAma HVE-50型高压灭菌器(日本HIRAYAma公司);DK-98-11A电热恒温水浴锅(天津市泰斯特仪器有限公司);台式离心机(上海安亭科学仪器厂);微量进样器(美国Eppendorf公司);ABI 9700 PCR仪(美国Applied Biosystems公司);DYY-10C型电泳仪(北京市六一仪器厂);UVP凝胶成像系统(美国UVP公司)。
1.1.4 各种培养基与试剂
MRS培养基参照文献[6]的方法配制。磷酸盐缓冲液、0.9%生理盐水和革兰氏染色液按照文献[6-8]的方法配制。H2S产生试验、硝酸盐还原试验以及各种碳水化合物发酵试验试剂为浙江省杭州天和微生物试剂有限公司生产的细菌微量生化反应管。1 mol·L-1Tris-HCl(pH 8.0)、TE缓冲液、TAE缓冲液按照文献[9]配制。基因组DNA提取试剂盒、marker购自天根公司。
1.2 方法
1.2.1 自然发酵酸菜样品的采集及前处理
酸菜样品采集后装于无菌试管中,封好放入冰盒内保持低温,立刻运回实验室进行分离。
1.2.2 菌株的分离与纯化
取酸菜汁以3%的接种量接入液体MRS培养基中进行富集培养,37℃厌氧培养48 h;然后将富集培养的菌液稀释成10-1~10-77个梯度,分别涂布于含有0.75%CaCO3的MRS平板上,37℃厌氧培养48 h,在菌落数为30~300个之间的平板上挑选出周围形成透明圈的菌落,进行革兰氏染色后镜检,凡革兰氏阳性菌落,继续划线于MRS平板,直至确定为纯菌,纯化后的菌株储存在MRS斜面培养基中,放置4℃冰箱中保存备用。
1.2.3 分离菌株的生理生化试验
凡是革兰氏染色阳性、过氧化氢酶试验阴性的菌株初步确定为乳酸菌。在初步鉴定的基础上,接着进行硝酸盐还原试验、明胶液化试验、H2S产生试验[6,10-11]。
1.2.4 分离菌株的糖发酵试验
种的鉴定主要依据分离菌株对碳水化合物的发酵产酸结果,即通过对单糖、二糖、三糖、多糖以及糖苷和糖醇类等碳水化合物的利用情况来区分[6,10-11]。
1.2.5 分离菌株基因组DNA的提取
基因组DNA的提取采用细菌基因组DNA提取试剂盒进行提取,具体步骤参照说明书。
1.2.6 PCR扩增16S rDNA基因序列
PCR扩增的引物[12]分别为:

PCR反应体系[13-15]:10×PCR Buffer(Mg2+Plus),5μL;dNTP Mixture(2.5 mmol·L-1each),4μL;引物 P1,1μL;引物 P2,1μL;模板,2μL;Ta-KaRa Taq 酶(5 U·μL-1),0.5μL;ddH2O,36.5μL。
PCR反应条件:预变性94℃、5 min,变性94℃、1 min,退火58℃、1 min,延伸72℃、2 min,末端延伸72℃、10 min。循环数为30个。
1.2.7 16S rDNA基因测序与同源性分析
目的基因的PCR扩增产物由上海生物工程技术服务有限公司纯化后测序。将测序结果输入GenBank数据库,利用BLAST软件,与数据库中已发表基因序列进行序列比对[16],从而最终确定目的基因的同源性。
2 结果与分析
2.1 分离菌株的形态学特征
本试验从15份自然发酵东北酸菜样品中共分离得到革兰氏阳性、过氧化氢酶试验阴性的杆菌21株。分离菌株的菌体细胞形态为短杆或短粗杆状,多数单独,或成对存在。在MRS固体培养基上生长,其菌落主要特征为表面光滑、边缘整齐、中间突起或扁平的白色菌落。在MRS液体培养基中生长,菌株的培养特性各不相同。有的上部液体澄清,底部菌体形成较厚的圆形沉淀;有的上部液体浑浊,底部菌体形成较薄的圆形沉淀;有的底部菌体有向上贴壁生长趋势。
2.2 分离菌株的生理生化特征及糖发酵试验结果
分离菌株的生理生化特征及糖发酵试验结果见表1。反应结果与《伯杰氏细菌鉴定手册》(第八版)进行比较[10],得出酸菜汁中分离得到的21株菌株的H2O2酶试验、硝酸盐还原试验、H2S产生试验、明胶液化试验均为阴性,符合乳杆菌属的特征,所以初步判定为乳杆菌属。从糖发酵鉴定结果来看,标准菌株ATCC4356与嗜酸乳杆菌(Lactobacillus acidophilus)的性质完全相同;而L4、L14、L17、L21、L22、L33与植物乳杆菌(Lactobacillus plantarum)的性质相近。L3、L5、L6、L12、L18、L19、L28、L30与短乳杆菌(Lactobacillus brevis)的性质相近。L7、L9、L10、L27、L31、L32与罗伊氏乳杆菌(Lactobacillus reuteri)的性质相近。L20与米酒乳杆菌(Lactobacillus sakei)的性质相近。
表中做标记的糖发酵试验结果与文献[17]中给出的标准菌株糖发酵试验结果有差别,可能是由于菌株自身的某种特性或在培养过程中发生变异导致菌株的部分糖醇发酵结果发生改变,造成了单纯利用生理生化试验结果对菌株进行鉴定存在一定的困难。因此,为了对目的菌株进行准确的鉴定,采用16S rDNA序列同源性分析的方法进行进一步鉴定。

表1 菌株的生理生化特征及糖发酵试验结果Table1 Physiological-biochemical properties and sugar fermentation results of these strains

续 表

续 表
2.3 分离菌株基因组DNA提取及PCR扩增产物
分离菌株的基因组DNA电泳见图1。结果表明,基因组DNA的质量满足后续试验要求,可用于16S rDNA序列的扩增。
分离菌株基因组16S rDNA序列PCR扩增产物的电泳图见图2。菌株基因组进行PCR扩增后,所得的扩增片段大小在1500bp左右的特异性条带,满足测序要求。
图1 基因组DNA电泳Fig.1 Gel electrophoresis of genomic DNA
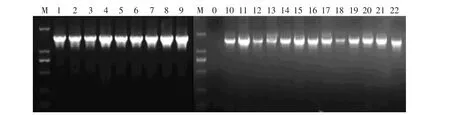
图2 PCR扩增产物电泳Fig.2 Gel electrophoresis of PCR-amplified fragments
2.4 测序结果及16S rDNA序列同源性分析
将标准菌株ATCC4356及21株分离菌株的16S rDNA序列输入到GenBank中,寻找与其同源性最高的菌种,结果见表2。

表2 分离菌株的16S rDNA序列同源性分析Table2 16S rDNA Homology Analysis
由表2可知,分离菌株的16S rDNA序列同GenBank中该属内菌株的16S rDNA已知序列做比对,结果发现ATCC4356与嗜酸乳杆菌(Lactobacillus acidophilus)同源性最高;L4、L14、L17、L21、L22、L33与植物乳杆菌(Lactobacillus plantarum)同源性最高;L3、L5、L6、L12、L18、L19、L28、L30与短乳杆菌(Lactobacillus brevis)同源性最高;L7、L9、L10、L27、L31、L32与罗伊氏乳杆菌(Lactobacillus reuteri)同源性最高;L20与米酒乳杆菌(Lactobacillus sakei)同源性最高;而且同源性均可达到99.4%以上。由此可以推断,对于表形特征发生变化的细菌,在用传统的生理生化方法不能对其进行准确鉴定的情况下,可以通过16S rDNA序列同源性分析的方法对其进行准确鉴定[18-19]。16S rDNA序列同源性分析结果试验结果表明:通过16S rDNA序列同源性分析的方法可以将试验菌株准确鉴定到种的水平。因此可以采用生理生化方法与16S rDNA基因序列测定相结合的方法对微生物进行鉴定。本试验分离得到6株植物乳杆菌,8株短乳杆菌,李文斌等的研究结果也表明:酸菜中的优势乳杆菌为植物乳杆菌和短乳杆菌[20]。除此之外,本试验还分离出1株米酒乳杆菌,6株罗伊氏乳杆菌。本试验的下一步工作将对分离的菌株的特性进行研究。
3 结论
a.从15份自然腌渍的东北酸菜样品中分离得到21株乳杆菌。
b.在传统生理生化初步鉴定的基础上,再利用16S rDNA序列同源性分析方法对21株乳杆菌进行鉴定,发现:6株为植物乳杆菌,8株为短乳杆菌,6株为罗伊氏乳杆菌,1株为米酒乳杆菌。16S rDNA序列同源性分析方法可以准确地将未知细菌鉴定到种的水平。
[1]Stanier k Y,A deberg E A,Lngraham J L.The Microbial World[M].1981.
[2]Uhlman L.Identification and characterization of two bacteriocinproducing strains of lactococcus lactis isolated from vegetables[J].International Journal and Microbiology,1992.
[3]葛菁萍,邹鹏,平文祥.酸菜发酵液中乳酸菌的分离与鉴定[J].食品工业科技,2007,28(10):83-84.
[4]陈晓平,刘华英,魏小川.自然发酵酸菜汁中乳酸菌的分离筛选与鉴定研究[J].食品科学,2006,27(2):91-94.
[5]李凤梅,王晓,张双灵.自然发酵酸菜汁中乳酸菌分离鉴定[J].中国酿造,2008,5(182):33-35.
[6]凌代文.乳酸细菌分类鉴定及实验方法[M].北京:中国轻工业出版社,1999.
[7]中国科学院微生物研究所细菌分类组.一般细菌常用鉴定方法[M].北京:科技出版社,1978.
[8]陈天寿.微生物培养基的制造与应用[M].北京:中国农业出版社,1995.
[9]萨姆布鲁克J,拉塞尔D W.分子克隆实验指南[M].8版.北京:科学出版社,2003.
[10]布坎南R E伯杰细菌鉴定手册[M].8版.北京:科学出版社,1984.
[11]东秀珠.常见细菌系统鉴定手册[M].北京:科学出版社,2001.
[12]mauro S,Diego M,Silvia C,et al.Development of genus/speciesspecific speciesspecific PCR analysis for identification of carnobacterium strains[J].Current Microbiology,2002(45):24-29.
[13]Antara N S,Sujaya I N,Yokota A,et al.Identification and succession of lactic acid bacteria during fermentation of'urutan',a Balinese indigenous fermented sausage[J].World Journal of Microbiology and Biotechnology,2002,18(3):255-262.
[14]Vogel R F,Bocker G,Stolz P,et al.Identification of Lactobacilli from sourdough and description of Lactobacillus pontis sp.Nov[J].Int J Syst Bacteriol,1994,44:223-229.
[15]乌日娜,张和平,孟和毕力格.酸马奶中乳杆菌Lb.casei.Zhang和ZL12-1的16S rDNA基因序列及聚类分析[J].中国乳品工业,2005,33(6):4-9.
[16]Rodolphe B,Sung-Sik Y,Frederick B,et al.Identification andcharacterization of Leuconostoc fallax strains isolated from anindustrial sauerkraut fermentation[J].Applied and Environ Microbiol,2002,68(6):2877-2883.
[17]张刚.乳酸细菌-基础、技术和应用[M].北京:化学工业出版社,2007.
[18]Woo P C,Chong K T,Leung K,et al.Identification of Arcobacterc-ryae rophilus isolated from a traffic accident victim with bacteremiaby 16S ribosomal RNA gene sequencing[J].Diagn Microbiol Infect Dis,2001,40:125-127.
[19]Woo P C,Cheung E Y,Leung K,et al.Identification by 16Sribosomal RNA gene sequencing of an Enterobacteriaceae species with ambiguous biochemical profile from a renal transplant recipient[J].Diagn Microbiol Infect Dis,2001,39:85-93.
[20]李文斌,宋敏丽.自然发酵泡菜微生物群落变化的研究[J].中国食物与营养,2008,11:22-24.
