利用代谢工程技术提高工业微生物对胁迫的抗性
2010-02-10付瑞燕李寅
付瑞燕,李寅
1 安徽农业大学茶与食品科技学院,合肥 230061
2 中国科学院微生物研究所,北京 100101
利用代谢工程技术提高工业微生物对胁迫的抗性
付瑞燕1,李寅2
1 安徽农业大学茶与食品科技学院,合肥 230061
2 中国科学院微生物研究所,北京 100101
代谢工程是工业微生物菌种改造的平台技术,不仅可用于改变微生物细胞内的代谢流向,也可以用于改善工业微生物的生理功能。在工业生产过程中,微生物细胞会面临多种胁迫作用,这些胁迫诱导的基因调节作用,都有可能影响细胞的许多重要生理功能,从而影响生物转化过程的效率。从工业应用的观点出发,选择生产性能良好、对发酵过程中的主要胁迫因素有较强耐受性的菌株至关重要。以下评述了借鉴传统代谢工程技术和反向代谢工程技术来提高工业微生物对胁迫抗性的若干研究策略,提出了该领域目前存在的问题,以及利用代谢工程技术改善微生物胁迫抗性——即微生物生理功能工程的发展方向。
代谢工程,工业微生物,胁迫抗性,生理功能工程
随着近年来代谢工程的迅猛发展,定向改变和优化微生物的生理功能即微生物生理功能工程也逐渐成为代谢工程的应用领域之一[1],以下就近年来借鉴代谢工程技术的思想,提高工业微生物胁迫抗性相关领域的最新方法和策略的进展作简要综述。
1 借鉴传统代谢工程技术构建胁迫抗性突变株
代谢工程发展早期,人们注重的是对生化和遗传背景清楚的菌株,利用DNA重组技术对特定的代谢途径进行目的性修饰、设计与构建,在相当程度上更侧重于分子生物学技术的表现形式。在具体的实践中,常采用的方法有:构建新的代谢途径、拓展已有的代谢途径和削弱已有的代谢途径。构建新的代谢途径是指引入外源抗性基因来改造和修饰代谢网络,使细胞从不能合成某种代谢产物转变为能够合成此代谢产物。拓展已有的代谢途径是指通过基因工程手段引入外源基因,使原有代谢途径进一步向前或向后延伸,从而与相关的代谢途径相连,组成完整有效的抗胁迫途径。削弱已有的代谢途径是指通过抑制内源性基因的表达或敲除内源性基因,降低细胞中已有代谢途径的流量。已有的研究表明,借鉴上述这 3种传统代谢工程手段都可以提高微生物对胁迫的抗性。
1.1 构建新的代谢途径合成胁迫抗性物质
乳酸乳球菌是乳球菌属最重要和最典型的一个种。一直以来,乳酸乳球菌被人们用来作为乳制品发酵剂,如今又被发展成为生产食品级代谢产物的超级“细胞工厂”[5],是原核细菌中一种重要的工业微生物。乳酸乳球菌的生长依靠发酵产能,不需要氧的参与。并且,乳酸乳球菌缺乏高效的抗氧胁迫系统,其好氧生长一般是弱于其静置或厌氧生长的。有氧呼吸代谢产生的能量要远远高出发酵产能,因此,当乳酸乳球菌作为宿主来生产外源基因产物时,较低生物量和较长发酵周期必然会影响生产强度。提高对氧的耐受性是工业生产对乳酸乳球菌提出的一个要求。
乳酸乳球菌的抗氧胁迫系统中没有过氧化氢酶(Catalase,CAT),但存在超氧化物歧化酶 (Superoxide dismutase,SOD)。SOD在清除超氧阴离子自由基时会产生 H2O2,且 H2O2往往会积累在乳酸乳球菌的发酵液中。H2O2不仅是酵解过程中多种酶的抑制剂,还会引发毒性更大的羟自由基的产生。所以,H2O2被认为是包括乳酸乳球菌在内的许多乳酸菌好氧代谢所产生的最重要的抑制性物质之一。绝大多数需氧微生物都含有CAT,CAT具有很高的转换数,一个CAT分子可以每秒钟将数百万个H2O2分子转化为水和氧气,从而使细胞免于遭受H2O2的毒害,是细胞内H2O2代谢的关键酶。因此,将编码CAT的外源基因导入乳酸乳球菌成为构建抗氧胁迫突变株的首选策略。Rochat等[6]从枯草芽胞杆菌中克隆编码依赖血红素的过氧化氢酶基因KatE,将其插入到乳酸乳球菌 NZ9000的 nisin启动子和信号肽SPUsp45下,结果有活性的 KatE分泌到了发酵液中。在4 mmol/L H2O2处理乳酸乳球菌1 h的氧胁迫条件下,重组菌的存活率比对照 (不含 KatE基因)提高800倍,表明KatE赋予乳酸乳球菌更强的H2O2抗性。并且,重组菌在振荡条件 (240 r/min) 下培养3 d的存活率是对照的160倍。这表明,通过构建新的代谢途径合成 CAT可以有效地提高宿主菌乳酸乳球菌抵抗氧胁迫的能力。
1.2 拓展已有的代谢途径组成完整有效的抗胁迫途径
由γ-谷氨酰半胱氨酸合成酶、谷胱甘肽合成酶、谷胱甘肽还原酶 (Glutathione reductase,GR)、谷胱甘肽过氧化物酶 (Glutathione peroxidase,GPx) 和NADPH组成的依赖于谷胱甘肽 (Glutathione,GSH)的GPx系统对于酿酒酵母细胞具有不可替代的抗氧化作用。乳酸乳球菌中存在 GR和依赖于 GSH的GPx。虽然乳酸乳球菌不能合成 GSH,但可以从培养基中吸收GSH,并且被乳酸乳球菌吸收到胞内的 GSH能够提高细胞抵抗氧胁迫的能力。以上研究表明乳酸乳球菌中可能存在着一个不完整的抗氧胁迫途径,因此,将GSH的合成代谢途径导入乳酸乳球菌,有可能可以拓展已有的 GSH相关代谢途径,从而构建出一条完整的依赖于GSH的抗氧胁迫防线。
作者以能够生物合成 GSH的乳酸乳球菌NZ9000 (pNZ3203) 为实验菌株,对GSH在乳酸乳球菌工程菌株抗氧胁迫的能力进行了研究[7],结果表明GSH可以提高宿主菌NZ9000对较高H2O2胁迫剂量 (50 mmol/L H2O2,15 min) 和甲萘醌 (超氧阴离子自由基生成剂) 所引发氧胁迫的抗性。由此表明,通过代谢工程手段在乳酸乳球菌 NZ9000中引入GSH合成能力,可以显著提高宿主菌对氧胁迫的抗性。
1.3 削弱已有的代谢途径
酿酒酵母具有其他微生物所不具备的高乙醇耐受性,因此自古以来被广泛地应用在乙醇生产中。但当酿酒酵母自身产生的乙醇达到一定浓度时,对它本身也会产生毒性。随着乙醇浓度的增加,酿酒酵母的增殖速度及存活率下降,进一步提高乙醇的浓度,酿酒酵母的发酵会停止。因而关于酿酒酵母的乙醇耐受性的研究始终是酵母研究者关注的重要课题之一。
海藻糖 (Trehalose) 能提供微生物细胞碳源和能量,稳定细胞膜和蛋白质,保护细胞抵御胁迫。Jung等[8]根据酿酒酵母 ATH1 (编码酸性海藻糖酶)基因序列,PCR扩增ATH1基因中+1~+500碱基序列的反义基因片段,分别与ADH1、CYC1和ATH1启动子融合,插入到质粒中,再将重组质粒导入酿酒酵母。结果表明,含有ATH1反义基因的3株酿酒酵母重组菌中的酸性海藻糖酶表达水平均有所降低,其中重组质粒中启动子为ADH1的酿酒酵母重组菌的下降幅度最大。进一步研究发现,与对照菌相比,酿酒酵母重组菌的乙醇产量更高,乙醇生成速率也更快。酿酒酵母重组菌在含有30%葡萄糖的YPD培养基中培养,乙醇产量达到最高所需的发酵时间从88 h缩短至44 h。在乙醇浓度为8%的胁迫条件下,与对照相比,重组菌生长速率快;并且,生长8 h后,重组菌的存活率是对照的1.5倍。以上研究表明抑制海藻糖酶的表达水平可以提高酿酒酵母对乙醇的抗性。
与乳酸乳球菌抗氧胁迫突变株相比,酿酒酵母乙醇胁迫突变株的构建效果并不理想,抗性提高幅度并不高。原因可能是目前对于酿酒酵母乙醇耐受机制还未深入了解,并且该胁迫是多基因参与的复杂胁迫过程[9],因此瞄准个别基因的做法收效甚微。对于这类机制较复杂的胁迫过程,必须寻找到所有必要的胁迫相关基因,才有可能获得优良的胁迫抗性突变株。
2 借鉴反向代谢工程技术构建胁迫抗性突变株
传统代谢工程虽然在氨基酸、核苷酸等高产菌的选育中取得成功,但对单个代谢途径进行改造,由于微生物代谢网络的全局调控,往往并不能获得预期的效果。基因组学和功能基因组学的发展,提供了从全局规模上深刻认识微生物生理和代谢特性的工具,从而推动了一种新的代谢工程设计策略——反向代谢工程。如前所述,生理功能工程特别是胁迫生理功能的改造也遇到了类似的困难,因此,反向生理功能工程策略也应运而生[1]。反向生理功能工程可以在对胁迫机理没有充分了解的基础上,直接找出所希望表型的关键基因,为构建特定微生物的胁迫突变株提供靶基因。目前,反向生理功能工程的研究主要借鉴反向代谢工程的手段来进行,其基本研究步骤主要由以下3个方面组成:
2.1 在异源微生物或相关模型系统中获得预期的表型
应用反向生理功能工程,第一步是获得预期的表型。目前可以采用的方法有结合高通量筛选方法的传统诱变技术、全转录工程 (Global transcription machinery engineering,gTME) 和基因组重排 (Genome shuffling) 等。
传统的诱变技术是一个行之有效的优良菌株的选育技术,借助高通量的检测手段,可以快速有效地从由化学诱变剂诱变过的突变群体中筛选出预期表型,如Klein-Marcuschamer等[10]用小剂量NTG (致死率40%~50%) 诱变植物乳杆菌野生菌株,先后以pH为4.6±0.05 (用质量浓度为5.5 g/L的L-乳酸调节)或pH为3.85±0.05 (用HCl调节) 为筛选条件获得耐受乳酸和低pH值的突变群体,继代培养2次,随后涂布平板以分离单菌落。将分离出的菌株以相同的起始OD值接种在96孔板中,在苹果酸终浓度为100 mmol/L (pH为4.0±0.1) 的胁迫条件下培养,以低于10%的选出率高通量免摇瓶筛选出了稳定期OD值 (30~40 h) 高于野生菌株的酸胁迫抗性突变株。然而同样条件下用gTME法获得的酸胁迫抗性突变株选出率高于25%。为探究此结果背后的原因,进一步作了深入研究。将用2种方法所获得的突变株库液体培养后高倍稀释涂布在96孔板上,以获得单菌落。在固体培养基中添加终浓度为900 mmol/L的NaCl、60 mmol/L的HCl和4 g/L的L-乳酸作为胁迫因子,培养后平板放置在 4℃下过夜以使细胞停止生长,再用高性能图像分析系统 AlphaImager 3400 System定量分析菌落生长速率。结果表明,用NTG诱变获得的突变株库表型分离度显著低于用gTME获得的突变株库,从而胁迫抗性突变株的筛出机率也随之降低。
基因的结构活化、转录起始、转录后加工及转运、mRNA降解、翻译及翻译后加工及蛋白质降解等均为基因表达调控的控制点。其中在转录起始水平上的调控是最为经济有效的重要方式。gTME就是这样一个基于转录起始水平上的调控方法,它通过使整个基因组的转录发生全局性改变,从而导致许多由多种基因控制的复杂的细胞表型得到改变,最终获得需要的优良表型[11]。利用该方法获得预期胁迫抗性突变株的基本程序为:首先通过突变几个影响全局转录的关键蛋白来造成多基因转录水平的改变,筛选出胁迫抗性突变株,然后通过比较出发菌株和突变菌株的转录谱,得到与某一胁迫抗性表型相关的基因。原核生物和真核生物的转录机制不同,因而可以在转录水平上对细胞表型调控产生全局性扰动的关键蛋白也各不相同。
原核生物仅含有一种RNA聚合酶,可以直接起始转录合成RNA,通常由5个亚基组成,即σ、β、β'和2个α亚基,研究表明RNA聚合酶σ亚基和α亚基的突变可以提高大肠杆菌对胁迫的耐受性。例如,Alper等[12]通过对将大肠杆菌中编码σ70因子的rpoD基因及其上游启动子之间的序列进行几轮易错 PCR扩增,使大肠杆菌整个基因组的转录发生全局性改变,最终获得了在高浓度乙醇环境下耐受能力大幅度提高的胁迫抗性突变株。Klein-Marcuschamer等[13]构建了大肠杆菌 RNA 聚合酶α亚基编码基因(rpoA) 的突变株库,结果表明,RNA聚合酶α亚基的突变有助于提高大肠杆菌对丁醇的耐受性。
真核生物有3种RNA聚合酶,分别催化不同类型RNA的合成,均不能独立转录RNA,都必须在蛋白质转录因子的协助下才能进行RNA的转录。转录因子分为通用转录因子与序列特异的转录因子两大类。前者因参与了几乎所有基因的转录而得名,是RNA聚合酶结合启动子所必需的一组因子,为所有mRNA转录起动共有,包括TFIIA、TFIIB、TFIID、TFIIE 以及 TFIIF 等成员。后者则为个别基因转录所必需,决定该基因的时间、空间特异性表达。Alper等[11]利用基于通用转录因子的gTME方法,运用易错 PCR改变酿酒酵母中 RNA聚合酶Ⅱ转录因子TFIID中TATA盒结合蛋白SPT15和TATA盒结合蛋白相关因子TAF25的编码基因,构建2个gTME突变文库后转化入酿酒酵母中。通过在培养基中逐步提高乙醇和葡萄糖的浓度,筛选出了乙醇和葡萄糖耐受性提高的突变株spt15-300。与对照相比,突变株spt15-300 的生长性能显著提高,对数期延长,生物量更高,乙醇产率也更高。可见,作为一种基于已有的基因知识来设计的代谢工程技术,gTME可以更理性地对工业微生物的表型进行优化。
利用基因组重组进行胁迫抗性突变株定向筛选的基本方法为:首先用传统的诱变方法获得一个突变株库,筛选出若干胁迫抗性提高的突变株作为出发株,然后以多轮递归原生质体融合的方式使众多基因随机重组,最终从获得的突变株库中筛选出抗性显著升高的突变株。克鲁斯假丝酵母是一种极具潜力的燃料乙醇生产菌种。但是发酵副产物乙酸会抑制酵母细胞的生长和代谢,加剧乙醇对细胞的抑制作用。并且,乙酸普遍存在于木质纤维素水解产物中,因此,提高酵母对乙酸的耐受性是需要解决的一个重要问题。Wei等[14]利用紫外线诱变克鲁斯假丝酵母 GL560的原生质体,随后涂布在0.7%含乙酸的YPDK平板上,获得了抗酸性能稳定提高的突变株。经过 4轮递归原生质体融合,最终筛选到了一个乙酸耐受性显著提高的突变株S4-3。在乙酸体积浓度为0.5%的平板上,突变株S4-3的存活率是出发菌株GL560的30倍。进一步研究发现,突变株抗性提高的机制可能与细胞膜完整性和胞内CAT活性的提高有关。基因组重排技术不必了解菌株的遗传特性,在细胞水平上即可进行定向进化,快速获得具有优良表型的工业微生物,是一个十分有效的工具。如今,基因组重排的方法已成功应用于多种微生物的胁迫耐受性的改良方面[15-16]。
除了上述几种成熟的方法外,近年来,核糖体工程 (Ribosome engineering) 和基因组多重自动修复技术 (Multiplex automated genome engineering,MAGE) 的研发也将为获得理想突变表型提供可借鉴的方法。
在微生物细胞生长后期都会面临营养物质缺乏、代谢产物积累的胁迫条件,此时的环境条件所引发的胁迫会抑制细胞的代谢活性,不利于生产强度的提高。核糖体是蛋白质的合成机器,也是细胞感知营养水平和对生长速率进行调控的重要位点[17]。研究表明,在生长后期,某些在生长前期沉默基因 (如编码次级代谢产物的基因) 的激活表达与核糖体的功能密切相关,利用“核糖体工程”育种技术向微生物核糖体中引入特定的突变可获得抗生素合成能力提高的突变株[18]。微生物可通过释放和感知信号分子控制基因表达以适应环境。例如,多种杆菌属所产生的氨基糖类抗生素 NTD (Neotrehalosadiamine) 就是一种称为自诱导物的信号分子,可诱导基因glcP(特异性控制细胞对葡萄糖的吸收)表达水平显著上调[19],表明 NTD可通过加强营养的吸收来帮助产生菌在恶劣的环境中生存,事实上,这也是选育抗营养胁迫突变株的一种尝试。因此,预期核糖体工程在获得抗营养胁迫突变株方面会有重要应用。
近来,《Nature》杂志报道了一种称为基因组多重自动修复技术的高速获得理想细胞表型的新方法[20]。Wang等[20]从大肠杆菌的4 500个基因中选取了有可能改善番茄红素生物合成能力的24个基因,将它们的DNA序列分割成易于处理的90个字母的片段,进行基因修饰后,每个片段都携带了单一的突变。利用这些特定序列,研究人员制成数千个独特的基因结构,通过电转化,将它们重新插回细胞。通过加速重组,研究小组在 3 d内创造出多达 150亿个基因突变体,并使番茄红素的产量提高了5倍,而用传统的克隆技术产生 150亿个突变需要花费数年时间。美国加利福尼亚大学洛杉矶分校的生物工程学家Liao指出:“这是一种普遍适用的方法”。因此有理由相信,随着人们对胁迫抗性相关基因的发现,利用MAGE平台将有力助推工业微生物胁迫抗性突变株的构建。
2.2 确定导致这一表型的遗传基因
应用反向生理功能工程,第二步是快速找出突变表型所对应的基因型。目前可以采用的技术包括“组学”技术、人工转录因子工程 (Artificial transcription factor engineering) 和文库富集尺度分析技术 (Scalar analysis of library enrichments,SCALEs) 等。
“组学”技术 (基因组学、转录组学、蛋白质组学、代谢物组学、代谢通量组学等) 在表型表征中起着极为重要的作用。例如,Hirasawa等[21]利用基因微阵列技术,在乙醇体积浓度为5%的条件下,对一株具有较高乙醇胁迫抗性的工业菌株酿酒酵母IFO2347和一株实验室菌株酿酒酵母FY834进行基因表达谱的描绘。随后对基因微阵列数据进行聚类分析,将目的基因的搜索范围从 6 000个基因降至400个基因。构建了这 400个基因的缺失突变株,测定所有突变株对乙醇胁迫体积浓度为 5%时的敏感度,最终找出色氨酸合成基因可能是与乙醇胁迫抗性密切相关的基因。向培养基中添加色氨酸、过量表达色氨酸透性酶基因,均能增加酵母细胞对乙醇胁迫的抗性。并且,过量表达色氨酸合成基因的FY834突变株,对乙醇胁迫的抗性与菌株 IFO2347相当。这些进一步的研究结果验证了预测的正确性。这是基于“转录组”的代谢工程。
然而,细胞的生理功能是许许多多基因产品(mRNA、蛋白质、代谢物) 共同参与的网络信息调控的结果,比如,基因表达不仅仅是从转录组到蛋白质组的单向流动,表达的蛋白质或者是细胞生化状态下其他的变化也会对转录组本身进行反馈控制,因而广泛的基因分析应该建立在基因产品的全部水平上,才能对细胞的响应有更好的理解。为此,不同“组学”技术的组合越来越多地应用在代谢工程中。例如,丁醇是一种重要的化工原料和极具潜力的新型生物燃料,然而丁醇对细胞具有高毒害性导致丁醇产量不高,这是发酵法生产丁醇的一个关键限制因素。Rutherford等[22]对大肠杆菌的蛋白质组、转录组和代谢物组进行了分析,以综合理解细胞对丁醇体积浓度为0.8%的胁迫环境的响应。结果表明丁醇胁迫会引发细胞呼吸功能的扰动、氧胁迫、热休克和胞外被膜应激反应 (Cell envelope stress),因而很难通过敲除或引入某个基因来显著提高对丁醇胁迫的抗性。进一步对高通量组学分析技术得出的数据进行整合,鉴定出了很多关键的基因靶点,从而为代谢工程进一步改造菌种提供了依据。例如候选基因malE、yqhD和ompF,通过构建相应的缺失突变株,证实这些突变株对丁醇的敏感性增加,表明有可能通过过量表达这些基因来减轻丁醇对细胞的毒害。
“组学”技术只能分析表型对应的基因型,而人工转录因子工程和文库富集尺度分析技术可以将获得期望表型和寻找基因型的过程“合二为一”,在创造新的基因型的同时较为精准地定位靶基因。
天然的序列特异转录因子通常由 DNA结合结构域与效应结构域两部分构成。DNA结合结构域能够特异地识别并且结合其对应的DNA序列,使该转录因子能够定位到靶基因;效应结构域通常是一个激活因子或者抑制因子,能够激活或者抑制靶基因的表达。研究发现,这 2个结构域是各自独立起作用的,于是可以将不同的DNA结合结构域与效应结构域组合在一起,人为地构建具有新的序列特异性与作用效果的转录因子,即人工转录因子[23]。其中,构建特异性的 DNA结合域是成功构建特异性人工转录因子的核心工作。锌指蛋白 (Zinc finger proteins,ZFPs) 以其小巧灵活、结构独特常作为DNA结合域的首选结构。研究发现,ZFPs可以作为揭示特定表型与基因型之间关系的新工具。Park等[24]将源自酵母ZFP文库中的三指和/或四指的ZFP编码基因随机重排,克隆入质粒载体pZL1,在大肠杆菌DH5α中表达,构建了一个ZFP文库,在50℃下培养2 h,从1×107个转化子中筛选出了23个存活细胞。从耐热表型中分离鉴定出10种ZFP,对其中诱导宿主菌产生抗热胁迫能力效果最好的T9 ZFP用定位突变法和染色质免疫沉淀实验进一步研究,发现T9 ZFP通过与目标基因 ubiX (编码辅酶 Q生物合成的基因) 结合,导致靶基因表达水平下调而增强细胞的耐热性,接下来的 ubiX敲除实验结果也证实了这一点。
Warnecke等[25]利用“文库富集尺度分析”技术(图1[26]),控制条件将大肠杆菌K12的基因组DNA切割成精确界定大小的片段,并与质粒载体pSMART-LCKAN重组后导入大肠杆菌 Mach1-T1进行克隆,构建了大肠杆菌的基因文库。通过逐步提高3-HP的浓度来营造胁迫环境,只有质粒中含有3-HP抗性基因的重组细胞才能存活下来。抽提质粒DNA,与基因芯片杂交,用SCALEs软件定量分析4×105个菌落的高精度生长表型数据,利用已知的代谢网络数据库EcoCyc、COGs等分析基因-基因交互作用网络 (Interaction networks),计算途径适应度和途径频度分析抗性细胞的代谢途径,结果显示通过确定受抑制的途径 (分支酸和苏氨酸途径),对这些途径进行组合修饰可以显著提高3-HP抗性 (最高可达 25倍),效果远远优于对编码个别酶的基因操作所达到的水平。
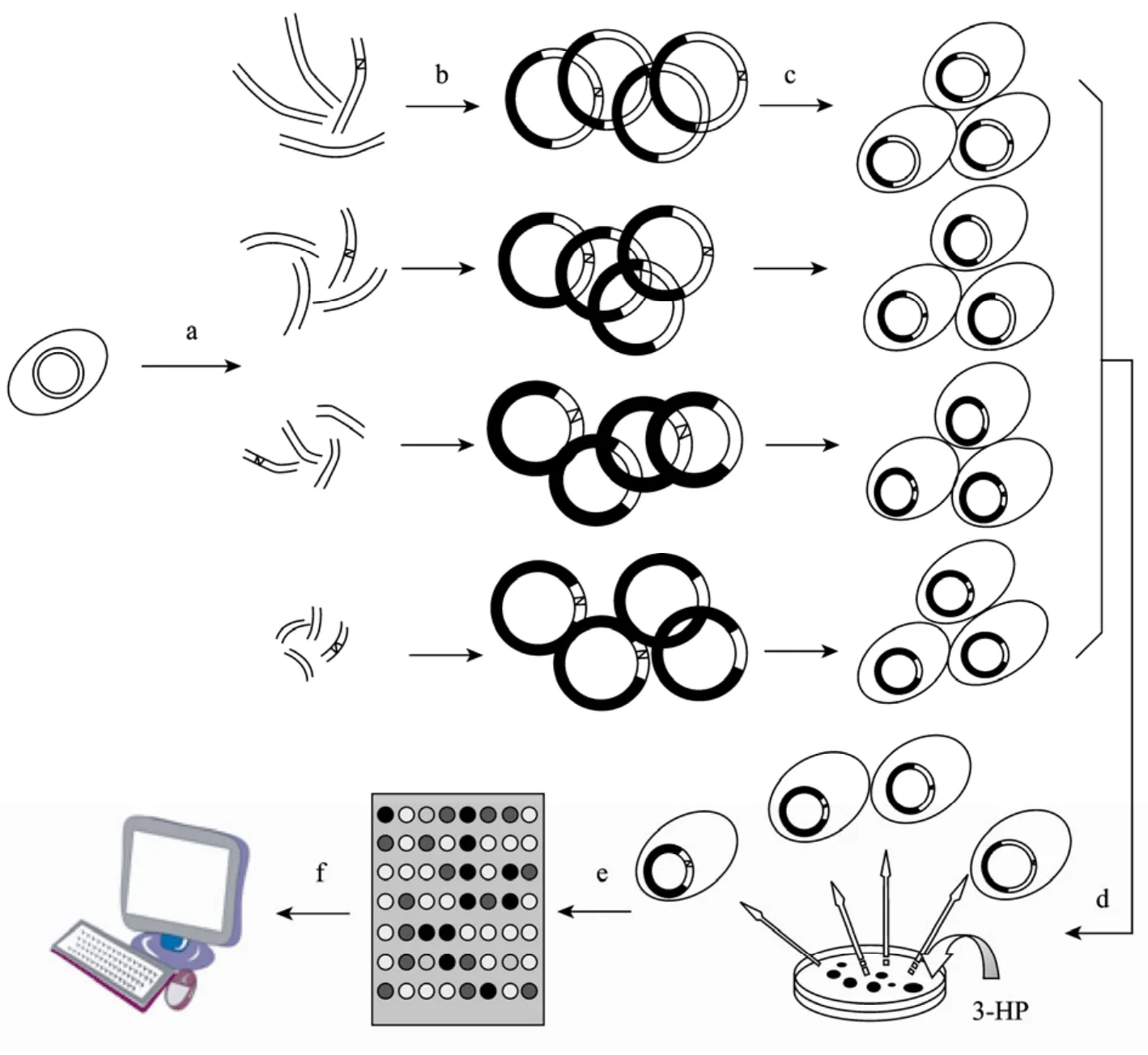
图1 文库富集尺度分析技术图解Fig. 1 Overview of SCALEs, which was adapted from Fig. 1 of reference 26. a-b: genomic DNA from wild-type Escherichia coli K12 fragmented to several specific sizes was ligated into vectors creating several libraries with defined insert sizes; c: these libraries were individually transformed into E. coli strain Mach1-T1 used for selections. Each of these libraries each contained enough clones to ensure with >99% probability that the entire genome was represented; d: the pools of transformants were mixed and subjected to selection with decreasing concentrations of 3-HP. Only clones bearing plasmids with insert increasing fitness survive; e: enriched plasmids were purified from the selected population, prepared for hybridization to Affymetrix Genechips. SCALEs algorithm was then used to obtain high resolution growth phenotype data; f: genetic level fitness data were mapped according to various definitions of gene-gene interaction such as metabolic networks from EcoCyc, GOGs, etc.
2.3 通过遗传改造使这一表型在特定微生物中表达
反向生理功能工程的最后一步是把鉴定出的目标基因导入特定微生物中,使目标菌株获得所希望的理想表型。虽然这一研究策略在改造微生物的代谢能力方面已有许多成功的实例[27-28],但是,在包括胁迫抗性在内的其他生理功能的改造方面至今还未见报道,这种状况可能正说明反向代谢工程的研究策略并不能完全适用于反向生理功能工程。
3 存在的问题与展望
工业环境下的微生物总是面临着不同种类的胁迫。要显著提高工业微生物对生产环境下胁迫的适应能力,需要首先了解微生物应答不同胁迫条件的生理机制,在此基础上才能发展出相应的方法,改善工业微生物细胞的生理功能。对于那些胁迫机理研究透彻,关键基因已经明确的胁迫生理响应过程来说,可以采用经典基因工程的方法,对个别基因/途径进行改造。但经典基因工程的应用中也存在着问题,首先,基因的相互依存性会导致一个基因的表达不一定会带来人们所预期的相关活性的产生,有时候还有可能会对细胞的代谢产生正效应或负效应。例如,在前期研究中,为改善乳酸乳球菌的生长性能,我们以轮枝链霉菌染色体DNA为模板,扩增得到编码谷氨酰胺转胺酶成熟酶的基因 mtg,将其克隆到乳酸乳球菌NZ9000,结果与对照菌 (不含mtg基因) 相比,重组菌不仅胞外pH 明显升高,而且对氧的耐受性也得到显著改善[29]。这种正效应表明mtg基因的导入对乳酸乳球菌的其他代谢途径产生了较大的扰动,需要进一步研究代谢网络及调控机理,验证有关基因或途径的功能,在此基础上再进行合理设计,理性地构建胁迫抗性突变株。
其次,外源途径的导入可能会打破宿主细胞的代谢平衡,对细胞产生毒害效应。例如,Martin等将异戊烯焦磷酸途径导入大肠杆菌生产类异戊二烯,虽然重组菌的类异戊二烯产量较高,但细胞生长明显受到了抑制。Piteraa等[30]采用基因滴度研究方法并结合液质联用分析代谢物谱发现,其原因在于内源 HMG-CoA还原酶不足以平衡外源途径的流量,导致中间产物HMG-CoA的积累使碳流量受限。通过调节HMG-CoA还原酶的产生,消除了此途径“瓶颈”,结果类异戊二烯产量得到进一步提高。因此,当需要对某条途径进行代谢工程改造时,可以利用前述的“精确调节表达工程”(Fine-tuning expression engineering) 在途径表达和细胞活性方面寻求最佳平衡点[31]。
胁迫所引发的细胞内mRNA、蛋白质、代谢流的变化不是孤立存在而是相互联系的,单一的“组学”分析不利于理解细胞对于突变的响应。如今,基于整体性研究为特点的系统生物学 (Systems biology)的系统代谢工程 (Systems metabolic engineering)已成为改造细胞复杂代谢调控网络的有力研究工具,通过研究所有相关基因、蛋白质间的所有相互关系,正确选择微生物遗传选育的操作点,使微生物的菌种选育更具正突变性,这项技术已在过量生成代谢产物方面取得成功[32]。可以相信,系统代谢工程技术定会为微生物生理功能工程的发展带来新的契机。随着更多的重要工业微生物的基因组序列完成测序及“组学”技术研究的深入发展,以及计算生物学的发展,有望在不远的将来,能够利用多尺度多层次的微生物生理功能工程技术快速探明工业生产菌株的所有胁迫抗性关键基因,构建出更能适应工业生产条件的生产菌株。
[1] Zhang YP, Zhu Y, Zhu Y, et al. The importance of engineering physiological functionality into microbes. Trends Biotech, 2009, 27(12): 664–672.
[2] Ka´da´r Z, Maltha SF, Szengyel Z, et al. Ethanol fermentation of various pretreated and hydrolysed substrates at low initial pH. Appl Biochem Biotechnol, 2007, 137-140(1/12): 847–858.
[3] Shaw AJ, Podkaminer KK, Desai SG, et al. Metabolic engineering of a thermophilic bacterium to produce ethanol at high yield. Proc Natl Acad Sci USA, 2008, 105(37): 13769–13774.
[4] Xu JJ, Liu PB, Tao CP, et al. 1, 3-Propanediol production under salt stress. Chin J Biotech, 2008, 24(6): 1098–1102.徐佳杰, 刘朋波, 陶春平, 等. 1,3-丙二醇发酵过程中盐浓度的胁迫作用. 生物工程学报, 2008, 24(6): 1098–1102.
[5] Mierau I, Leij P, van Swam I, et al. Industrial-scale production and purification of a heterologous protein in Lactococcus lactis using the nisin-controlled gene expression system NICE: the case of lysostaphin. Microb Cell Fact, 2005, 4: 15–23.
[6] Rochat T, Miyoshi A, Gratadoux JJ, et al. High-level resistance to oxidative stress in Lactococcus lactis conferred by Bacillus subtilis catalase KatE. Microbiology, 2005, 151: 3011–3018.
[7] Fu RY, Bongers RS, van Swan II, et al. Introducing glutathione biosynthetic capability into Lactococcus lactis subsp cremoris NZ9000 improves the oxidative-stress resistance of the host. Metab Eng, 2006, 8: 662–671.
[8] Jung YJ, Park HD. Antisense-mediated inhibition of acid trehalase (ATH1) gene expression promotes ethanol fermentation and tolerance in Saccharomyces cerevisiae. Biotechnol Lett, 2005, 27: 1855–1859.
[9] Nevoigt E. Progress in metabolic engineering of Saccharomyces cerevisiae. Microbiol Mol Biol Rev, 2008, 72(3): 379–412.
[10] Klein-Marcuschamer D, Stephanopoulos G. Assessing the potential of mutational strategies to elicit new phenotypes in industrial strains. Proc Natl Acad Sci USA, 2008, 105(7): 2319–2324.
[11] Alper H, Moxley J, Nevoigt E, et al. Engineering yeast transcription machinery for improved ethanol tolerance and production. Science, 2006, 314: 1565–1568.
[12] Alper H, Stephanopoulos G. Global transcription machinery engineering: a new approach for improving cellular phenotype. Metab Eng, 2007, 9(3): 258–267.
[13] Klein-Marcuschamer D, Santos CNS, Yu H, et al. Mutagenesis of the bacterial RNA polymerase alpha subunit for improvement of complex phenotypes. Appl Environ Microbiol, 2009, 75(9): 2705–2711.
[14] Wei PY, Li ZL, He P, et al. Genome shuffling in the ethanologenic yeast Candida krusei to improve acetic acid tolerance. Biotechnol Appl Biochem, 2008, 49: 113–120.
[15] Wang Y, Li Y, Pei X, et al. Genome-shuffling improved acid tolerance and L-lactic acid volumetric productivity in Lactobacillus rhamnosus. J Biotechnol, 2007, 129(3): 510–515.
[16] Shi DJ, Wang CL, Wang KM. Genome shuffling to improve thermotolerance, ethanol tolerance and ethanol productivity of Saccharomyces cerevisiae. J Ind Microbiol Biotechnol, 2009, 36: 139–147.
[17] Xie SJ, Xiao J, Xu J. Advance in microbial ribosome engineering. Acta Microbiol Sin, 2009, 49(8): 981–986.谢庶洁, 肖静, 徐俊. 微生物核糖体工程研究进展. 微生物学报, 2009, 49(8): 981–986.
[18] Ochi K. From microbial differentiation to ribosome engineering. Biosci Biotechnol Biochem, 2007, 71(6): 1373–1386.
[19] Inaoka T, Ochi K. Glucose uptake pathway-specific regulation of synthesis of neotrehalosadiamine, a novel autoinducer produced in Bacillus subtilis. J Bacteriol, 2007, 189(1): 65–75.
[20] Wang HH, Isaacs FJ, Carr PA, et al. Programming cells by multiplex genome engineering and accelerated evolution. Nature, 2009, 460: 894–898.
[21] Hirasawa T, Yoshikawa K, Nakakura Y, et al. Identification of target genes conferring ethanol stress tolerance to Saccharomyces cerevisiae based on DNA microarray data analysis. J Biotechnol, 2007, 131: 34–44. [22] Rutherford BJ, Dahl RH, Price RE, et al. Functional genomic study of exogenous n-butanol stress in Escherichia coli. Appl Environ Microbiol, 2010, 76(6): 1935–1945.
[23] Zhao XH, Zhu XD, Huang PT. Artificial transcription factors as tools for gene expression manipulation. Chin J Biotech, 2005, 21(3): 341–347.赵兴卉, 朱旭东, 黄培堂. 人工转录因子研究进展. 生物工程学报, 2005, 21(3): 341–347.
[24] Park KS, Jang YS, Lee H, et al. Phenotypic alteration and target gene identification using combinatorial libraries of zinc finger proteins in prokaryotic cells. J Bacteriol, 2005, 187(15): 5496–5499.
[25] Warnecke TE, Lynch MD, Karimpour-Fard A, et al. Rapid dissection of a complex phenotype through genomic-scale mapping of fitness altering genes. Metab Eng, 2010, 12(3): 241–250.
[26] Lynch MD, Warnecke T, Gill RT. SCALEs: multiscale analysis of library enrichment. Nature Methods, 2007, 4(1): 87–93.
[27] Park JH, Lee KH, Kim TY, et al. Metabolic engineering of Escherichia coli for the production of L-valine based on transcriptome analysis and in silico gene knockout simulation. Proc Natl Acad Sci USA, 2007, 104(19): 7797–7802.
[28] Alper H, Miyaoku K, Stephanopoulos G. Construction of lycopene-overproducing E. coli strains by combining systematic and combinatorial gene knockout targets. Nat Biotechnol, 2005, 23: 612–616.
[29] Fu RY, Chen J, Li Y. Heterologous leaky production of transglutaminase in Lactococcus lactis significantly enhances the growth performance of the host. Appl Environ Microbiol, 2005, 71(12): 8911–8919.
[30] Piteraa DJ, Paddonb CJ, Newman JD, et al. Balancing a heterologous mevalonate pathway for improved isoprenoid production in Escherichia coli. Metab Eng, 2007, 9(2): 193–207.
[31] Santos CNS, Stephanopoulos G. Combinatorial engineering of microbes for optimizing cellular phenotype. Curr Opin Chem Biol, 2008, 12: 168–176.
[32] Kim TY, Sohn SB, Kim HU, et al. Strategies for systems-level metabolic engineering. Biotechnol J, 2008, 3: 612–623.
Improving industrial microbial stress resistance by metabolic engineering: a review
Ruiyan Fu1, and Yin Li2
1 School of Tea and Food Science, Anhui Agricultural University, Hefei 230061, China
2 Institute of Microbiology, Chinese Academy of Sciences, Beijing 100101, China
Metabolic engineering is a technologic platform for industrial strain improvement and aims not only at modifying microbial metabolic fluxes, but also improving the physiological performance of industrial microbes. Microbes will meet multiple stresses in industrial processes. Consequently, elicited gene responses might result in a decrease in overall cell fitness and the efficiency of biotransformation. Thus, it is crucial to develop robust and productive microbial strains that can be integrated into industrial-scale bioprocesses. In this review, we focus on the progress of these novel methods and strategies for engineering stress-tolerance phenotypes referring to rational metabolic engineering and inverse metabolic engineering in recent years. In addition, we also address problems existing in this area and future research needs of microbial physiological functionality engineering.
metabolic engineering, industrial microbes, stress resistance, physiological functionality engineering
代谢工程是微生物菌种改造的平台技术,它通过修饰和优化微生物代谢网络与表达调控网络,改善细胞的生理功能,从而将微生物改造成“细胞工厂”生产有用的物质,是一个极具吸引力的研究热点。工业微生物的生理功能包括微生物的代谢能力、关键酶对终产物反馈抑制或阻遏的脱敏、恶劣环境下的鲁棒性 (Robustness)、对高浓度底物或产物的耐受性、生产全过程的适合性 (Fitness) 等[1]。以往的代谢工程研究主要集中在改造微生物的代谢能力方面,而增强生物技术过程的产率及生产能力需要的不仅是定向改变代谢流的方向并使通量最大化,微生物的其他生理功能特别是胁迫抗性也需要改造。环境胁迫是指环境中不利于生物生长的因素。在工业生产过程中,微生物细胞会面临多种因素的胁迫作用,包括酸胁迫[2]、高温胁迫[3]、渗透压胁迫[4]等。这些胁迫诱导的基因调节作用,都有可能影响细胞的许多重要生理功能,从而影响生物转化过程的效率。因此,从工业应用的角度出发,开发出生产性能良好、对发酵过程中的主要胁迫因素有高耐受性的超能菌株至关重要。
February 27, 2010; Accepted: April 29, 2010
Supported by: National Basic Research and Development Program of China (973 Program) (No. 2007CB707803), Foundation of Human Resource of Anhui Agricultural University.
Yin Li. Tel: +86-10-64807485; E-mail: yli@im.ac.cn
国家重点基础研究发展计划 (973计划) (No. 2007CB707803),安徽农业大学稳定和引进人才科研资助项目资助。
