亚硝酸盐氧化细菌研究进展与展望
2025-02-16胡露星蒋先军
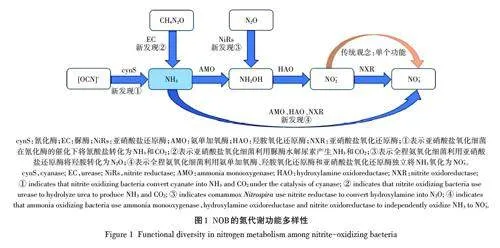
摘要:亚硝酸盐氧化细菌(Nitrite Oxidizing Bacteria,NOB)是全球氮循环过程中的关键微生物,主要功能是将亚硝酸盐氧化为硝酸盐。近十年来基于现代分子生态技术的发展,NOB的研究取得了很大的进展,主要包括:NOB的碳氮代谢途径及多样性远远超过以往的认知,如NOB不仅可以氧化亚硝酸盐,也可以氧化尿素、氨以及氰酸盐获取能量;丰富了NOB与环境因素的响应及生态位分化特征的研究,其中Nitrospira 更偏好于低氧的贫营养环境,中温的富营养环境更有利于Nitrobacter 的生存;对NOB Nitrospira与全程氨氧化细菌(Complete Ammonia Oxidizer,comammox Nitrospira)之间的联系与差异进行了初步探索,二者虽然同属于Nitrospira 谱系并且都具备亚硝酸盐氧化能力,但comammox Nitrospira 对亚硝酸盐的亲和力较低而对氨具有较高的亲和力,主要分布在低氨的寡营养生境,而NOB Nitrospira 对亚硝酸盐有较高亲和力,主要分布在低亚硝酸盐环境。本文基于上述最新研究进展进行了介绍与总结,并对该领域今后的研究进行了展望:在今后的研究中土壤环境或许可以成为NOB研究的新切入点;迫切需要探究缩短NOB分离和培养的方法并结合分子生态技术揭示NOB代谢多样性的实际依据以及NOB在碳氮循环中的真正贡献;探究NOB在代谢和生物化学方面的新机制,为揭示和预测NOB在环境中的响应提供理论支持。
关键词:亚硝酸盐氧化细菌;亚硝酸盐氧化;代谢多样性;全程氨氧化细菌;土壤
中图分类号:X172 文献标志码:A 文章编号:2095-6819(2025)01-0011-11 doi: 10.13254/j.jare.2023.0791
氮循环是生态系统中物质能量转化的重要组成部分,与生物代谢有着密不可分的关系[1]。而硝化作用是将氮的最低价态(NH3)转化为最高价态(NO-3)的唯一过程[2]。在经典的两步硝化理论中,硝化作用是一个两步过程:第一步氨氧化反应是限速步骤,由氨氧化古菌(Ammonia Oxidizing Archaea,AOA)和氨氧化细菌(Ammonia Oxidizing Bacteria,AOB)将NH3 氧化为NO-2[3-5];第二步亚硝酸盐氧化作用将NO-2 氧化成NO-3,由亚硝酸盐氧化细菌(Nitrite-Oxidizing Bacteria,NOB)催化[3,5]。传统的观念认为NOB代谢功能单一,对于NOB的代谢特点研究较少。事实上,NOB是全球氮循环过程中的关键微生物,在生态系统的氮素生物地球化学循环中具有重要作用[6]。亚硝酸盐的去向决定了被活化的氮是留在土壤里还是被释放到大气中,并且NOB的丰度和活性是影响土壤中氮素固持的重要生物因素,对于维持土壤肥力具有重要意义,所以NOB 在氮循环中的重要调节作用不容忽视[6-7]。在过去的几十年里,不断有新的氮循环过程和氮循环功能微生物被发现,使人们对全球氮循环的理解发生了革命性的变化[3,5]。在NOB的Nitrospira 谱系Ⅱ(Cluster Ⅱ)中,全程氨氧化细菌(Complete Am⁃monia Oxidizers,comammox Nitrospira)的发现,即单个生物可以单独完成将氨氧化为硝酸盐[3,5],打破了传统的两步硝化理论,使人们再次意识到NOB的物种多样性和生理代谢机制复杂性。近年来,学者获得了越来越多的Nitrospira 纯培养物或富集培养物[3,5,8-9],并发现了NOB类群中的部分菌株具备全新的代谢功能。近10 年来,新型生物检测技术的发展推动了NOB的研究进展,NOB生理生态功能单一、代谢受限的认知也被打破,NOB 的系统发育多样性和潜在的能量代谢功能得到了更多的重视。
1 NOB在氮循环的其他途径中具有代谢多样性
传统的研究认为NOB依赖亚硝酸获得能量并利用无机碳合成有机物[10],具有高度功能特异性,自身代谢多样性低,是严格的自养硝化微生物,而环境中的亚硝酸盐累积较少,因此对NOB的研究并未受到过多重视。2006 年和2008 年,Starkenburg 等[11-12]对Nitrobacter 基因组序列的分析揭示了NOB 的基因组组成,发现Nitrospira 类群存在多种能量代谢的功能基因序列,推动了NOB 代谢功能相关研究的发展。而comammox Nitrospira 的发现[3,5]更加验证了NOB的Nitrospira 类群在硝化过程中发挥的重要作用。
1.1 NOB具有尿素代谢活性
以往的研究认为亚硝酸盐是NOB 的唯一底物,维系着NOB的亚硝酸盐氧化能力[10]。而最近的研究却发现,NOB还可以利用尿素为底物。2015年,Koch等[8]分析了Nitrospira 谱系Ⅱ的代表菌群N. moscoviensis基因组,并在Nitrospira 基因组中发现了尿素转运蛋白和脲酶的编码基因[8]。进一步将N. moscoviensis 纯培养物置于含有1 mmol尿素的液体矿物培养基中孵育,几小时后培养基上清液中游离氨浓度的升高证实了N.moscoviensis 的尿素代谢活性[8]。NOB 对尿素的利用这一重要发现打开了硝化微生物生存策略新视角:具有尿素代谢活性的NOB反过来可以为氨氧化微生物提供底物来源,而氨氧化微生物又为NOB提供亚硝酸盐作为能量底物,维持了硝化微生物之间稳定的共生机制[8]。2017 年,Ushiki 等[13]对活性污泥中分离得到的Nitspira sp. ND1基因组序列分析也发现了脲酶活性位点。2020 年,Kitzinger 等[14]利用13CCO2和15N-尿素、15N-铵态氮、15N-氰酸盐及15N-亚硝态氮稳定同位素核酸探针技术和单细胞测序技术,结合宏基因组学和转录亚组学,监测了墨西哥湾海洋中的Nitrospinae 细胞对不同氮源的原位同化作用及细胞生长速率,结果证明Nitrospinae 主要利用铵和尿素来满足同化氮的需求,并将获得的能量主要用于自身的生长。NOB在氮代谢过程中的多样性证明其在地球氮循环过程中具有举足轻重的作用。作为Nitrospi⁃ra 中的一员,comammox Nitrospira 基因组中同样含有脲酶基因,因此可通过降解尿素获得氨[15],这与部分AOA 和AOB 功能相似[16]。但不同的是,comammoxNitrospira 具有多个不同的尿素转运体,除了高亲和力的尿素ABC 转运体[17],还有两个额外的尿素转运体——外膜孔蛋白(fmdC)和尿素羧化转运体(uctT),能够参与对极低浓度短链酰胺和尿素的吸收[18]。另外,comammox Nitrospira 基因组还包括ureABCDFG 脲酶操纵子和urtABCDE 尿素转运系统,表明comammoxNitrospira 具有对尿素的高亲和力摄取系统[19]。这种特性可支持comammox Nitrospira 在尿素浓度低或波动较大的环境中生长,例如森林土壤[20]、污水处理厂[21]和反应器[22]。2021年,Li等[23]利用尿液废水成功富集培养了三种包含完整尿素分解基因的comam⁃mox Nitrospira 富集物,并指出尿素可能是comammoxNitrospira 能够选择性富集的关键因素。然而,在分离的AOA 菌株N. viennensis[24]和N. gargensis[25]以及一些AOB菌株[26]中也发现了编码脲酶和尿素转运蛋白的基因。这些发现表明,comammox Nitrospira 将直接与AOA 和AOB 竞争环境中的尿素,并提高comammoxNitrospira 在氮代谢竞争中的优势。
NOB 对氰酸盐([OCN]-)的利用与尿素类似,N.gargensis 能够通过氧化氰酸盐为NH3 和CO2 获得能量,同时形成的NH3 也可以为氨氧化微生物提供底物[27]。2020年,Kitzinger等[14]在墨西哥湾的研究也表明,NOB中的Nitrospinae 更倾向于同化利用铵和尿素而不是亚硝酸盐,超过半数的Nitrospinae 细胞氮需量是来自尿素和氰酸盐。因此以尿素或氰酸盐为底物的生境有利于维持AOA/AOB-NOB共生体系[28]。
1.2 NOB在硝化过程中具有更广泛的代谢多样性
在硝化作用中,NOB 主要参与硝化作用中的第二个过程[4,27],即催化亚硝酸氧化为硝酸盐。研究发现归属于Nitrospira 谱系的comammox Nitrospira,不仅具有亚硝酸盐氧化能力和尿素代谢能力,在comammox Nitrospira 细胞内还包含了硝化过程所需的氨单加氧酶(Ammonia Monooxygenase,AMO)、羟胺氧化还原酶(Hydroxylamine Oxidoreductase,HAO)和亚硝酸盐氧化还原酶(Nitrite Oxidoreductase,NXR),能将NH3完全氧化成硝态氮[29]。2018年,Palomo等[30]对comammox Nitrospira 的基因组进行分析,并指出其关键代谢功能是进行全程硝化过程。为了验证comammox Nitrospira 在不同生态环境中对硝化作用的贡献,目前应用最多的研究方法是通过抑制剂法和稳定性同位素核酸探针技术(DNA/RNA-based StableIsotope Probing,DNA / RNA - SIP)测定comammoxNitrospira 的活性[29]。多个研究团队先后采用氯酸钾(KClO3)和辛烷(C8H14)的双抑制剂法评估了comammox Nitrospira 对旱地农田土壤和湿地沉积物氨氧化的贡献[31-33],其中KClO3主要是抑制亚硝酸氧化过程及硝酸盐还原过程,C8H14 则可以在不影响AOA活性的情况下抑制AOB中amoA 基因的活性,从而达到选择性抑制AOB 的效果[34]。但目前为止,KClO3 和C8H14 的双抑制剂法对comammox Nitrospira的抑制机理尚未得到很好的研究,因此该方法对测定comammox Nitrospira 在硝化作用的贡献仍存在局限性,需要进一步探究选择抑制剂以确定comammoxNitrospira 对硝化作用的贡献率。微宇宙培养试验结合DNA-SIP技术同样是验证微生物代谢活性的重要方法。2019 年,Wang 等[35]利用微宇宙培养试验结合13CO2 DNA-SIP技术证实了comammox clade B参与了森林和水稻土的硝化过程。2023年,Zheng等[36]利用13CO2 DNA-SIP技术证明了comammox Nitrospira 在三个典型污水处理厂的活性污泥中参与氨氧化,并且根据目前可用的代表性物种动力学参数量化了硝化微生物对硝化作用的相对贡献,结果表明comammoxNitrospira 对氨氧化的贡献是AOB的5∼106倍,这不仅证明了comammox Nitrospira 在生态环境中的普遍存在,更佐证了comammox Nitrospira 在实际环境中对底物NH3的直接利用。稳定性同位素核酸探针技术在探究活性硝化微生物中起到关键作用,其不仅包括DNA-SIP 还包括RNA-SIP,这两者对于鉴定关键活性微生物同样重要,而在大多数研究中只单独应用其中一种技术,DNA-SIP 技术可以检测微生物吸收同化的同位素,而RNA-SIP 则可以检测具有活性的潜在缓慢生长或非生长细胞。在今后的研究中若可以结合两种SIP技术并通过进一步的测序技术检测标记的微生物类群分类情况,将更有助于提高研究的分类分辨率并以更确凿的证据证明硝化过程中起关键作用的硝化微生物。
本文汇总了NOB的氮代谢功能多样性,见图1。传统观点认为,由于碳源不足,N2O主要在异养反硝化过程中产生[37],而大量研究证明,硝化作用除了产生硝态氮之外,还能够产生温室气体N2O[38-39]。自2016年以来,已有多个研究指出AOB 和AOA 是N2O的生产者[39-41],在comammox Nitrospira 基因组中同样包含了与N2O产生有关的HAO和NXR[3],而没有关于comammox Nitrospira 产生N2O 的明确报道。直到2019 年,Kits 等[42]报道comammox N.inopinata 可能通过非生物羟胺途径转化产生N2O,其释放的N2O水平与AOA 相当,但远低于AOB。N2O 的大量释放将影响全球气候变化,若是利用comammox Nitrospira 与AOA、AOB的竞争,在土壤和其他生态环境中采用更利于comammox Nitrospira 而不是AOB的生长条件,或许可以减少因硝化作用产生的N2O排放。因此在今后的研究中若能将comammox Nitrospira 耦合其他脱氮过程,或许可以在深度脱氮、降低成本、减少温室气体(N2O)排放等方面发挥重要作用。
2 NOB的碳代谢多样性
碳循环也是生态系统中最重要的物质循环之一,NOB在调控和驱动碳循环的过程中同样发挥着不可替代的作用[43]。以亚硝酸盐为唯一电子源,二氧化碳为唯一碳源,氧为终端电子受体,NOB能够进行自养碳固定过程[10]。
2.1 NOB具有多途径的固碳模式
基因组分析显示,Nitrobacter 利用卡尔文循环(Calvin Benson Bassham,CBB)进行碳固定,但Nitrospira 的固定途径未确定[11,44]。直到2012 年,Sorokin 等[45] 在研究中指出Nitrospira 中的Nitrospinagracilis 通过还原性三羧酸(Reverse Tricarboxylic AcidCycle,rTCA)循环进行碳固定。2016年,Daims等[4]在NOB 菌株基因组学和细胞生理生态研究中再次指出,NOB存在多种固碳模式,包括rTCA循环及CBB循环,其中Nitrospira、Nitrospina、Candidatus Nitromaritima以及新发现的comammox Nitrospira均利用微需氧的rTCA循环进行碳固定,而Nitrobacter、Nitrococcus 、Thiocapsa、Ca. Nitrotoga和Nitrolancea 则利用耐氧的CBB循环进行自养碳固定[12,45-47]。相比之下,rTCA循环进行碳固定消耗的ATP更少,能源利用效率更高[4]。不同的碳固定方式也反映出NOB进化的多元性。
而在深海中,化能自养微生物的固碳作用对全球海洋内部的碳循环和生态环境具有重要影响[48]。2017年,Pachiadaki等[49]通过单细胞组学和宏基因组学研究指出Nitrospinae 是海洋中的优势NOB,在深海化能自养过程中起重要作用,促进了海洋中的碳氮耦合。同一研究还指出包括Nitrospinae 和Nitrospirae 在内的多种NOB单扩增组装基因组都包含了三磷酸腺苷柠檬酸裂解酶(ATP Citrate Lyase,ACL)和其他指示参与rTCA碳固定的基因,表明这些NOB在海洋碳循环中可能发挥着重要作用[49]。研究者利用放射性溶解无机碳(14DIC)孵育培养结合显微放射自显影技术与催化报告沉积荧光原位杂交技术指出,Nitrospi⁃nae 主要依靠亚硝酸盐氧化产能固定了北大西洋西部中远海15%~45%的无机碳,在深海固碳过程中发挥了重要作用[49]。那么NOB如何参与土壤中的固碳过程、贡献率大小还需要进行进一步的探究。虽然该研究为探索NOB在固碳领域的研究提供了参考,但目前学者对于Nitrospinae 进行CO2 固定的贡献仍存在争议[14,22],主要还是因为缺乏具有代表性的富集培养物。
2.2 NOB的其他碳代谢多样性
Nitrobacter 并不是严格意义上的化能自养生物,在缺少亚硝酸盐时,Nitrobacter 可以利用甲酸盐或者丙酮酸进行碳同化维持生长[4]。从海洋生态系统和活性污泥中获得的一些Nitrospira 也可以利用丙酮酸、甲酸盐、甘油等简单有机碳源代谢产能[50-51]。在重建的Ca. N. defluvii完整基因组中也发现了编码代谢降解醋酸盐、丙酮酸盐和甲酸盐同化的途径[28]。2021 年,Bayer 等[52] 发现在海洋环境中的Nitrospiramarina Nb-295T能以甲酸为能源进行生长,并非完全的自养微生物。一氧化碳脱氢酶(Carbon MonoxideDehydrogenase,CODH)能够催化CO 氧化为CO2 的反应或其逆反应,在微生物的代谢途径中发挥关键作用[53]。2020年,Zhang等[48]在200 m和3 000 m深度海洋Nitrospina 的宏基因组中发现了大量编码CODH的基因转录本,在北太平洋和南太平洋的转录组中同样发现了CODH基因的高度表达,因此推测CO可以作为NOB的替代能源,支持NOB在海洋中的生存。
3 NOB在碳氮循环外的代谢多样性
研究发现Nitrospira moscoviensis 的生长不仅只依靠氨氧化或硝酸盐还原微生物提供的亚硝酸盐,同时还可以通过氧化氢来进行呼吸作用[54]。2020年,Park等[10]在海洋沉积物中得到的NOB富集物中发现了编码氢化酶(Hydrogenase,H2ase)的基因,这种酶具有氧化氢作为替代电子源的基因组潜力,可促进海洋NOB在厌氧条件下存活。并且以氧化氢气产能进行代谢生长所获得的能量(ΔG0=-237 kJ·mol-1 H2)要高于传统氧化亚硝酸盐体系所获得的能量(ΔG0=-74kJ·mol-1 NO-2)[54]。在生物膜的基底区、海洋沉积物、地下含水层、稻田、深海热液区沉积物和温泉等低氧生态位中,好氧氢气氧化可以提供额外的能量,并使至少一部分NOB独立于氨氧化或硝酸盐还原微生物提供的亚硝酸盐[54],有助于NOB 在环境中的广泛分布。2020年,Park等[10]在海洋中富集培养得到的两组NOB 中还发现了可能具有产生钴胺素(又称维生素B12)的遗传潜力,因此可能在海洋沉积物钴胺素的再循环中发挥重要作用。此外,comammox Nitrospira基因组还含有一个额外的胍丁胺酶(Agmatinase,AGM)基因,可以水解胍丁胺产生腐胺和尿素,这是comammox Nitrospira 区别于典型的硝化螺旋菌(Nitrospira spp.)系统的一个独特性质[55]。尤为重要的是,全程氨氧化细菌基因组中缺乏一种同化亚硝酸还原酶(nirA)或octaheme 细胞色素C(OctahemeCytochrome C,OCC)的基因[17],所以在以亚硝酸盐作为唯一氮源时,comammox Nitrospira 将无法作为NOB生长。而当comammox Nitrospira inopinata 以氨氧化微生物生长在适宜的环境时,亚硝酸盐可以在其细胞内累积,从而克服这种明显的缺陷。NOB 的代谢多样性使其能在缺乏亚硝酸盐期间维持NOB 种群生长,以稳定硝化作用并占据新的生态位。
4 NOB在环境中具有明确的生态位分化特征
对替代底物的代谢能力反映了NOB对缺乏氨和亚硝酸盐环境的生态适应,而这种对波动环境条件的适应和响应也推动了NOB在不同生态系统中的广泛分布[56]。目前大部分研究都集中于氨氧化过程的影响因素,而对亚硝酸盐氧化过程的影响因素研究较少,主要是因为NOB 纯菌分离培养的难度大,时间长,一般需要4~12年[28]。不同NOB之间存在基因组差异,可能使NOB 在环境中的生态位分化[2]。由于NOB生长速度对各种环境因素敏感[57],因此不同环境因子也显著影响着NOB的生态位分化。在NOB中,Nitrospira 和Nitrobacter 在环境中发挥着重要作用[58-59],目前对NOB 的研究也主要集中于Nitrobacter和Nitrospira[60-61]。
4.1 养分有效性是影响NOB分布的重要因子
目前的共识是,亚硝酸盐是NXR的首选底物,因此对亚硝酸盐的亲和力研究一直是NOB 的研究重点。NOB之间的生态位分化差异主要是因为对亚硝酸盐的亲和力不同,在环境中亚硝酸盐并不容易累积,而具有高亚硝酸盐亲和力的Nitrospira 适应于低亚硝酸盐浓度,这有助于Nitrospira 在自然生态系统中广泛分布[28]。与之相反的是,Nitrobacter 具有更低的底物亲和力,因此在富氮环境(接近腐烂的尸体、落叶后土壤、施肥土壤等)占主导地位时,丰度可能远远超过Nitrospira[60]。研究发现长期施用氮肥能显著提高土壤Nitrobacter 的丰度[62]。与单施无机肥(NPK)相比,施用有机无机混合肥更能显著提高土壤中Nitro⁃bacter 的丰度以及亚硝酸盐氧化势[7]。2018 年Han等[7]在对湖南亚热带地区的长期施肥实验点研究中发现长期施用有机无机混合肥能显著提高Nitrobacter的丰度,而Nitrospira 却显著受到施肥效应的抑制。然而,2020 年Ouyang 等[63]在美国犹他州的一个农用地中发现Nitrospira 是优势NOB,其占比大于97%。因此,施肥会对NOB 的群落结构产生影响,整体而言,富含氮素的环境可能更利于Nitrobacter 的生长,其丰度值远远超过Nitrospira。
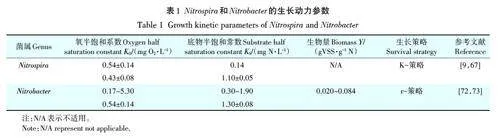
根据r/K选择理论,根据NOB生长速度和对底物的亲和力,其被划分为r-策略者和K-策略者。一般来说,r型微生物对养分输入反应迅速,在底物充足的环境中生长速度较快,但对底物亲和力较低,在与其他微生物竞争有限资源时可能处于劣势[64];K型微生物具有较高的底物亲和力,生长速率较慢,在面对有限的底物浓度时能更好地占据和维持生态位[62,64-65]。基于Nitrobacter 和Nitrospira 对亚硝酸盐的亲和力不同,Nitrospira 可能属于K-策略者[66],在很低的亚硝酸盐环境中也可以达到很高的数量。相比之下,Nitro⁃bacter 是r-策略者[32],喜欢更高的亚硝酸盐浓度并且比Nitrospira 生长更快[67]。然而,在2013 年,Ollivier等[68]就已经指出Nitrospira 与Nitrobacter 对氮素环境的偏好并非是绝对的,研究者在对施用粪肥后的草地研究中发现Nitrospira 更占优势,其丰度明显要高于Ni⁃trobacter,可能是由于施肥后土壤AOB丰度以及活性受到有机粪肥的抑制,导致了土壤中亚硝酸盐有效含量降低[68]。事实上,以往研究发现在亚硝酸盐浓度较低环境下,Nitrospira 比Nitrobacter 更具竞争优势,且Nitrospira 丰度通常与较低亚硝酸盐浓度呈正相关[69]。但是全程氨氧化细菌的纯培养物comammox Nitrospi⁃ra inopinata 对亚硝酸盐的亲和力较低,comammox Ni⁃trospira inopinata 亚硝酸盐半饱和常数与NOB 中的Nitrobacter 相当,却是传统NOB中Nitrospira 亚硝酸盐半饱和常数的15.7~49.9倍[70-71](表1)。
4.2 氧气浓度对NOB的活性产生重要影响
亚硝酸盐氧化作用是好氧过程,氧气浓度可在抑制或增强NOB的活性方面发挥重要作用[74],从而影响NOB 在生态系统中的生态位分化[75]。生物膜、沉积物、土壤以及海洋不同深度的氧梯度差异也可能为共存的NOB提供生态位分化条件。水稻生长过程中由淹水管理引起的氧浓度变化可能对NOB的丰度产生很大影响[6]。2013 年,Olliver 等[68] 在研究中发现Nitrobacter 更倾向于栖息在水稻根际,而土壤表面的Nitrospira 比根际更为丰富。在缺氧环境中观察到添加高于0.6 μmol·L−1的O2抑制了NO-2的氧化,这表明适应缺氧环境的NOB 主导了亚硝酸盐氧化过程[76]。研究者不仅在土壤中发现了NOB 对氧气的偏好差异,在海洋氧气最低区域的不同深度同样发现了NOB 的生态位分化[76],表明不同NOB 之间的O2 耐受性差异。Han 等[6]在油菜籽-水稻轮作试验中得出,Nitrospira 中相同系统发育谱系的个体之间生理特性同样存在差异。2015 年,Fitzgerald 等[77] 也在Nitrospira-NOB中发现偏好低溶氧的类群(代表纯菌为Nitrospira moscoviensis)和缺乏抗氧化防御机制酶的偏好高溶氧的类群( 代表纯菌为Nitrospiradefluvii),证明了Nitrospira 中不同菌种对氧有效性的不同偏好。在实际应用中,通过调节氧气含量可在缺氧条件下选择性抑制NOB,这是硝化反应器中调控AOB 与NOB 之间竞争以控制硝化作用的策略之一[78]。
对氧的亲和力及产氧能力可能是影响不同NOB之间生态位分化和竞争的重要原因[59]。NOB 中Ca.N. defluvii的亚氯酸盐歧化酶(Chlorite dismutase,Cld)已被证明可将亚氯酸盐还原为氯化物和O2,而研究同样指出水样中增加的溶解氧(Dissolved Oxygen,DO)能抑制Ca. N. defluvii 的活性[75]。因此需要进一步的研究来检测环境中产氧代谢对NOB代谢活性的影响[59]。此外,低氧水平被认为是comammox Nitrospi⁃ra 与其他硝化菌竞争的关键因素[79-80],而Li等[23]在DO浓度高于4 mg·L−1时实现了comammox Nitrospira 的富集。2020年,一项研究调查了14个污水处理厂,结果表明氧水平与comammox Nitrospira 丰度之间并没有明显的相关性[81],这些结果表明氧水平可能不是决定其他硝化微生物与comammox Nitrospira 之间竞争优势的关键因素。
4.3 温度是影响亚硝酸盐氧化过程的关键因素
温度在亚硝酸盐氧化的生物学过程中起着关键作用,温度变化会造成微生物的生理学变化,不仅影响细胞膜的流动性以及酶的活性,还会影响NOB的生长速度,改变自然生境中NOB的群落结构[57]。例如污水处理厂(WWTP)的关键NOB是Nitrospira 支系Ⅰ和Ⅱ[82],Nitrotoga 是工程系统[83] 中另一组重要的NOB,Nitrospira 能够在很宽的温度范围内(10~28 ℃)生长,在南海海洋约4~29.3 ℃的温度范围内发现了Nitrospira 的广泛分布,表明Nitrospira 对南海不同温度具有较好的适应性[84]。在南海深海区发现了与Ni⁃trospira 高度相关的OTU,其相对丰度与温度呈负相关,意味着南海中可能存在未知的耐低温Nitrospira新物种[84]。
不同NOB 对温度的偏好也有差异,富集培养的Nitrotoga 菌株最适温度和pH值分别为22 ℃和6.8,而Nitrospira defluvii(来自WWTP的Nitrospira 谱系Ⅰ)在32 ℃和pH 7.3下生长最好[85]。在温度为15~30 ℃时,Nitrobacter 的活性随着温度的增加而增加[57]。在污水处理厂中,季节性的温度变化也为适应中温环境的Nitrospira 和偏好低温的Nitrotoga 提供了共存条件[4]。2021 年,Sun 等[57]阐述了Nitrobacter 中N. wiogradskyi和N. hamburgensis 的共培养物对亚硝酸盐氧化动力学与温度的关系,研究指出培养物的最大活性(rmax)为93.58 mmol·L-1 ·g-1 ·d-1,生长速率常数(μmax)为1.311 d-1,最适温度为30 ℃。该研究还指出共培养物的活性和亚硝酸盐氧化能力对温度敏感,当温度为35 ℃时,共培养物的亚硝酸盐氧化能力丧失,并且温度对共培养物的影响是不可逆的,在温度恢复至25 ℃后仍无法恢复共培养物的亚硝酸盐氧化能力[57]。2013年一项Meta分析的结果表明,温度对草地土壤的氮循环过程影响并不明显[86]。2020年,Séneca等[87]在奥地利的一个高寒草原长期气候变化实验站开展的研究同样支持这一观点,研究指出温度改变并不能影响土壤氨转化过程,温度升高后土壤中氮相关微生物的丰度、组成和活性并未受到影响。以上研究结果表明不同生态系统中的硝化微生物对温度的敏感程度仍存在差异,还需后续进一步的研究探究不同NOB菌群对温度的偏好。
4.4 pH是影响NOB富集组成的关键因素
pH也会显著影响硝化微生物的活性以及硝化作用的强弱,同时pH值是调控NOB富集的重要因素[66]。pH值从根本上影响氨氧化微生物的底物NH3与NH+4的平衡,进而影响亚硝酸盐氧化过程的底物有效性,最终影响NOB[88-89],甚至pH还可能会对NOB细胞产生毒害作用[89]。与氨氧化细菌AOB相比,NOB对pH的敏感性似乎更高。研究发现NOB在中性或微偏碱性的环境中具有更高的活性,Nitrospira 最适生长pH为7.6~8.3[66],Nitrobacter 最适生长pH 为7.6~8.2[90],因此在过酸或者过碱的环境下亚硝酸盐氧化作用均会明显减弱[62]。前人的研究一直未找到严格嗜酸NOB,2006年,Hawkins等[91]在长期施用有机肥的红壤中,发现了对pH值适应范围更广(4.83~6.54)的NOB类群,极大地拓宽了人们对亚硝酸盐氧化作用在酸性环境下的理解。直到2012 年,Kuang 等[92]在一些强酸性(pH=2.6±0.45)的环境下也仍然发现有大量Nitrospira(Leptospirillum spp.)的分布,说明在Nitrospira 种群内也可能存在对pH有不同适应偏好的菌种。因此,在今后的研究中应该考虑到嗜酸或耐酸NOB存在的可能性。Liang等[93]在为期四年的田间试验土壤样品中发现土壤pH 值也是影响Nitrospira 群落结构的主要驱动因素。2021年,王智慧等[94]在三种pH紫色土中发现不同NOB 类群之间的生态位分化,其中Nitrospira在中性紫色土中丰度最高,而Nitrobacter 则偏好石灰性紫色土生境,验证了在土壤环境中NOB对不同pH的偏好策略。
目前的研究多通过人为控制单一环境变量来探讨单一因素对NOB的影响,而在实际环境中,各种环境因子之间也有着密不可分的联系。同时土壤环境复杂多变,在土壤中究竟是单一因素还是不同环境因子之间的综合作用(如拮抗作用或协同作用等)影响亚硝酸盐氧化过程尚不能做出一个明确的判断。在今后的研究中,还需要进行更进一步探索实际原位土壤环境中多个变量对NOB菌群影响的交叉研究。
5 comammox Nitrospira 与NOB Nitrospira 的生态位分化
虽然comammox Nitrospira 属于NOB 中的Nitrospira 谱系[3,5],但两者在环境中可能具有明确的生态位分化。comammox Nitrospira 同时具有AMO和NXR编码基因[3,5],既可以发挥AOB的氨氧化功能也可以完成NOB 在亚硝酸盐氧化过程中的作用。与NOB Nitrospira 相比较,comammox Nitrospira 在硝化过程中可以获得更多的能量,而代价则是生长速率缓慢[3]。这些生理和代谢的差异意味着comammoxNitrospira 可能在不同的环境中占据优势,体现出生态位分化的特征。同时,硝化微生物之间又存在一种共存的关系:NOB 依赖于氨氧化微生物产生的亚硝酸盐,而氨氧化微生物AOA、AOB 和comammoxNitrospira 同样又得益于NOB 对亚硝态氮的解毒,因此硝化微生物之间是一种互利共生的关系,在同一环境中出现时会产生生态位分化以减少资源竞争[95]。2020年,Li等[96]研究指出comammox Nitrospira 与NOB对于长期施肥响应一致,有机肥的施用促进了comammox Nitrospira、Nitrobacter 和Nitrospira 群落之间的耦合联系,提高了土壤硝化速率。comammoxNitrospira inopinata 以亚硝酸盐为底物时的基质比利用率相较于NOB(Nitrospira 和Nitrobacter)要低1~2个数量级[70-71,97-98],因此推测当comammox Nitrospirainopinata 进行亚硝酸盐氧化过程时,其丰度至少为NOB的8.6倍才能与NOB竞争并在亚硝酸盐氧化过程中发挥主要作用[98]。基于comammox Nitrospira 在氮循环过程中功能多样性,究竟是哪些因素保持comammox Nitrospira 的氨氧化速率和亚硝酸盐氧化速率之间的平衡尚不清楚。今后的研究可以更具体地探究在何种条件下comammox Nitrospira 是作为氨氧化微生物进行活动,又是在何种条件下comammoxNitrospira 与NOB协同完成亚硝酸盐氧化过程。
6 结论与展望
本文旨在强调亚硝酸盐氧化细菌的代谢多样性及NOB 对不同环境的偏好性。传统意义上广泛认为NOB的代谢功能单一,本文总结了NOB对一些替代能源的代谢能力,而对NOB潜在的新代谢能力预测和探究还需要进行进一步的探索。亚硝酸盐氧化细菌具有进行亚硝酸盐氧化作用和碳固定能力,可建立氮碳循环之间的桥梁,同时也表明了碳氮循环之间以复杂的方式交织在一起,并不是两个完全独立的过程。
(1)目前关于NOB 代谢能力的研究大多数都出自海洋环境,因此土壤环境或许可成为今后NOB研究的新切入点,为揭示NOB的作用机制和生态功能提供更多依据。
(2)纯培养菌株对于确定NOB 的生理生化特点以及理解NOB的各类碳氮代谢运作机制至关重要。但是亚硝酸盐氧化细菌的分离与纯培养难度更高(NOB纯菌分离培养需要4~12年),目前仍缺乏可以系统表征NOB的纯培养菌株,因此对研究中所描述的NOB生态生理分析仍存在阻碍。因此迫切需要探究缩短NOB的分离和培养方法,并结合分子生态技术揭示NOB代谢多样性的实际依据以及NOB在碳氮循环中的真正贡献。
(3)新型硝化微生物comammox Nitrospira 的发现同样带来了很多未解决的问题,comammox Nitrospira具有氨氧化和亚硝酸盐氧化的双重功能,目前尚不清楚comammox Nitrospira 能否在氨氧化和亚硝酸盐氧化两种模式之间切换,功能切换的条件及原因又是什么?今后的研究应该关注于探究在何种条件下comammoxNitrospira 是作为氨氧化微生物进行活动,又是在何种条件下与NOB协同完成亚硝酸盐氧化过程。
(4)与实验室可进行精密培养条件调控相比,环境中的NOB暴露于复杂变化的条件中,其中底物浓度、含氧量、温度、pH等条件会对NOB的生长和活性产生较大影响。而在自然环境中NOB如何调节自身以应对环境变化及其生存和繁衍需要依靠哪些类型的代谢仍存在许多知识空白。关于亚硝酸盐氧化细菌在代谢和生物化学方面的新机制理解,如关键酶的活性及调节、亚硝酸盐氧化细菌的结构和功能以及新的代谢途径的发现,对于揭示和预测亚硝酸盐氧化细菌在环境中的响应,以及环境因子如何共同影响环境中的硝化速率等都有很大的帮助。
参考文献:
[1] KUYPERS M M, MARVHANT H K, KARTAL B. The microbial
nitrogen-cycling network[J]. Nature Reviews Microbiology, 2018, 16
(5):263-276.
[2] 石秀丽, 郭萌萌, 张莹, 等. 单步硝化作用与全程氨氧化微生物研究
进展[J]. 草业学报, 2018, 27(7):196-203. SHI X L, GUO M M,
ZHANG Y, et al. Progress in understanding of one-step nitrification
(complete ammonia oxidizing) microorganisms:a review[J]. Acta
Prataculturae Sinica, 2018, 27(7):196-203.
[3] DAIMS H, LEBEDEVA E V, PJEVAC P, et al. Complete nitrification
by Nitrospira bacteria[J]. Nature, 2015, 528(7583):504-509.
[4] DAIMS H, LÜCKER S, WAGNER M. A new perspective on microbes
formerly known as nitrite-oxidizing bacteria[J]. Trends in Microbiology,
2016, 24(9):699-712.
[5] VAN K M, SPETH D R, ALBERTSEN M, et al. Complete nitrification
by a single microorganism[J]. Nature, 2015, 528(7583):555-559.
[6] HAN S, LUO X S, LIAO H, et al. Nitrospira are more sensitive than
Nitrobacter to land management in acid, fertilized soils of a rapeseedrice
rotation field trial[J]. Science of the Total Environment, 2017, 599/
600:135-144.
[7] HAN S, ZENG L Y, LUO X S, et al. Shifts in Nitrobacter - and
Nitrospira-like nitrite-oxidizing bacterial communities under longterm
fertilization practices[J]. Science of the Total Environment, 2018,
124(9):118-125.
[8] KOCH H, LÜCKER S, ALBERTSEN M, et al. Expanded metabolic
versatility of ubiquitous nitrite-oxidizing bacteria from the genus
Nitrospira[J]. The Proceedings of the National Academy of Sciences,
2015, 112(36):11371-11376.
[9] 姚倩, 彭党聪, 赵俏迪, 等. 活性污泥中硝化螺菌(Nitrospira)的富集
及其动力学参数[J]. 环境科学, 2017, 38(12):5201-5207. YAO Q,
PENG D C, ZHAO Q D, et al. Enrichment of Nitrospira in activated
sludge and kinetic characterization[J]. Environmental Science, 2017, 38
(12):5201-5207.
[10] PARK S J, ANDREI A S, BULZU P A, et al. Expanded diversity and
metabolic versatility of marine nitrite-oxidizing bacteria revealed by
cultivation - and genomics-based approaches[J]. Applied and
Environmental Microbiology, 2020, 86(22):e1620-1667.
[11] STARKENBURG S R, CHAIN P S G, SAYAVEDRA-SOTO L A, et al.
Genome sequence of the chemolithoautotrophic nitrite-oxidizing
bacterium Nitrobacter winogradskyi Nb-255[J]. Applied and
Environmental Microbiology, 2006, 72(3):2050-2063.
[12] STARKENBURG S R, LARIMER F W, STEIN L Y, et al. Complete
genome sequence of Nitrobacter hamburgensis X14 and comparative
genomic analysis of species within the genus Nitrobacter[J]. Applied
and Environmental Microbiology, 2008, 74(9):2852-2863.
[13] USHIKI N, FUJITANI H, SHIMADA Y, et al. Genomic analysis of
two phylogenetically distinct Nitrospira species reveals their genomic
plasticity and functional diversity[J]. Frontiers in Microbiology, 2017,
8(1):2637-2649.
[14] KITZINGER K, MARVHANT H K, BRISTOW L A, et al. Single cell
analyses reveal contrasting life strategies of the two main nitrifiers in
the ocean[J]. Nature Communications, 2020, 11(1):767-779.
[15] KOCH H, VAN K M, LÜCKER S. Complete nitrification:insights into
the ecophysiology of comammox Nitrospira[J]. Applied Microbiology
and Biotechnology, 2019, 103(1):177-189.
[16] KITZINGER K, PADILLA C C, MARVHANT H K, et al. Cyanate and
urea are substrates for nitrification by Thaumarchaeota in the marine
environment[J]. Nature Microbiology, 2019, 4(2):234-243.
[17] LI S W, JIANG X J, WANG X L, et al. Tillage effects on soil
nitrification and the dynamic changes in nitrifying microorganisms in
a subtropical rice-based ecosystem:a long-term field study[J]. Soil
and Tillage Research, 2015, 150(7):132-138.
[18] MILLS J, GREENWOOD J A, WYBORN N R, et al. An outermembrane
porin inducible by short-chain amides and urea in the
methylotrophic bacterium Methylophilus methylotrophus[J].
Microbiology, 1997, 143(7):2373-2379.
[19] CAMEJO P Y, DOMINGO J S, MCMAHON K D, et al. Genomeenabled
insights into the ecophysiology of the comammox bacterium
“Candidatus Nitrospira nitrosa”[J]. Msystems, 2017, 2(5):e00059-
17.
[20] LI C Y, HU H W, CHEN Q L, et al. Niche differentiation of clade A
comammox Nitrospira and canonical ammonia oxidizers in selected
forest soils[J]. Soil Biology and Biochemistry, 2020, 149(10):
107925.
[21] HU H W, HE J Z. Comammox:a newly discovered nitrification
process in the terrestrial nitrogen cycle[J]. Journal of Soils and
Sediments, 2017, 17(12):2709-2717.
[22] ZHAO Y X, HU J J, YANG W L, et al. The long-term effects of using
nitrite and urea on the enrichment of comammox bacteria[J]. Science
of the Total Environment, 2021, 755(7):246-252.
[23] LI J Y, HUA Z S, LIU T, et al. Selective enrichment and metagenomic
analysis of three novel comammox Nitrospira in a urine-fed
membrane bioreactor[J]. ISME Communications, 2021, 1(1):7.
[24] TOURNA M, STIEGLMEIER M, SPANG A, et al. Nitrososphaera
viennensis, an ammonia oxidizing archaeon from soil[J]. The
Proceedings of the National Academy of Sciences, 2011, 108(20):
8420-8425.
[25] SPANG A, POEHLEIN A, OFFRE P, et al. The genome of the
ammonia-oxidizing Candidatus Nitrososphaera gargensis:insights
into metabolic versatility and environmental adaptations[J].
Environmental Microbiology, 2012, 14(12):3122-3145.
[26] BURTON S A Q, PROSSER J I. Autotrophic ammonia oxidation at
low pH through urea hydrolysis[J]. Applied and Environmental
Microbiology, 2001, 67(7):2952-2957.
[27] LÜCKER S, WAGNER M, MAIXNER F, et al. A Nitrospira
metagenome illuminates the physiology and evolution of globally
important nitrite-oxidizing bacteria[J]. The Proceedings of the
National Academy of Sciences, 2010, 107(30):13479-13484.
[28] PALATINSZKY M, HERBOLD C W, JEHMLICH N, et al. Cyanate as
energy source for nitrifiers[J]. Nature, 2015, 524(7523):105-108.
[29] ZHU G B, WANG X M, WANG S Y, et al. Towards a more laborsaving
way in microbial ammonium oxidation:a review on complete
ammonia oxidization (comammox) [J]. Science of the Total
Environment, 2022, 829(33):154590.
[30] PALOMO A, PEDERSEN A G, FOWLER S J, et al. Comparative
genomics sheds light on niche differentiation and the evolutionary
history of comammox Nitrospira[J]. The ISME Journal, 2018, 12(7):
1779-1793.
[31] WANG S Y, ZHU G B, ZHUANG L J, et al. Anaerobic ammonium
oxidation is a major N-sink in aquifer systems around the world[J].
The ISME Journal, 2020, 14(1):151-163.
[32] WANG X M, WANG S Y, JIANG Y Y, et al. Comammox bacterial
abundance, activity, and contribution in agricultural rhizosphere soils
[J]. Science of the Total Environment, 2020, 727(30):138563.
[33] WANG D Q, ZHOU C H, NIE M, et al. Abundance and niche
specificity of different types of complete ammonia oxidizers
(comammox)in salt marshes covered by different plants[J]. Science of
the Total Environment, 2021, 768(19):144993.
[34] TAYLOR A E, VAJRALA N, GIGUERE A T, et al. Use of aliphatic
n-Alkynes to discriminate soil nitrification activities of ammoniaoxidizing
Thaumarchaea and bacteria[J]. Applied and Environmental
Microbiology, 2013, 79:6544-6551.
[35] WANG Z H, CAO Y Q, ZHU-BARKER X, et al. Comammox
Nitrospira clade B contributes to nitrification in soil[J]. Soil Biology
and Biochemistry, 2019, 135(8):392-395.
[36] ZHENG M S, TIAN Z C, CHAI Z M, et al. Ubiquitous occurrence and
functional dominance of comammox Nitrospira in full-scale
wastewater treatment plants[J]. Water Research, 2023, 236(10):
119931.
[37] VILAIN G, GARNIER J, DÉCUQ C, et al. Nitrous oxide production
from soil experiments:denitrification prevails over nitrification[J].
Nutrient Cycling in Agroecosystems, 2014, 98(2):169-186.
[38] SHI X Z, HU H W, ZHU-BARKER X, et al. Nitrifier-induced
denitrification is an important source of soil nitrous oxide and can be
inhibited by a nitrification inhibitor 3, 4-dimethylpyrazole phosphate
[J]. Environmental Microbiology, 2017, 19(12):4851-4865.
[39] WRAGE-MÖNNIG N, HORN M A, WELL R, et al. The role of
nitrifier denitrification in the production of nitrous oxide revisited[J].
Soil Biology and Biochemistry, 2018, 123(8):A3-A16.
[40] KLEINER M, MOIR J, PEREZ J, et al. Comparison of nitrogen oxide
metabolism among diverse ammonia-oxidizing bacteria[J]. Frontiers
in Microbiology, 2016, 12(7):1-9.
[41] LIU S R, HAN P, HINK L, et al. Abiotic conversion of extracellular
NH2OH contributes to N2O emission during ammonia oxidation[J].
Environmental Science and Technology, 2017, 51(22):13122-13132.
[42] KITS K D, JUNG M, VIERHEILIG J, et al. Low yield and abiotic
origin of N2O formed by the complete nitrifier Nitrospira inopinata[J].
Nature Communication, 2019, 10(1):1836.
[43] WU H W, CUI H L, FU C X, et al. Unveiling the crucial role of soil
microorganisms in carbon cycling:a review[J]. Science of the Total
Environment, 2024, 909(4):168627.
[44] SPIECK E, HARTWIG C, MCCORMACK I, et al. Selective
enrichment and molecular characterization of a previously uncultured
Nitrospira-like bacterium from activated sludge[J]. Environmental
Microbiology, 2006, 8(3):405-415.
[45] SOROKIN D Y, LÜCKER S, VEJMELKOVA D K, et al. Nitrification
expanded:discovery, physiology and genomics of a nitrite-oxidizing
bacterium from the phylum Chloroflexi[J]. The ISME Journal, 2012, 6
(12):2245-2256.
[46] BODDICKER A M, MOSIER A C. Genomic profiling of four
cultivated Candidatus Nitrotoga spp. predicts broad metabolic
potential and environmental distribution[J]. The ISME Journal, 2018,
12(12):2864-2882.
[47] HEMP J, LÜCKER S, SCHOTT J, et al. Genomics of a phototrophic
nitrite oxidizer:insights into the evolution of photosynthesis and
nitrification[J]. The ISME Journal, 2016, 10(11):2669-2678.
[48] ZHANG Y, QIN W, HOU L, et al. Nitrifier adaptation to low energy
flux controls inventory of reduced nitrogen in the dark ocean[J]. The
Proceedings of the National Academy of Sciences, 2020, 117(9):
4823-4830.
[49] PACHIADAKI M G, SINTES E, BERGAUER K, et al. Major role of
nitrite-oxidizing bacteria in dark ocean carbon fixation[J]. Science,
2017, 358(6366):1046-1051.
[50] DAIMS H, NIELSEN J, NIELSEN P, et al. In situ characterization of
Nitrospira-like nitrite-oxidizing bacteria active in wastewater
treatment plants[J]. Applied and Environmental Microbiology, 2001, 67
(11):5273-5284.
[51] GRUBER-DORNINGER C, PESTER M, KITZINGER K, et al.
Functionally relevant diversity of closely related Nitrospira in
activated sludge[J]. The ISME Journal, 2015, 9(3):643-655.
[52] BAYER B, SAITO M A, MCILVIN M R, et al. Metabolic versatility of
the nitrite-oxidizing bacterium Nitrospira marina and its proteomic
response to oxygen-limited conditions[J]. The ISME Journal, 2021, 15
(4):1025-1039.
[53] KING G M, WEBER C F. Distribution, diversity and ecology of
aerobic CO-oxidizing bacteria[J]. Nature Reviews Microbiology, 2007,
5(2):107-118.
[54] KOCH H, GALUSHKO A S, ALBERTSEN M, et al. Growth of nitriteoxidizing
bacteria by aerobic hydrogen oxidation[J]. Science, 2014,
345(6200):1052-1054.
[55] JIA Z J, CONRAD R. Bacteria rather than Archaea dominate
microbial ammonia oxidation in an agricultural soil[J]. Environmental
Microbiology, 2009, 11(7):1658-1671.
[56] WRIGHT C L, LEHTOVIRTA-MORLEY L E. Nitrification and
beyond:metabolic versatility of ammonia oxidizing archaea[J]. The
ISME Journal, 2023, 17(9):1358-1368.
[57] SUN H W, ZHANG H, SHI W Y, et al. Temperature affects the
oxidation kinetics of co-cultured Nitrobacter winogradskyi and
Nitrobacter hamburgensis in nitrite-oxidizing bioreactor[J]. Journal of
Environmental Chemical Engineering, 2021, 9(5):106262.
[58] BARTOSCH S, HARTWIG C, SPIECK E, et al. Immunological
detection of Nitrospira-like bacteria in various soils[J]. Microbial
Ecology, 2002, 43(1):26-33.
[59] KIM D, KIM S. Effect of nitrite concentration on the distribution and
competition of nitrite-oxidizing bacteria in nitratation reactor systems
and their kinetic characteristics[J]. Water Research, 2006, 40(5):
887-894.
[60] ATTARD E, POLY F, COMMEAUX C, et al. Shifts between
Nitrospira - and Nitrobacter-like nitrite oxidizers underlie the
response of soil potential nitrite oxidation to changes in tillage
practices[J]. Environmental Microbiology, 2010, 12(2):315-326.
[61] WANG B Z, ZHAO J, GUO Z Y, et al. Differential contributions of
ammonia oxidizers and nitrite oxidizers to nitrification in four paddy
soils[J]. The ISME Journal, 2015, 9(5):1062-1075.
[62] MA W, JIANG S J, ASSEMIEN F, et al. Response of microbial
functional groups involved in soil N cycle to N, P and NP fertilization
in Tibetan alpine meadows[J]. Science of the Total Environment, 2016,
101(10):195-206.
[63] OUYANG Y, NORTON J. Nitrite oxidizer activity and community are
more responsive than their abundance to ammonium-based fertilizer
in an agricultural soil[J]. Frontiers in Microbiology, 2020, 11:1-10.
[64] YANG Y, DOU Y X, WANG B R, et al. Deciphering factors driving
soil microbial life-history strategies in restored grasslands[J]. iMeta,
2023, 2(1):1-22.
[65] GILBERT E M, AGRAWAL S, BRUNNER F, et al. Response of
different Nitrospira species to anoxic periods depends on operational
DO[J]. Environmental Science and Technology, 2014, 48(5):2934-
2941.
[66] BLACKBURNE R, VADIVELU V M, YUAN Z, et al. Kinetic
characterisation of an enriched Nitrospira culture with comparison to
Nitrobacter[J]. Water Research, 2007, 41(14):3033-3042.
[67] SCHRAMM A, BEER D D, HEUVEL J, et al. Microscale distribution
of populations and activities of Nitrosospira and Nitrospira spp. along
a macroscale gradient in a nitrifying bioreactor:quantification by in
situ hybridization and the use of microsensors[J]. Applied and
Environmental Microbiology, 1999, 65(8):3690-3696.
[68] OLLIVIER J, SCHACHT D, KINDLER R, et al. Effects of repeated
application of sulfadiazine-contaminated pig manure on the
abundance and diversity of ammonia and nitrite oxidizers in the rootrhizosphere
complex of pasture plants under field conditions[J].
Frontiers in Microbiology, 2013, 4:22.
[69] ROUX X, NIBOYET A, TEREDA A, et al. Predicting the responses of
soil nitrite-oxidizers to multi-factorial global change:a trait-based
approach[J]. Frontiers in Microbiology, 2016, 17(7):628.
[70] KITS K D, SEDLACEK C J, LEBEDEVA E V, et al. Kinetic analysis
of a complete nitrifier reveals an oligotrophic lifestyle[J]. Nature,
2017, 549(7671):269-272.
[71] NOWKA B, DAIMS H, SPIECK E. Comparison of oxidation kinetics
of nitrite-oxidizing bacteria:nitrite availability as a key factor in
niche differentiation[J]. Applied and Environmental Microbiology,
2014, 81(2):745-753.
[72] BECCARI M, MARANI D, RAMADORI R. A critical analysis of
nitrification alternatives[J]. Water Research, 1979, 13(2):185-192.
[73] 于雪, 孙洪伟, 李维维, 等. 温度对硝化杆菌(Nitrobacter)活性动力
学影响[J]. 环境科学, 2019, 40(3):1426-1430. YU X, SUN H W,
LI W W, et al. Effect of temperature on the activity kinetics of
Nitrobacter[J]. Environmental Science, 2019, 40(3):1426-1430.
[74] MEHRANI M J, SOBOTKA D, KOWAL P, et al. The occurrence and
role of Nitrospira in nitrogen removal systems[J]. Bioresource
Technology, 2020, 303(9):122936.
[75] HONG Y G, WU J P, JIAO L J, et al. Shifts in the abundance and
community composition of particle-associated and free-living
Nitrospira across physicochemical gradients in the Pearl River estuary
[J]. Estuaries and Coasts, 2021, 44(7):1931-1945.
[76] SUN X, FREY C, GARCIA-ROBLEDO E, et al. Microbial niche
differentiation explains nitrite oxidation in marine oxygen minimum
zones[J]. The ISME Journal, 2021, 15(5):1317-1329.
[77] FITZGERALD C M, CAMEJO P, OSHLAG J Z, et al. Ammoniaoxidizing
microbial communities in reactors with efficient nitrification
at low-dissolved oxygen[J]. Water Research, 2015, 70(3):38-51.
[78] AKABOCI T R V, GICH F, RUSCALLEDA M, et al. Assessment of
operational conditions towards mainstream partial nitritationanammox
stability at moderate to low temperature: reactor
performance and bacterial community[J]. Chemical Engineering
Journal, 2018, 350(20):192-220.
[79] KEENE-BEACH N, NOGUERA D R. Design and assessment of
species-level qPCR primers targeting comammox[J]. Frontiers in
Microbiology, 2018, 10:36.
[80] ROOTS P, WANG Y B, ROSENTHAL A F, et al. Comammox
Nitrospira are the dominant ammonia oxidizers in a mainstream low
dissolved oxygen nitrification reactor[J]. Water Research, 2019, 157
(10):396-405.
[81] COTTO I, DAI Z H, HUO L X, et al. Long solids retention times and
attached growth phase favor prevalence of comammox bacteria in
nitrogen removal systems[J]. Water Research, 2020, 169(2):115268.
[82] AOI Y. In situ identification of microorganisms in biofilm communities
[J]. Journal of Bioscience and Bioengineering, 2002, 94(6):552-556.
[83] KRUSE M, ZUMBRÄGEL S, BAKKER E, et al. The nitrite-oxidizing
community in activated sludge from a municipal wastewater treatment
plant determined by fatty acid methyl ester-stable isotope probing[J].
Systematic and Applied Microbiology, 2013, 36(7):517-524.
[84] SUN W, JIAO L J, WU J P, et al. Existence and distribution of novel
phylotypes of Nitrospira in water columns of the South China Sea[J].
iScience, 2022, 25(9):104895.
[85] HÜPEDEN J, WEGEN S, OFF S, et al. Relative abundance of
Nitrotoga in a biofilter of a cold freshwater aquaculture plant appears
to be stimulated by a slightly acidic pH-value[J]. Applied and
Environmental Microbiology, 2016, 82(6):3115-3163.
[86] BAI E, LI S L, XU W H, et al. A meta-analysis of experimental
warming effects on terrestrial nitrogen pools and dynamics[J]. The
New Phytologist, 2013, 199(2):441-451.
[87] SÉNECA J, PJEVAC P, CANARINI A, et al. Composition and activity
of nitrifier communities in soil are unresponsive to elevated
temperature and CO2, but strongly affected by drought[J]. The ISME
Journal, 2020, 14(12):3038-3053.
[88] GUBRY-RANGIN C, HAI B, QUINCE C, et al. Niche specialization
of terrestrial archaeal ammonia oxidizers[J]. The Proceedings of the
National Academy of Sciences, 2011, 108(52):21206-21211.
[89] LEHTOVIRTA-MORLEY L E, SAYAVEDRA-SOTO L A, GALLOIS
N, et al. Identifying potential mechanisms enabling acidophily in the
ammonia-oxidizing archaeon“Candidatus Nitrosotalea devanaterra”
[J]. Applied and Environmental Microbiology, 2016, 82(9):2608-
2619.
[90] GRUNDITZ C, DALHAMMAR G. Development of nitrification
inhibition assays using pure cultures of Nitrosomonas and Nitrobacter
[J]. Water Research, 2001, 35(2):433-440.
[91] HAWKINS S A, ROBINSON K G, LAYTON A C, et al. A comparison
of ribosomal gene and transcript abundance during high and low
nitrite oxidizing activity using a newly designed real-time PCR
detection system targeting the Nitrobacter spp. 16S-23S intergenic
spacer region[J]. Environmental Engineering Science, 2006, 23(3):
521-532.
[92] KUANG J, HUANG L, CHEN L, et al. Contemporary environmental
variation determines microbial diversity patterns in acid mine
drainage[J]. The ISME Journal, 2013, 7(5):1038-1050.
[93] LIANG F, WEN Y K, DONG X, et al. Response of activity and
community composition of nitrite-oxidizing bacteria to partial
substitution of chemical fertilizer by organic fertilizer[J].
Environmental Science and Pollution Research, 2021, 28(23):29332-
29343.
[94] 王智慧, 蒋先军. 宏基因组技术研究泥岩母质发育的三种不同pH
紫色土硝化微生物群落演变规律[J]. 微生物学报, 2021, 61(7):
1933 - 1944. WANG Z H, JIANG X J. Metagenomics - centric
assembly of nitrifying communities in three pH distinct soils with the
same origin of parent material[J]. Acta Microbiologica Sinica, 2021, 61
(7):1933-1944.
[95] SILVERTOWN J. The ghost of competition past in the phylogeny of
island endemic plants[J]. Journal of Ecology, 2004, 92(1):168-173.
[96] LI X, HAN S, WAN W J, et al. Manure fertilizes alter the nitrite
oxidizer and comammox community composition and increase
nitrification rates[J]. Soil and Tillage Research, 2020, 204(9):
104701.
[97] LAWSON C E, LÜCKER S. Complete ammonia oxidation:an
important control on nitrification in engineered ecosystems? [J].
Current Opinion in Biotechnology, 2018, 50(2):158-165.
[98] 许少怡, 肖锐, 柴文波, 等. 全程硝化菌微生物学特性及在水处理
领域的应用潜力[J]. 微生物学报, 2021, 61(2):315-332. XU S Y,
XIAO R, CAI W B, et al. Microbial characteristics and application
potential of complete ammonia oxidation bacteria in water treatment
system[J]. Acta Microbiologica Sinica, 2021, 61(2):315-332.
