融合图Transformer和Vina-GPU+的多模态虚拟筛选新方法
2025-01-22张豪张堃然阮晓东沐勇吴建盛



关键词:虚拟筛选,Graph Transformer,Vina⁃GPU+,多模态,知识蒸馏,主动学习
中图分类号:Q811. 4 文献标志码:A
现代药物发现面临从大型化合物库中进行大规模筛选的挑战[1],提高筛选的速度与精度已是迫切的需求[2]. 随着化合物库规模的不断扩大,传统的筛选方法耗时费力,因此,虚拟筛选成为加快药物研发的常用手段[3]. 虚拟筛选利用计算机模拟分子间的相互作用,能够快速识别潜在的活性化合物,所以显著提升了筛选效率. 另外,在提升虚拟筛选速度的同时,保证其筛选的准确率也至关重要,这直接关系到发现潜在药物候选物的成功率[4].
分子对接是虚拟筛选的一种重要方法,其利用计算机来模拟分子之间的相互作用,揭示靶点与化合物之间的结合模式[5]. 研究人员通常利用分子对接软件对小分子化合物与药物靶标进行分子对接,再进行活性化合物筛选. 分子对接可快速从几十至上百万分子中选出可能成药的活性化合物,大大降低了实际筛选化合物的数目,缩短了研究周期,降低了药物研发的成本. AutoDock Vi⁃na 是分子对接的首选工具之一,以速度和准确性闻名[6]. 在CASF ⁃ 2016基准测试中,AutoDockVina 展示了最佳的对接性能和在蛋白质⁃配体复合物测试集上最佳的评分能力[7-8]. 由于Auto ⁃Dock Vina 整体上是串行设计的,以前的加速方法主要依赖计算资源的叠加. GPU (GraphicsProcessing Unit)以其易用性和成本效益已被广泛应用于加速计算,我们开发的Vina⁃GPU[9]和Vina⁃GPU+[10]利用GPU显著提高了AutoDock Vina 的效率,分别实现了最高50倍和65. 6倍的加速,对于在个人电脑、工作站服务器或云计算等上广泛实现大规模虚拟筛选至关重要.
结合亲和力预测是虚拟筛选中的另一种重要方法. 机器学习包含的多种模型可以解决分类、回归和聚类的问题,结合亲和力预测并通过机器学习模型来估算受体和配体间的结合强度[11],这种信息的有机结合可以更加全面地考虑分子之间的结构和性质,提升虚拟筛选的精度. 例如,2021年Yang et al[12]提出一种基于主动学习的机器学习增强型分子对接协议,能识别得分最高的化合物并探索大范围的化学空间,和纯贪婪的方法相比,展现了更优异的虚拟筛选性能. 2022 年Gen⁃tile et al[4]提出一种深度对接平台,通过对化学库的一个子集的对接来迭代同步预测其余的配体,可实现最多100倍的筛选加速. 2023年Sivula etal[13]提出一种机器学习增强的分子对接方法,实现了在超大规模枚举化学库上的快速虚拟筛选. 2023 年Yu et al[14]提出一种深度学习新方法GEM⁃Screen,利用与特定靶点对接化合物的几何增强分子表示,通过主动学习策略,对库的一小部分进行对接评分训练,可以增强基于分子对接的大规模虚拟筛选的性能. 2024 年Popov et al[15]提出一种基于生成模型增强的分子对接方法HID⁃DEN GEM,整合了机器学习、生成化学和大规模化学相似性搜索,加速了虚拟筛选过程.
随着深度学习技术的快速发展,深度学习在医学领域的应用越来越广泛,大规模预训练模型在自然语言处理和计算机视觉领域取得了巨大成功[16]. 例如,OpenAI 推出的ChatGPT 和Sora 等模型,依托于海量数据的预训练,构建了强大的语言理解和图像识别能力,在多项任务上超越了传统方法. 这一技术也为药物虚拟筛选带来了新思路,为解决数据量大、复杂性高等挑战提供了可能性[17]. 例如,2022 年同济大学刘琦教授课题组与百度公司合作发布X⁃MOL 模型,采用混合注意力Transformer 模型,利用海量训练数据对小分子进行有效表征,显著提升了虚拟筛选性能[18].2022年IBM 研究院发布MOLFORMER 模型,通过预训练处理11 亿未标记分子的SMILES,在分子属性预测等任务中表现出色[19]. 近期,Mifflin et al[20]的知识引导的预训练框架KPGT 集成了专门为分子图设计的图转换器和知识引导的预训练策略,进一步改善了分子表示学习.
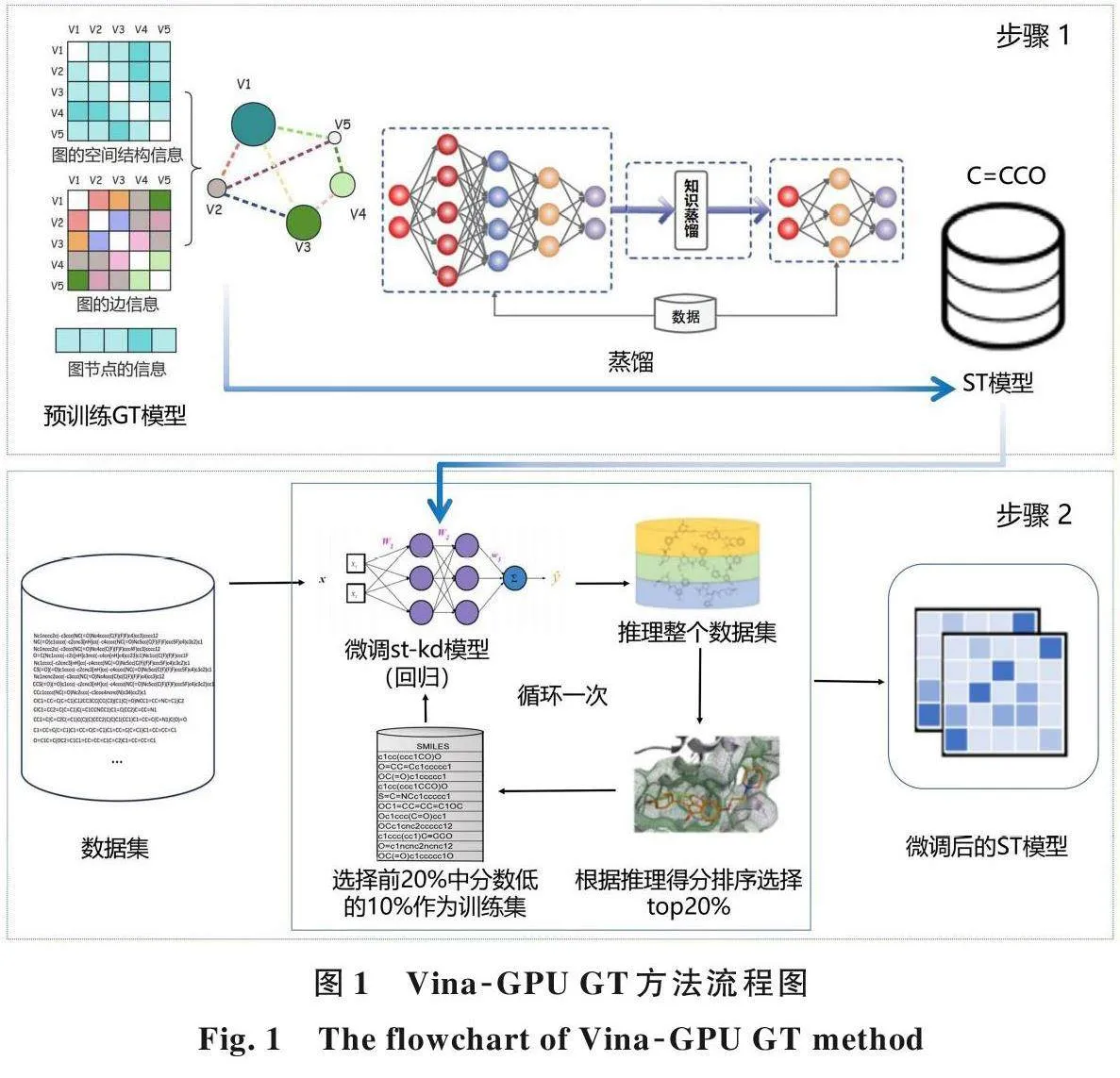
为了进一步提高虚拟筛选的速度和精度,本文提出一种新的多模态虚拟筛选方法Vina⁃GPUGT,结合Vina ⁃ GPU+ 分子对接技术和预训练Graph Transformer (GT)模型,能实现快速准确的虚拟筛选过程. 该方法结合了两种不同维度的分子信息,即分子的序列信息(SMILES)和3D 结构信息(pdbqt)来共同进行筛选,可以增强虚拟筛选的性能. Vina⁃GPU GT 包括三个步骤. 首先,基于已经进行分子属性预测的预训练GT 模型Graphormer 设计了一种全新的知识蒸馏方法来训练一个轻量级SMILES Transformer (ST)模型,它可以更高效地学习并表示复杂的分子结构信息;其次,使用训练好的ST 模型对化合物库中的所有分子进行推理,根据主动学习规则选择2% 的分子,利用Vina⁃GPU+进行分子对接,根据对接得分对ST 模型进行微调,以优化ST 模型的性能;最后,使用微调的ST 模型来预测化合物库分子,并根据其得分对分子进行排序,完成药物的虚拟筛选过程.
在三个重要蛋白靶点(RIPK1,RIPK3,AmpCβ ⁃lactamase)和两个化合物库(DrugBank 和Sel⁃leck)上进行虚拟筛选实验,并与两种不同的虚拟筛选方法进行比较,证明Vina⁃GPU GT 的虚拟筛选性能最佳. 具体地,和Vina ⁃ GPU 以及Vina ⁃GPU+相比,其命中率(HIT 1%)平均提升362. 6% 和354%,富集因子(Enrichment Factor,EF 1%)平均提升332. 7% 和323. 1%.
1 数据与方法
1. 1数据
1. 1. 1蛋白质靶标 选择三种非常重要并被广泛研究的蛋白质作为实验对象. 第一种是RIPK1蛋白[21],它在细胞的增殖、分化以及死亡等关键的生命活动过程中发挥信号传导作用. 第二种是RIPK3 蛋白[22],以其在坏死性凋亡中的作用而知名,坏死性凋亡是细胞程序性死亡的一种形式,通常在细胞的标准凋亡途径受阻时发生. RIPK1和RIPK3对细胞死亡和炎症调控有至关重要的作用,而这两个过程是维持组织内稳态及免疫响应不可或缺的,这两种蛋白质的功能失调与包括癌症、神经退行性疾病和炎症性疾病在内的多种疾病的发生有关. 第三种是AmpC β⁃lactamase,这是一种由某些细菌产生的酶,可导致细菌对包括青霉素类、头孢菌素类和碳青霉烯类在内的多种β ⁃ 内酰胺类抗生素产生耐药性. AmpC β ⁃ lac⁃tamase 细菌的出现和传播已成为一个重要的公共卫生问题,因为它降低了众多抗生素的疗效,并使得由这些细菌引起的感染的治疗复杂化.
1. 1. 2化合物库 选择两个广泛应用的分子数据库进行虚拟筛选. 第一个是DrugBank 数据库[23],它是一个综合性的数据库,提供药物及其靶点和作用机制的信息,本研究从DrugBank 网站(https://go. drugbank. com/releases/latest#)选取11582 个分子及其结构. 第二个是Selleck 数据库,包含一系列小分子化合物,包括各种细胞靶标的抑制剂、激动剂、拮抗剂和活化剂,本研究从L460⁃TargetMol 天然化合物库和L1400⁃Selleck天然产物库的131cpds 板中共获得5148 个分子.
1. 2方法 Vina ⁃ GPU 是AutoDock Vina 的GPU 加速版本,通过大幅增加蒙特卡罗搜索中随机初始构象的个数,显著降低了每个初始构象对应的搜索深度,增加了算法的并行度,充分利用GPU 上数千个计算核心,实现了大规模的并行化和加速. Vina⁃GPU+在Vina⁃GPU 的基础上,对能量的网格表计算进行了优化,减少了冗余计算,进一步加速了虚拟筛选过程.
本文提出的方法结合了Vina⁃GPU+分子对接技术和预训练GT 模型,实现了快速而准确的虚拟筛选过程. 该方法总体分两个阶段,首先是模型的准备,然后是基于主动学习策略的模型微调. 阶段一选用GT 模型作为教师模型,ST 模型作为学生模型,使用知识蒸馏方法得到具有先验知识的ST 模型,在此基础上使用LIT⁃PCBA 数据集[24]中的ALDH1 靶标和小分子的Vina⁃GPU+对接得分对蒸馏后的ST 模型进行微调. 阶段二主要进行基于主动学习策略的分子挑选. 使用阶段一准备好的模型对待筛选的整个化合物库进行推理,升序排列推理得分并选取前20% 的后10%的分子作为对接分子,再使用Vina⁃GPU+进行对接,其打分则用于对ST 模型的进一步微调. 微调好ST 模型之后再一次对整个待筛选的化合物库进行推理,根据推理得分选取目标分子. 本方法的流程框架如图1 所示.
1. 2. 1模型的蒸馏与预训练
1.2.1.1模型的选取 GT 模型是一种专为解析和处理图结构信息而设计的神经网络模型,具有强大的表征能力,其利用自注意力机制能有效地捕获长距离节点之间的关系[25]. GT 模型经过预训练后具备丰富的先验知识,能显著加速其在下游任务中的收敛过程,减少计算资源及时间的消耗,还可以实现知识迁移,提高模型的性能.
同样基于Transformer 架构但和GT 模型不同,ST 模型使用SMILES 字符串作为输入,规避了由SMILES 到图结构转换的效率损失. 为了进一步提升ST 模型的效果,将预训练GT 模型作为教师模型,ST 模型作为学生模型,用模型蒸馏技术实现从预训练GT 模型到ST 模型的知识迁移.
综上,本文选用预训练GT 和ST 模型,并利用Vina⁃GPU+进行得分的微调,以实现不同模态间的信息融合,从而加快模型推理的速度,并在虚拟筛选应用中进一步提高模型的准确性.
1.2.1.2知识蒸馏方法 分别选取GT和ST模型作为教师和学生模型. 选取小分子数据集,使用RDKIT工具包提取SMILES表示中的原子特征和化学键特征. 将SMILES 转换为ST模型的输入embedding:
1. 2. 2 基于主动学习策略的分子挑选、对接与模型微调 为了提升ST 模型在预测Vina⁃GPU+打分方面的性能,采用LIT⁃PCBA 数据集中的AL⁃DH1 靶标及其相应小分子的Vina⁃GPU+对接得分,对经过知识蒸馏处理的ST 模型进行微调,使ST 模型掌握部分Vina⁃GPU+打分的先验知识.
以经过预训练得到的ST 模型为基础,进入基于主动学习策略的微调阶段. 首先利用ST 模型对待筛选数据集进行推理,按升序规则对推理得分排序;然后,依托主动学习策略,选择具有最高信息价值的分子,即排序前20% 的后10% 的分子,作为进一步微调ST 模型的训练集;使用Vina⁃GPU+对这部分分子打分,并据此训练ST模型以拟合这些分数. 此过程迭代执行,直至完成模型的微调.
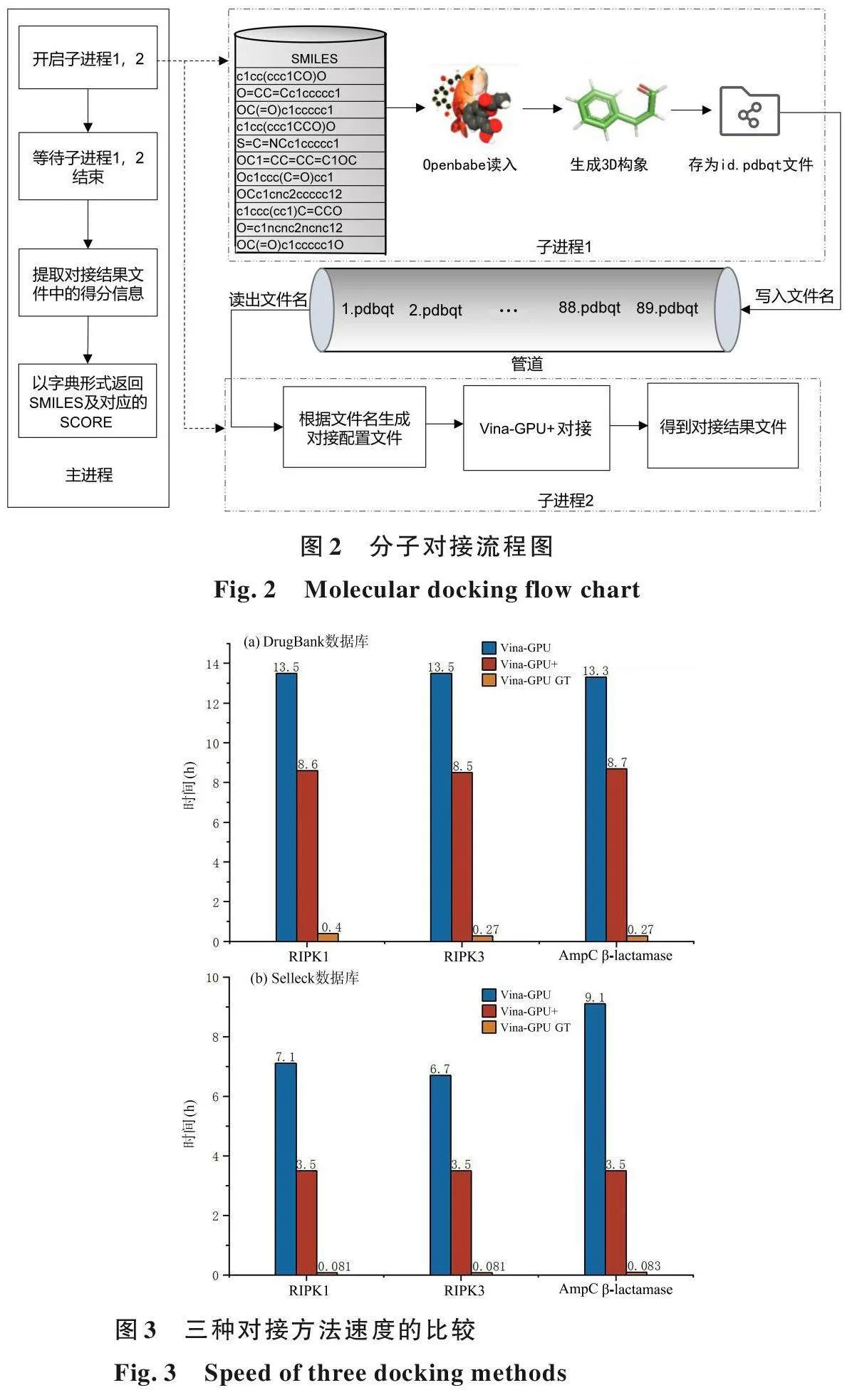
分子对接的过程如图2 所示,考虑到分子构象生成的速度通常快于分子对接,为了提升对接效率,采用基于流水线的处理结构,由两个子进程分别负责分子构象生成与Vina⁃GPU+对接,从而提高整个分子对接模块的吞吐量. 获取每个分子的对接得分后,将其按SMILES 与SCORE键值对的格式整理,构成训练数据集. 训练过程中选用MAE 作为损失函数,当MAE 收敛至较小值时,模型微调阶段完成.
最终,使用经过微调的ST 模型对整个虚拟筛选数据集进行推理,并根据模型的推理得分进行排序. 根据研究需求,选取得分最高的Topk 分子,作为虚拟筛选的目标分子.
2 结果与讨论
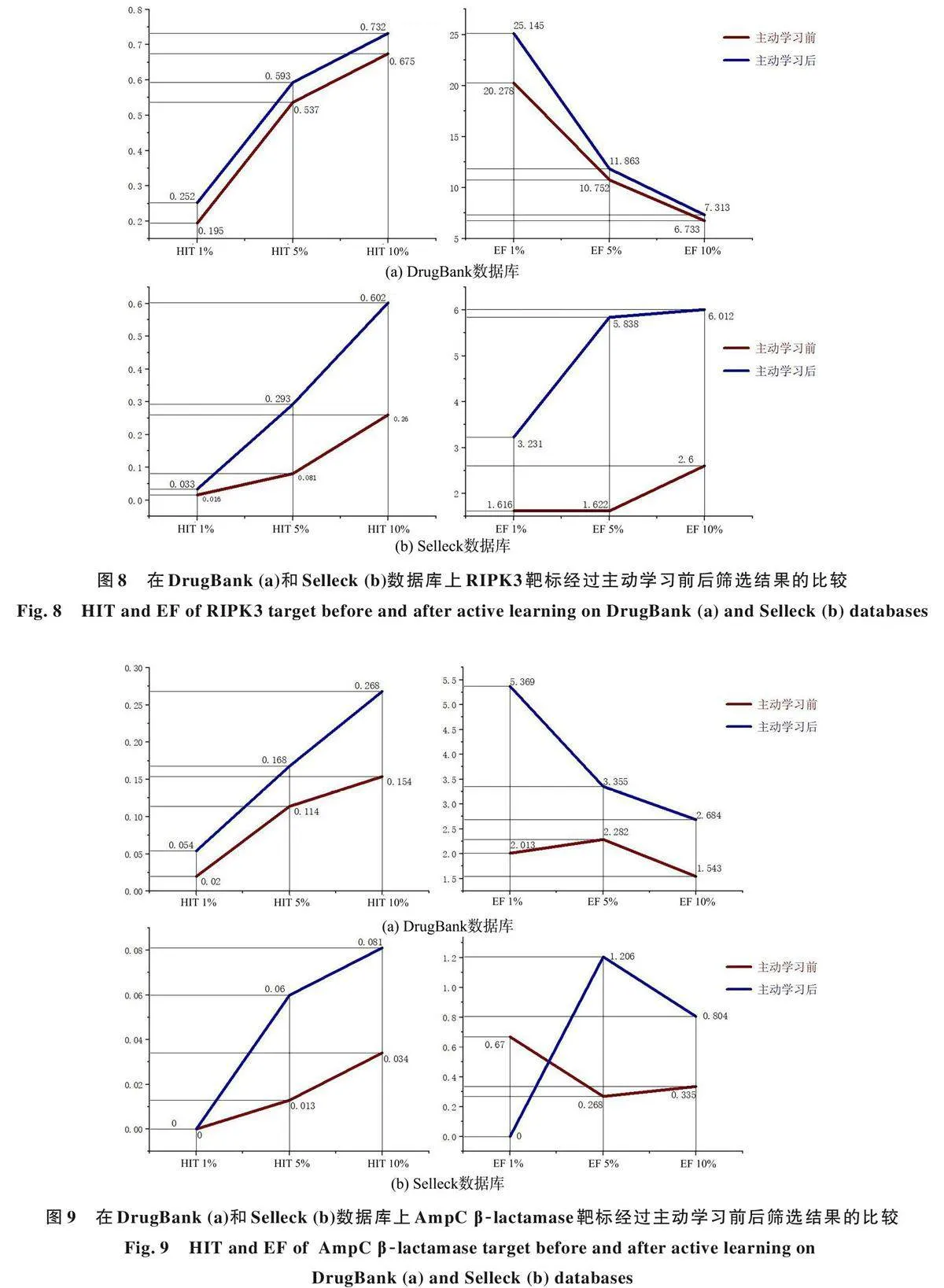
2. 1虚拟筛选速度比较 图3 展示了在Drug⁃Bank 和Selleck 数据库上使用三种对接方法对三个靶标进行虚拟筛选的时间. 与Vina⁃GPU 和Vi⁃na⁃GPU+相比,Vina⁃GPU GT 在DrugBank 数据库上进行虚拟筛选的时间平均减少97. 6% 和96. 3%,在Selleck 数据库上进行虚拟筛选的时间平均减少98. 9% 和97. 7%. 总体而言,Vina ⁃GPU GT 和Vina⁃GPU+相比实现了31 倍的虚拟筛选加速,和Vina⁃GPU 相比实现了53 倍的虚拟筛选加速.
2. 2在DrugBank数据库上的虚拟筛选结果
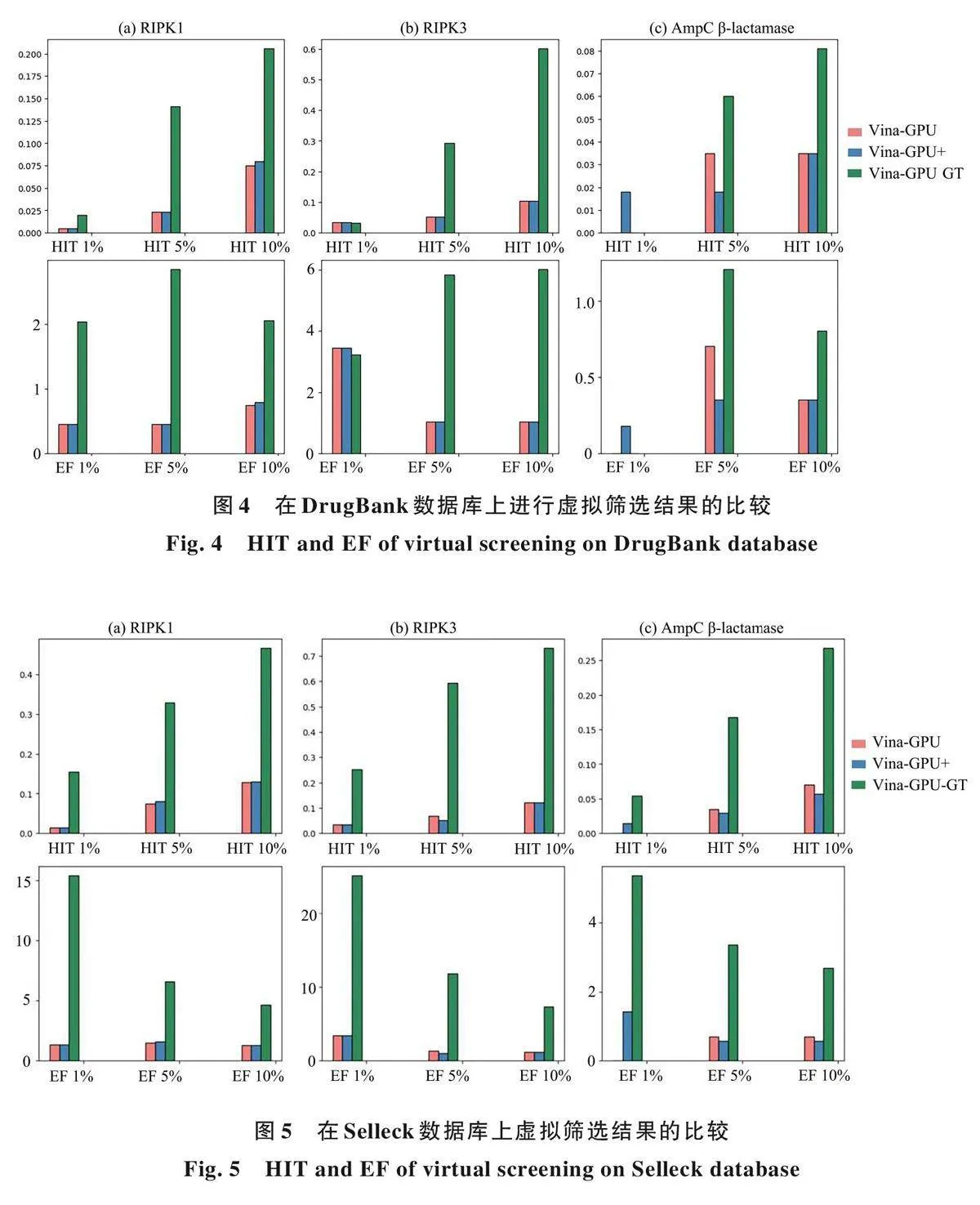
图4 展示了在DrugBank 数据库上三种对接方法对三个受体的虚拟筛选结果,由图可见,Vina ⁃GPU GT 的HIT 和EF 显著优于Vina⁃GPU 和Vi⁃na⁃GPU+. 具体地,与Vina⁃GPU 相比,Vina⁃GPUGT 的HIT(1%,5%,10%)平均提升547. 1%,492. 7%,350. 9%,EF(1%,5%,10%)平均提升552. 1%,491. 8%,351. 3%. 与Vina⁃GPU+相比,Vina⁃GPU GT 的HIT(1%,5%,10%)平均提升642. 2%,610. 3%,378. 1%,EF(1%,5%,10%)平均提升644. 9%,616%,378. 8%. 在AmpC β⁃lactamase 分子对接结果中,Vina ⁃ GPU+ 的HIT1% 和EF 1% 高于Vina⁃GPU GT,这可能是由于在AmpC β⁃lactamase 这个靶标的数据集中,作为正样本的活性分子有许多SMILES 含有“. ”这种Vina⁃GPU 和Vina⁃GPU+分子对接软件无法计算的分子,无法得到这些分子的打分结果,导致在微调时训练的数据量不足. 并且,作为活性分子,这些分子的打分信息对于模型训练可能更关键,这部分信息的缺失使模型的较严苛的HIT 1% 和EF 1% 指标的表现不佳.
综上,对DrugBank 数据库的虚拟筛选,与Vi⁃na⁃GPU 和Vina⁃GPU+两种方法相比,Vina GPUGT 的筛选结果更优.
2. 3在Selleck 数据库上的虚拟筛选结果 图5展示了在Selleck 数据库上三种对接方法对三个受体的虚拟筛选结果,由图可见,Vina⁃GPU GT的筛选精度显著优于Vina⁃GPU 和Vina⁃GPU+.具体地,与Vina ⁃ GPU 相比,Vina ⁃ GPU GT 的HIT(1%,5%,10%)平均提升99%,349. 3%,263. 5%,EF(1%,5%,10%)平均提升113. 3%,353. 7%,261. 5%. 与Vina ⁃ GPU+ 相比,Vina ⁃GPU GT 的HIT(1%,5%,10%)平均提升65. 7%,403. 2%,257. 8%,EF(1%,5%,10%)平均提升80. 3%,411. 4%,256. 6%.
综上,对Selleck 数据库的虚拟筛选,与Vina⁃GPU 和Vina ⁃ GPU+ 两种方法相比,Vina ⁃ GPUGT 的筛选结果更优.
2. 4虚拟筛选的得分比较 图6 展示了在Drug⁃Bank 数据库上三个受体的虚拟筛选得分. 由图可见,Vina⁃GPU GT 对RIPK1 受体的打分主要集中在-8 以内,对RIPK3 受体的打分主要集中在-9以内,对AmpC β ⁃ lactamase 的打分主要集中在-10 以内. 与Vina⁃GPU 和Vina⁃GPU+的得分相比,Vina⁃GPU GT 在虚拟筛选时面对不同的受体,对配体的打分差异更明显. 此外,Vina⁃GPU GT,Vina⁃GPU,Vina⁃GPU+在DrugBank 数据库上对RIPK1 的平均得分分别为-4. 8,-6. 6,-6. 6;对RIPK3 的平均得分分别为-7. 6,-6. 6,-6. 6;对AmpC β ⁃lactamase 的平均得分分别为-7. 2,-7. 0,-7. 0. 根据对不同受体的得分数据,可以说明Vina⁃GPU GT 在进行虚拟筛选时更好地考虑了受体的信息. 同时,结合2. 2和2. 3 中三种方法的虚拟筛选结果,也证明在三种受体上,Vina⁃GPU GT 对于配体的打分和排序更加准确.
2. 5消融实验 为了评估主动学习策略对模型筛选性能的影响,开展了一系列针对模型主动学习部分的消融实验. 实验结果如图7~9 所示,分别展现了三个不同靶标在DrugBank和Selleck 两个数据库上的虚拟筛选性能. 由图可见,除了AmpC β⁃lactamase 靶标在Selleck 数据库上的虚拟筛选中,主动学习后的EF 1% 稍低于未经主动学习的结果外,其他所有实验场景中,主动学习策略均带来了指标的显著提升,证明将主动学习集成至模型中可以有效地提高虚拟筛选的准确性.
3 结论
本文提出一种多模态虚拟筛选新方法Vina⁃GPU GT,融合了Vina⁃GPU+分子对接方法和预训练图Transformer 模型. 实验结果证明,Vina⁃GPU GT 的筛选速度和精度都优于Vina⁃GPU 和Vina⁃GPU+. 不过,其仍然存在一些局限. 例如,该方法的性能高度依赖于预训练模型的质量和用于知识蒸馏的数据,并且,只选择了2% 的分子进行对接和微调,可能忽略了其他具有潜力的分子. 另外,该方法在不同类型的靶点或化合物库上的泛化能力有待验证. 未来将进一步优化Vina⁃GPU GT 方法,以提高其在更广泛靶点和化合物库中的适用性和准确性.
(责任编辑 杨可盛)
